-
工程纳米颗粒(engineered nanoparticles, ENPs)是指至少在一维尺度上介于1—100 nm的颗粒,凭借其优异的理化性质得到各个领域的“青睐”,广泛用于个人护理产品、油漆、催化剂、半导体、医学成像以及太阳能电池等[1 − 5]. 随着纳米技术的不断发展,工程纳米材料的产量迅猛增加,从2010年的21千吨增长到2020年的58千吨[6]. 然而,ENPs的生产和使用,不可避免地导致其暴露在环境中,并与生态系统中不同营养级生物相互作用,引起了人们对ENPs生物暴露和潜在风险的担忧[7 − 8]. 水环境是ENPs的一个重要归宿,ENPs会通过地下水修复、污水排放等各个渠道进入其中[9 − 12]. 进入水环境中的ENPs会被生物摄入并积累,进而产生毒性效应,如DNA损伤、产生氧化应激、产生组织病理学变化以及生长受到抑制等[13 − 21]. 有研究表明,只有被摄入并积累的ENPs才是对水生生物产生毒性效应的关键[22]. 因此,有必要研究水生生物对ENPs的积累,以提供毒理学研究所必需的信息.
负荷量(body burden, BB)代表给定时间生物体内的ENPs浓度,是定量ENPs生物积累的基础参数[23 − 25]. 现有的研究多通过化学消解、放射性元素标记或者荧光标记等实验方法测定ENPs的负荷量,过程复杂且耗时耗力[19, 24, 26]. ENPs的种类繁多,很难通过实验来满足毒理学研究对ENPs负荷量数据的需要. 因此,亟需发展可代替传统实验方法的ENPs负荷量预测模型. 目前,关于ENPs生物积累的预测模型的研究十分有限[23, 27]. Dong等[23]应用机器学习(ML)算法对不同ENPs的负荷量进行预测,以此来满足毒代动力学模型的数据需求,实现对ENPs生物积累潜力的评估. 然而,现有模型对BB的预测是在个体水平上的[23]. ENPs的生物积累具有组织器官依赖性,了解ENPs在生物体内的分布,以及在关键靶器官中的生物积累尤为重要[27 − 28]. 此外,现有的许多ML算法是一些“黑箱模型”,如随机森林,极端梯度提升等[29]. 因此,除了要关注模型的预测性能外,还需要考虑模型的可解释性. SHAP (SHapley Additive exPlanations)方法整合了几种具有坚实理论背景的方法(如局部可解释的模型-诊断性解释、博弈论),并提供了强大的估计算法和软件,它与机器学习的结合成为识别特征重要性以及探索数据关系的有力工具[29 − 31].
本研究构建了涵盖17种ENPs和23种水生生物的BB数据集(n =
1303 ). 基于该数据集,将ENPs的本征特性、暴露参数、生物物种以及组织器官类型等43个参数作为描述符,综合考虑前人研究以及不同机器学习算法的适应性,采用多元线性回归、支持向量机、随机森林、梯度提升决策树和极端梯度提升算法建立了ENPs的BB定量预测模型. 本研究通过增加数据集中生物种类,扩大了模型应用域,实现了特定暴露时间、暴露浓度下生物体内ENPs负荷量的预测,并将负荷量的预测精准到组织器官水平. 此外,为了弥补机器学习算法在可解释性方面的不足,本研究结合SHAP方法解析不同特征对ENPs负荷量的影响,拓展了对ENPs生物积累的认识与理解. -
本研究共收集了
1303 个BB数据,数据来源于文献[23, 27, 32 − 47]. 数据集涵盖了17种ENPs (Ag、Au、Cu、CuO、TiO2、CeO2、Al2O3、Fe2O3、Fe3O4、Zn、ZnO、Mn2O3、Co3O4、SiO2、Pt、C60、CNTs)、23种测试物种(表1)的BB数据. 此外,该数据集还包括ENPs在生物整体以及不同组织器官[脑、肝脏、鳃、肠道、肾脏、脾脏、肌肉、排泄腺、肝胰腺、足、幔、血淋巴以及胃]中的BB数据. 生物整体与组织器官的BB数据的比例约为3:1. -
将BB实测值(μg·g−1,干重)进行以10为底数的对数转换,以lgBB作为预测终点. 本研究考虑的特征包括以下4种类型:ENPs本征特性,包括密度[g·(cm3)−1]、分子量、电负性和原始粒径(nm);暴露条件,即暴露浓度(mg·L−1)和暴露时间(h);生物特性,即23个生物物种;ENPs在生物体内积累的位置,包括生物整体和不同组织器官. 首先对于分类变量,即生物的种类和积累部位进行独热编码(OneHotEncoder),将文字类型的特征转换为数值型,编码后得到43个候选特征. 由于大量的特征会增加数据的噪声,增加机器学习算法的误差,因此有必要剔除掉不相关或者冗余的特征[48]. 按照3个步骤完成特征的选择:去除方差为0的变量;去除皮尔逊相关系数大于0.9的变量;通过交叉验证递归特征消除法选定最优特征数[49]. 交叉验证递归特征消除法采用的模型算法为极端梯度提升(XGBoost),每次迭代中的特征剔除数量为1,交叉验证所使用的折数为10折,评估模型性能的评价指标为10折交叉验证系数(Q210-CV). 本研究采用Scikit-Learn库中的StandardScaler方法对数据进行标准化.
-
将数据集按照4:1的比例随机划分为训练集和测试集,分别用于模型的构建和模型的外部验证. 综合考虑前人研究以及不同ML算法的适应性,最终选择了线性方法、非线性方法以及集成学习算法,包括多元线性回归(MLR)、支持向量机(SVM)、随机森林(RF)、梯度提升决策树(GBDT)和极端梯度提升(XGBoost),进行模型的构建[50 − 56]. 使用Scikit-Learn的GridSearchCV来调整模型参数,以确定模型最优参数. 本研究使用经自由度调整后的决定系数(R2adj)和均方根误差(RMSE)来表征模型的拟合优度,使用测试集外部验证系数(Q2test)和均方根误差(RMSEtest)表征模型的预测能力;使用训练集的10折交叉验证系数(Q210-CV)表征模型的稳健性[51]. 此外,为了确保模型的稳健性,检验因变量和自变量之间的偶然关系,本研究还对最优的模型进行Y随机化验证. Y随机化验证是通过扰乱训练集中样本的因变量值进行排列组合测试[57]. 本研究使用相同的描述符和最优的机器学习算法建立了500个基于因变量值排列组合的模型. Y随机化验证结果的Q2Y-scrambling-10-CV, R2Y-scrambling和RMSEY-scrambling均是取500个模型的平均值. 采用基于杠杆值(hi)和标准残差(δ)的Williams图进行模型的应用域表征[58]. 本研究所有程序均基于Python 3.9.13实现.
-
本研究采用Python中的SHAP包对模型的结果进行解释. SHAP是一种事后解释方法,在整合了其他可解释方法的基础上,该方法借鉴了博弈论思想,主要侧重于对已经构建好的复杂机器学习模型的结果进行解释,通过计算模型中各个特征的边际贡献(即Shapley值)来量化以及衡量各个特征对预测结果的影响大小,进而对普遍缺乏可解释性的“黑箱”模型进行解释[29]. SHAP的主要贡献是生成局部加性特征归因,如公式(1)所示[29]:
式中,f(xi)是第i个观测值(样本)的预测值;SHAP0 = E(f(x))是所有观测值(样本)预测值的平均值;而
$ \text{S}\text{H}\text{AP}({X}_{ji}) $ 指的是第i个观测值(样本)的第j个特征对预测值的SHAP值,表示该特征对预测的边际贡献. SHAP的绝对值反映了特征对模型预测的影响强度,可以作为特征的重要性评分[29]. -
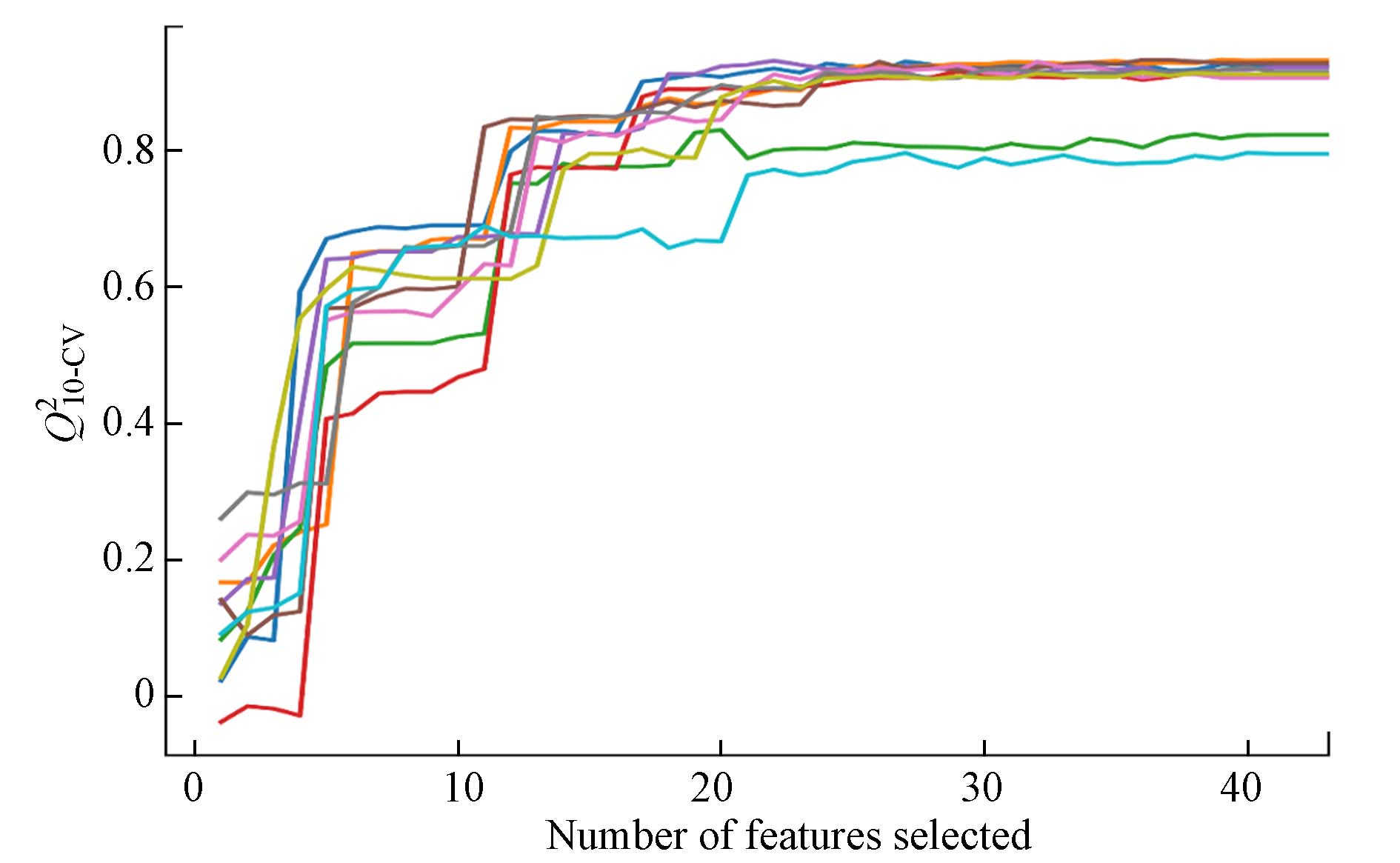
本研究以lgBB为预测终点,将43个候选特征作为描述符构建模型. 基于交叉验证递归特征消除法的特征选择结果如图1所示. 依据特征重要性排序结果,当特征数量达到24个以后,随着特征数量的增加,模型的交叉验证系数并没有明显提升,最终选定的特征数量为24个. 最终保留的特征包括2个暴露参数(暴露浓度、暴露时间),4个ENPs本征特征(密度、电负性、分子量、粒径),12个生物物种[大型溞(Daphnia magna)、海蠕虫(Capitella teleta)、棕二鬚鲃(Capoeta fusca)、金鱼(Carassius auratus)、网纹溞(Ceriodaphnia dubia)、小球藻(Chlorella sp.)、虹鳟鱼(Oncorhynchus mykiss)、沙蚕(Nereis diversicolo)、克氏原螯虾(Procambarus clarkii)、条纹鲮脂鲤(Prochilodus lineatus)、莱茵衣藻(Chlamydomonas reinhardtii)以及角类扁卷螺(Planorbarius corneus)]以及6个积累部位(生物整体、血淋巴、脑、脾脏、肠道以及肌肉).
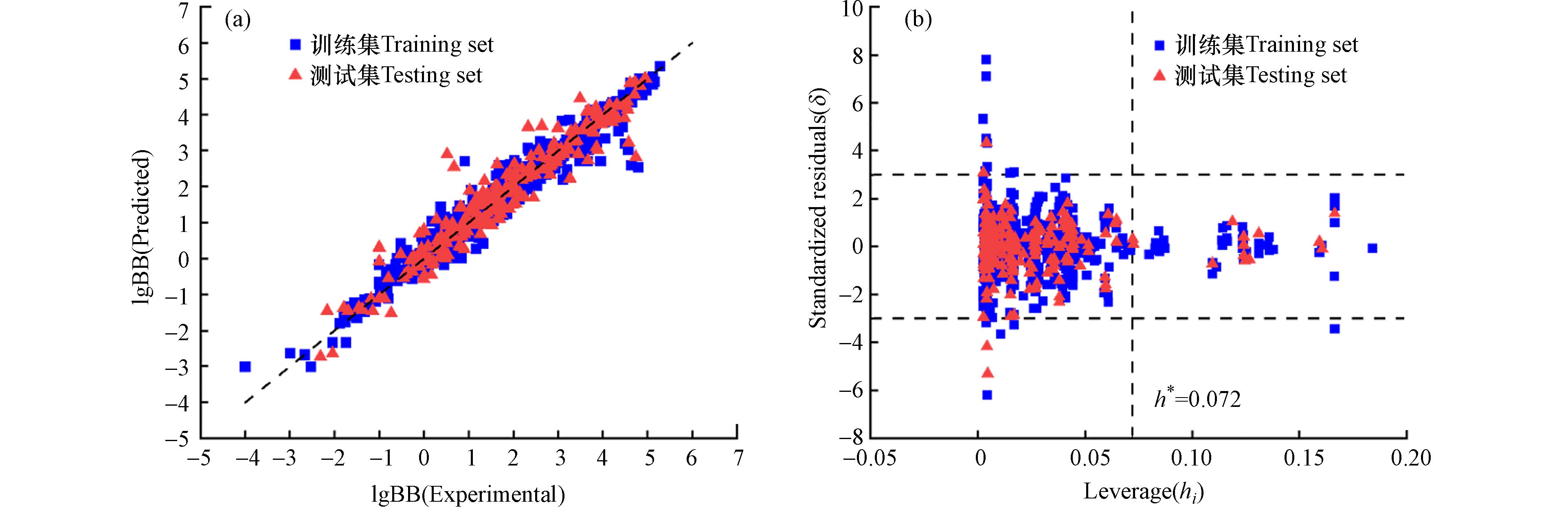
基于24个筛选的特征,所构建的模型统计学参数见表2. 相比于线性模型(MLR),非线性模型(SVM, RF, GBDT, XGBoost)的性能有了较大提升. 综合考虑各项评价参数,XGBoost模型(n_estimators = 51, max_depth = 7, random_state =10)的拟合效果(R2adj-train = 0.971, RMSEtrain = 0.232),预测能力(Q2test = 0.909, RMSEtest = 0.489),以及稳健性(Q210-CV = 0.887)都是最优的. 图2a为XGBoost模型对ENPs的lgBB的预测值和实验值的拟合图. 本研究还对XGBoost模型进行了Y随机化验证,500次建模结果的Q2Y-scrambling-10-CV = −0.398, R2Y-scrambling = 0.575, RMSEY-scrambling = 2.945. 该结果表明,当随机置换预测终点lgBB值时,模型的稳健性、拟合效果极度下降,训练集的均方根误差显著增加,因此排除了XGBoost模型的偶然性.
-
本研究采用Williams图对XGBoost模型进行应用域表征(如图2b所示),以确定模型适用的ENPs范围. 该模型的警戒杠杆值(h*)为0.072,只有当杠杆值hi < h*且|δ| < 3.0的ENPs才适用于本模型. 本研究训练集和测试集中分别有14和4个数据点的δ落在(−3.0, +3.0)以外,被认为是离群点,分别占训练集的1.34%、测试集的1.53%. 这18个离群点对应的ENPs及其数目分别为Ag (5)、 ZnO (2)、 TiO2 (7)、Au (3)以及Fe2O3 (1). 因为数据来源于不同的实验室,异常值可能是由数据的异质性造成的,也可能是由于实验误差引起的. 此外,ENPs的hi > h*但是|δ| < 3.0的数据点共计68个. 这说明该模型具有较好的泛化能力[51].
-
相比于传统线性模型(例如MLR)的简单、机理透明,XGBoost模型几乎是一个“黑箱模型”,无法深入探究lgBB与各特征之间的关系[29]. 本研究应用SHAP解释方法对XGBoost模型进行可视化分析. 基于SHAP概要图(图3),可以识别重要特征并量化其对模型的贡献. SHAP概要图中每个点都表示单个的样本,每行表示一个特征,其重要程度自上而下依次降低[31]. SHAP值越大表示特征对于lgBB预测结果影响越大,SHAP值大于0代表特征对ENPs的负荷量具有正向影响,SHAP值小于0则相反. 由图3可知,对lgBB影响程度从高到低的前10个特征依次是大型溞(Daphnia magna)、密度、暴露浓度、粒径、生物整体(积累部位)、暴露时间、电负性、棕二鬚鲃(Capoeta fusca)、分子量、克氏原螯虾(Procambarus clarkii).
ENPs的本征特征,即密度、粒径、电负性和分子量,是影响其负荷量的关键特征. 密度是影响ENPs负荷量的首要本征特征. 该特征的数据分布较为分散,代表该特征影响很大. 对于该特征,SHAP值较大的红点大部分集中于垂直线(SHAP为0)左侧,SHAP值较低的蓝点集中在垂直线右侧,表明该特征对ENPs的lgBB产生了负向影响. 基于目前的数据集得到结果是,密度大的ENPs的负荷量越小. Dong等[23]的研究也表明ENPs的密度是影响其在生物体内积累的首要基本特征,这可能是由于ENPs在生物体中的质量传递是由悬浮液的密度决定. 其次是粒径,大量的研究表明ENPs的粒径在其被生物体摄取时发挥着重要作用,ENPs的吸收高度依赖于粒径[28, 35, 59 − 61]. 例如,粒径在4—10 nm之间的颗粒可以直接穿透细胞膜双分子层[28]. 但是,由图3可以看到,粒径这一特征的蓝点和红点堆叠在一起,因此基于目前的数据不易明确其对ENPs负荷量的影响. 电负性是构建ENPs生物毒性模型中较为常用的描述符之一[57, 62 − 63]. 本研究首次应用电负性作为描述符,预测了ENPs的负荷量. 此外,目前对ENPs在生物体内的定量数据多是基于质量浓度(ng·g−1)而不是摩尔浓度(mol·kg−1),这就导致ENPs的负荷量与其分子量之间存在关联.
暴露浓度和暴露时间是与ENPs生物积累直接相关的两个暴露条件. 由图3可以看出,这两个特征的红点和蓝点部分重叠,但是存在一定的趋势,即红点集中在SHAP值大于0的一侧,大部分的蓝点在SHAP值小于0的一侧,代表这两个特征对ENPs的负荷量产生了一定的正向影响. 这是因为暴露浓度和暴露时间通过改变生物体与ENPs之间相互作用的强度和时间,直接影响对ENPs的摄取和积累[23, 64].
生物是影响ENPs负荷量的重要因素,不同的生物对ENPs的“敏感”程度不一样,对ENPs的摄取和积累也存在差异. 由图3可以看出,大型溞(Daphnia magna)这一特征的红点集中在SHAP值大于0一侧,表明大型溞比其他生物更容易摄取和积累ENPs. 而棕二鬚鲃(Capoeta fusca)和克氏原螯虾(Procambarus clarkii)则相反,说明它们对ENPs的积累能力相对较低. 这可能是由于物种之间的差异引起的,如体型大小、摄食行为、代谢能力等[23, 65]. 无脊椎动物大型溞作为一种被广泛用于评估化学品毒性和生物积累的小体型滤食性浮游动物,其“饮水”行为促进了对ENPs的摄取和积累[66]. 此外,大型溞在水生食物链中的位置处于初级生产者(例如绿藻)与次级消费者(例如斑马鱼)之间,在食物链的物质平衡以及能量循环中发挥着重要作用[25]. 研究表明,大型溞可以通过改变ENPs的颗粒大小极大地影响其在较高营养级中的生物归趋以及在整个水生食物链中的分布[67]. 相比于大型溞,棕二鬚鲃和克氏原螯虾的体型较大,其摄取水和食物的能力较弱,对ENPs的积累能力较低[23].
ENPs的生物蓄积被证明是器官依赖性的[27]. 本研究识别出肌肉组织和脑部是ENPs最不易积累的位置,而肠道、脾脏以及血淋巴是ENPs相对较易积累的位置. 相比于其他组织,肌肉是ENPs生物积累水平最低的组织,肠道比其他组织积累更多的ENPs,这与先前的研究结果[27, 68]是一致的. 本研究首次实现了对脑、脾脏、肠道以及肌肉等不同组织器官中的ENPs负荷量的预测. 然而,关于ENPs在不同组织器官中的负荷量数据较少,有待进一步的分析.
图4是本研究采用SHAP方法对数据集中AgNPs的1个BB数据进行的局部解释. 图4的横轴为SHAP值,纵轴是该样本的不同特征值. 该样本的预测值f(x) = 1.296. E[f(x)] = 1.856表示的是所有样本f(x)值的期望值. 红色和蓝色以及色块上的数值分别代表了特征对最终输出的f(x)值产生的正向或负向贡献以及贡献大小. 由图4可知,对于该AgNP样本,粒径、暴露时间等因素对负荷量产生负向影响,而密度、电负性等因素对其产生了正向影响.
-
目前,有关ENPs负荷量的预测模型非常少(表3),本研究旨在这一研究领域进行新的探索. Dong等[23]首次构建了ENPs的lgBB的预测模型,研究了9种材料特性、7种动物特性和8种暴露条件对ENPs负荷量的影响. 本研究在Dong等[23]的研究基础上,首先通过增加数据集中生物种类(包括浮游植物、浮游动物、底栖动物以及鱼类等)和ENPs的种类,扩大了模型的应用域,使得其应用范围更加广泛,并且对模型的应用域进行了表征,明确了模型的适用范围. 虽然模型效果并未得到明显提升,但仍具有较高的拟合效果、预测能力和稳健性. 此外,本研究将负荷量的预测精准到组织器官水平(脑、脾脏、肌肉等),有助于加深ENPs在生物体内关键组织器官分布和积累的认识. 此外,本研究选取一些易于获取的ENPs本征特征作为描述符,代替实验测定描述符参与模型的构建,发现构建的模型性能较好. 本研究还采用SHAP解释方法进一步解析了关键影响因素对负荷量预测结果的影响.
-
本研究首先构建了一个涵盖17种ENPs、23种测试生物的lgBB数据集(n =
1303 ). 基于该数据集,采用5种机器学习算法建立了用于定量ENPs在不同生物体内负荷量的预测模型,其中XGBoost模型表现出优异稳健性(Q210-CV = 0.887)和预测能力(Q2test = 0.909, RMSEtest = 0.489). Y随机化验证结果排除了模型的偶然性. 基于SHAP方法的模型特征重要性分析,发现ENPs的本征特性(密度、粒径等)、暴露参数(暴露浓度、暴露时间)以及组织器官类型是影响负荷量的关键因素. 本研究通过增加数据集中生物种类,扩大了模型应用域,实现了特定暴露时间、暴露浓度下ENPs负荷量的预测,并将预测结果精准到组织器官水平(脑、脾脏、肌肉等),为ENPs生物积累的研究提供了有效方法. 然而,目前的研究仍然存在一些局限性,因为收集的数据有限,无法全面考虑各种可能的影响因素,因此可能忽略一些关键特征,从而对模型的预测结果产生一定误差. 此外,本研究收集的均为单一ENPs暴露情况下的生物负荷量,并没有考虑到实际环境中多种ENPs的复合暴露,这可能会影响ENPs在生物体内负荷量的变化. 针对以上的局限性,有待后续进一步的研究.
工程纳米颗粒生物负荷量的机器学习预测模型
Machine learning prediction model on body burden of engineered nanoparticles in organisms
-
摘要: 工程纳米颗粒(ENPs)在生物体内的动态浓度即负荷量(BB),是评估ENPs生物积累的基础参数. 大多数研究都通过实验手段来测定BB,其过程繁琐复杂. 构建BB预测模型成为代替动物实验的有效途径. 现有预测模型涵盖的ENPs以及生物种类较少,而且是基于生物整体的BB数据构建的,很难用于预测关键组织器官的负荷情况. 了解ENPs在生物体内的分布,以及在关键靶器官中的积累尤为重要. 本研究通过文献挖掘,构建了涵盖17种ENPs以及23种水生生物的BB数据集(n =
1303 ),采用5种机器学习算法构建模型,并结合SHAP方法解析了不同特征对lgBB的影响. 结果表明,非线性模型要优于线性模型,极端梯度提升(XGBoost)模型的效果最佳(R2adj-train = 0.971,Q2test = 0.909,Q210-CV = 0.887). 通过模型特征重要性分析,发现ENPs的本征特性(密度、粒径、电负性、分子量)、暴露参数(暴露浓度、暴露时间)、生物种类以及组织器官类型是影响ENPs负荷量的关键因素. 其中,密度是影响ENPs负荷量的首要本征特征,负荷量会随着材料密度的增加而减少. 本研究通过增加数据集中生物种类,扩大了模型应用域,实现特定暴露时间和暴露浓度下ENPs负荷量的预测,并将预测结果精准到组织器官水平(脑、脾脏、肌肉等),为ENPs生物积累的研究提供了有效方法.Abstract: Dynamic concentration of engineered nanoparticles (ENPs) in an organism, commonly known as body burden (BB), is a fundamental parameter for assessing the bioaccumulation of ENPs. Prediction models on BB are effective alternatives to complex and time-consuming experimental methods. However, existing prediction models involved relatively few types of ENPs and biological species, and were based on BB data of whole organisms, making it difficult to predict BB in specific tissues or organs. Furthermore, the tissue-organ dependence of ENPs bioaccumulation highlights the importance of understanding their distribution in organisms and their bioaccumulation in key target organs. In this study, a BB dataset (n =1303 ) covering 17 types of ENPs, 23 aquatic organisms, and 13 different tissue organs was compiled through literature mining. Five machine learning algorithms were used to build models, and SHapley Additive exPlanations (SHAP) analysis was conducted to examine how different features of ENPs affect BB. The modeling results demonstrated that nonlinear models outperformed linear models, and the extreme gradient boosting (XGBoost) model performed best (R2adj-train = 0.971, Q2test = 0.909, Q210-CV = 0.887). SHAP analysis showed that the intrinsic properties of ENPs (density, particle size, electronegativity and molecular weight), exposure parameters (exposure concentrations and time), biological species, and organ or tissues types were key factors affecting BB. Among them, density was identified as the primary basic feature affecting BB, i.e., the BB decreased with increasing material density. By increasing the number of biological species and types of ENPs in the dataset, this study expands the application domain of the model, and achieves the prediction of the BB of ENPs under specific exposure time and exposure concentrations. The model developed in this study could predict BB accurately at the tissue/organ level (brain, spleen, muscle, etc.), which provided an effective method for the assessment of ENPs bioaccumulation.-
Key words:
- engineering nanoparticles /
- bioaccumulation /
- body burden /
- tissue and organ /
- machine learning.
-
-
表 1 物种名称
Table 1. Name of the species
拉丁学名
Latin scientific name中文名称
Chinese Name拉丁学名
Latin scientific name中文名称
Chinese NameDaphnia magna 大型溞 Capitella teleta 海蠕虫 Artemia salina 卤虫 Capoeta fusca 棕二鬚鲃 Danio rerio 斑马鱼 Carassius auratus 金鱼 Cyprinus carpio 鲤鱼 Ceriodaphnia dubia 网纹溞 Chlorella sp. 小球藻 Oncorhynchus mykiss 虹鳟鱼 Gammarus fossarum 片脚类动物 Leptocheirus plumulosus 片脚类动物 Hyalella azteca 端足虫 Lymnaea stagnalis 静水椎实螺 Mytilus galloprovincialis 紫贻贝 Nereis diversicolo 沙蚕 Oreochromis niloticus 罗非鱼 Procambarus clarkii 克氏原螯虾 Prochilodus lineatus 条纹鲮脂鲤 Planorbarius corneus 角类扁卷螺 Chlamydomonas reinhardtii 莱茵衣藻 Chlorella pyrenoidosa 蛋白核小球藻 Desmodesmus subspicatus 近具棘链带藻 表 2 模型相关统计参数汇总
Table 2. Summary of statistical parameters of models
模型
ModelR2adj-train Q2test Q210-CV RMSEtrain RMSEtest MLR 0.552 0.478 0.523 0.991 1.170 SVM 0.917 0.812 0.834 0.337 0.702 RF 0.917 0.899 0.879 0.249 0.516 GBDT 0.917 0.829 0.796 0.422 0.670 XGBoost 0.971 0.909 0.887 0.232 0.489 表 3 本研究与其他模型的比较
Table 3. Comparison of the current model with previous models
模型
Model特征
数目
Number of
features数据量
Number of
datasetsENPs种类
Types of
ENPs物种数量
Number of
species生物结构层次
Biological
structure
hierarch最优算法
Optimal
algorithmR2train RMSEtrain Q2test RMSEtest Dong等[23] 24 577 11 9 个体 XGBoost 0.98 0.19 — — 本研究 24 1303 17 23 个体/组织器官 XGBoost 0.97 0.23 0.91 0.45 注:Dong等的研究中并未提供关于测试集的预测效果,故无法进行比较.
Note: Prediction ability on test set was not provided in the study of Dong et al. and therefore could not be compared. -
[1] LEAD J R, BATLEY G E, ALVAREZ P J J, et al. Nanomaterials in the environment: Behavior, fate, bioavailability, and effects—An updated review[J]. Environmental Toxicology and Chemistry, 2018, 37(8): 2029-2063. doi: 10.1002/etc.4147 [2] SRIVASTAVA V, GUSAIN D, SHARMA Y C. Critical review on the toxicity of some widely used engineered nanoparticles[J]. Industrial & Engineering Chemistry Research, 2015, 54(24): 6209-6233. [3] CHRISTIAN P, VON DER KAMMER F, BAALOUSHA M, et al. Nanoparticles: Structure, properties, preparation and behaviour in environmental media[J]. Ecotoxicology, 2008, 17(5): 326-343. doi: 10.1007/s10646-008-0213-1 [4] JOO S H, ZHAO D Y. Environmental dynamics of metal oxide nanoparticles in heterogeneous systems: A review[J]. Journal of Hazardous Materials, 2017, 322: 29-47. doi: 10.1016/j.jhazmat.2016.02.068 [5] BATHI J R, MOAZENI F, UPADHYAYULA V K K, et al. Behavior of engineered nanoparticles in aquatic environmental samples: Current status and challenges[J]. Science of the Total Environment, 2021, 793: 148560. doi: 10.1016/j.scitotenv.2021.148560 [6] WU X, WANG W, ZHU L Z. Enhanced organic contaminants accumulation in crops: Mechanisms, interactions with engineered nanomaterials in soil[J]. Environmental Pollution, 2018, 240: 51-59. doi: 10.1016/j.envpol.2018.04.072 [7] ADAM V, WU Q, NOWACK B. Integrated dynamic probabilistic material flow analysis of engineered materials in all European countries[J]. NanoImpact, 2021, 22: 100312. doi: 10.1016/j.impact.2021.100312 [8] VALSAMI-JONES E, LYNCH I. NANOSAFETY. How safe are nanomaterials?[J]. Science, 2015, 350(6259): 388-389. doi: 10.1126/science.aad0768 [9] YANG Y, ZHANG C Q, HU Z Q. Impact of metallic and metal oxide nanoparticles on wastewater treatment and anaerobic digestion[J]. Environ Sci:Processes Impacts, 2013, 15(1): 39-48. doi: 10.1039/C2EM30655G [10] ZHANG F, WANG Z, VIJVER M G, et al. Prediction of the joint toxicity of multiple engineered nanoparticles: The integration of classic mixture models and In silico methods[J]. Chemical Research in Toxicology, 2021, 34(2): 176-178. doi: 10.1021/acs.chemrestox.0c00300 [11] BUNDSCHUH M, FILSER J, LÜDERWALD S, et al. Nanoparticles in the environment: Where do we come from, where do we go to?[J]. Environmental Sciences Europe, 2018, 30(1): 1-17. doi: 10.1186/s12302-017-0129-6 [12] TURAN N B, ERKAN H S, ENGIN G O, et al. Nanoparticles in the aquatic environment: Usage, properties, transformation and toxicity—a review[J]. Process Safety and Environmental Protection, 2019, 130: 238-249. doi: 10.1016/j.psep.2019.08.014 [13] LI F M, LIANG Z, ZHENG X, et al. Toxicity of nano-TiO2 on algae and the site of reactive oxygen species production[J]. Aquatic Toxicology, 2015, 158: 1-13. doi: 10.1016/j.aquatox.2014.10.014 [14] MILLER R J, LENIHAN H S, MULLER E B, et al. Impacts of metal oxide nanoparticles on marine phytoplankton[J]. Environmental Science & Technology, 2010, 44(19): 7329-7334. [15] MATOUKE M M, ELEWA D T, ABDULLAHI K. Binary effect of titanium dioxide nanoparticles (nTiO2) and phosphorus on microalgae (Chlorella ‘Ellipsoides Gerneck, 1907)[J]. Aquatic Toxicology, 2018, 198: 40-48. doi: 10.1016/j.aquatox.2018.02.009 [16] MAHANA A, GULIY O I, MEHTA S K. Accumulation and cellular toxicity of engineered metallic nanoparticle in freshwater microalgae: Current status and future challenges[J]. Ecotoxicology and Environmental Safety, 2021, 208: 111662. doi: 10.1016/j.ecoenv.2020.111662 [17] FAN W H, SHI Z W, YANG X P, et al. Bioaccumulation and biomarker responses of cubic and octahedral Cu2O micro/nanocrystals in Daphnia magna[J]. Water Research, 2012, 46(18): 5981-5988. doi: 10.1016/j.watres.2012.08.019 [18] CHEN X J, ZHU Y, YANG K, et al. Nanoparticle TiO2 size and rutile content impact bioconcentration and biomagnification from algae to daphnia[J]. Environmental Pollution, 2019, 247: 421-430. doi: 10.1016/j.envpol.2019.01.022 [19] XIAO B W, WANG X L, YANG J, et al. Bioaccumulation kinetics and tissue distribution of silver nanoparticles in zebrafish: The mechanisms and influence of natural organic matter[J]. Ecotoxicology and Environmental Safety, 2020, 194: 110454. doi: 10.1016/j.ecoenv.2020.110454 [20] LV X H, YANG Y, TAO Y, et al. A mechanism study on toxicity of graphene oxide to Daphnia magna: Direct link between bioaccumulation and oxidative stress[J]. Environmental Pollution, 2018, 234: 953-959. doi: 10.1016/j.envpol.2017.12.034 [21] CHEN J Y, DONG X, XIN Y Y, et al. Effects of titanium dioxide nano-particles on growth and some histological parameters of zebrafish (Danio rerio) after a long-term exposure[J]. Aquatic Toxicology, 2011, 101(3/4): 493-499. [22] HOU W C, WESTERHOFF P, POSNER J D. Biological accumulation of engineered nanomaterials: A review of current knowledge[J]. Environmental Science:Processes & Impacts, 2013, 15(1): 103-122. [23] DONG S P, WU Z H, WANG M J, et al. Assessing comparable bioconcentration potentials for nanoparticles in aquatic organisms via combined utilization of machine learning and toxicokinetic models[J]. SmartMat, 2023,4(3):e1155. [24] MUNA M, HEINLAAN M, BLINOVA I, et al. Evaluation of the effect of test medium on total Cu body burden of nano CuO-exposed Daphnia magna: A TXRF spectroscopy study[J]. Environmental Pollution, 2017, 231: 1488-1496. doi: 10.1016/j.envpol.2017.07.083 [25] 胡奕, 王艳龙, 林道辉. 纳米颗粒对大型蚤毒性效应的研究进展[J]. 科学通报, 2017, 62(24): 2734-2748. doi: 10.1360/N972017-00476 HU Y, WANG Y L, LIN D H. A review of the toxicity of nanoparticles to Daphnia magna[J]. Chinese Science Bulletin, 2017, 62(24): 2734-2748 (in Chinese). doi: 10.1360/N972017-00476
[26] SKJOLDING L M, KERN K, HJORTH R, et al. Uptake and depuration of gold nanoparticles in Daphnia magna[J]. Ecotoxicology, 2014, 23(7): 1172-1183. doi: 10.1007/s10646-014-1259-x [27] ZHENG Y F, NOWACK B. Meta-analysis of bioaccumulation data for nondissolvable engineered nanomaterials in freshwater aquatic organisms[J]. Environmental Toxicology and Chemistry, 2022, 41(5): 1202-1214. doi: 10.1002/etc.5312 [28] VIJVER M G, ZHAI Y J, WANG Z, et al. Emerging investigator series: The dynamics of particle size distributions need to be accounted for in bioavailability modelling of nanoparticles[J]. Environmental Science:Nano, 2018, 5(11): 2473-2481. doi: 10.1039/C8EN00572A [29] LI Z Q. Extracting spatial effects from machine learning model using local interpretation method: An example of SHAP and XGBoost[J]. Computers, Environment and Urban Systems, 2022, 96: 101845. doi: 10.1016/j.compenvurbsys.2022.101845 [30] PARIMBELLI E, BUONOCORE T M, NICORA G, et al. Why did AI get this one wrong?—Tree-based explanations of machine learning model predictions[J]. Artificial Intelligence in Medicine, 2023, 135: 102471. doi: 10.1016/j.artmed.2022.102471 [31] DOLK D, KRIDEL D, DINEEN J, et al. Model interpretation and explainability towards creating transparency in prediction models[C]//Proceedings of the Annual Hawaii International Conference on System Sciences, 2020. [32] ZHAO C M, WANG W X. Biokinetic uptake and efflux of silver nanoparticles in Daphnia magna[J]. Environmental Science & Technology, 2010, 44(19): 7699-7704. [33] ZHAO C M, WANG W X. Comparison of acute and chronic toxicity of silver nanoparticles and silver nitrate to Daphnia magna[J]. Environmental Toxicology and Chemistry, 2011, 30(4): 885-892. doi: 10.1002/etc.451 [34] KUEHR S, KAEGI R, MALETZKI D, et al. Testing the bioaccumulation potential of manufactured nanomaterials in the freshwater amphipod Hyalella azteca[J]. Chemosphere, 2021, 263: 127961. doi: 10.1016/j.chemosphere.2020.127961 [35] SIKDER M, EUDY E, CAI B, et al. Particle size determines the accumulation of platinum nanoparticles in the estuarine amphipod, Leptocheirus plumulosus[J]. Environmental Science:Nano, 2022, 9(2): 499-510. doi: 10.1039/D1EN00713K [36] ZHANG Y, ZHU L, ZHOU Y, et al. Accumulation and elimination of iron oxide nanomaterials in zebrafish (Danio rerio) upon chronic aqueous exposure[J]. Journal of Environmental Sciences, 2015, 30: 223-230. doi: 10.1016/j.jes.2014.08.024 [37] HEINLAAN M, MUNA M, JUGANSON K, et al. Exposure to sublethal concentrations of Co3O4 and Mn2O3 nanoparticles induced elevated metal body burden in Daphnia magna[J]. Aquatic Toxicology, 2017, 189: 123-133. doi: 10.1016/j.aquatox.2017.06.002 [38] GÜRKAN M, GÜRKAN S E, YıLMAZ S, et al. Comparative toxicity of alpha and gamma iron oxide nanoparticles in rainbow trout: Histopathology, hematology, accumulation, and oxidative stress[J]. Water, Air, & Soil Pollution, 2021, 232(2): 1-14. [39] CONG Y, BANTA G T, SELCK H, et al. Toxicity and bioaccumulation of sediment-associated silver nanoparticles in the estuarine polychaete, Nereis (Hediste) diversicolor[J]. Aquatic Toxicology, 2014, 156: 106-115. doi: 10.1016/j.aquatox.2014.08.001 [40] HULL M S, VIKESLAND P J, SCHULTZ I R. Uptake and retention of metallic nanoparticles in the Mediterranean mussel (Mytilus galloprovincialis)[J]. Aquatic Toxicology, 2013, 140/141: 89-97. doi: 10.1016/j.aquatox.2013.05.005 [41] RIVERO ARZE A, MOUNEYRAC C, CHATEL A, et al. Comparison of uptake and elimination kinetics of metallic oxide nanomaterials on the freshwater microcrustacean Daphnia magna[J]. Nanotoxicology, 2021, 15(9): 1168-1179. doi: 10.1080/17435390.2021.1994668 [42] SAYADI M H, PAVLAKI M D, MARTINS R, et al. Bioaccumulation and toxicokinetics of zinc oxide nanoparticles (ZnO NPs) co-exposed with graphene nanosheets (GNs) in the blackfish (Capoeta fusca)[J]. Chemosphere, 2021, 269: 128689. doi: 10.1016/j.chemosphere.2020.128689 [43] JOO H S, KALBASSI M R, YU I J, et al. Bioaccumulation of silver nanoparticles in rainbow trout (Oncorhynchus mykiss): Influence of concentration and salinity[J]. Aquatic Toxicology, 2013, 140/141: 398-406. doi: 10.1016/j.aquatox.2013.07.003 [44] BENAVIDES M, FERNÁNDEZ-LODEIRO J, COELHO P, et al. Single and combined effects of aluminum (Al2O3) and zinc (ZnO) oxide nanoparticles in a freshwater fish, Carassius auratus[J]. Environmental Science and Pollution Research, 2016, 23(24): 24578-24591. doi: 10.1007/s11356-016-7915-3 [45] ZHAO J, WANG Z Y, LIU X Y, et al. Distribution of CuO nanoparticles in juvenile carp (Cyprinus carpio) and their potential toxicity[J]. Journal of Hazardous Materials, 2011, 197: 304-310. doi: 10.1016/j.jhazmat.2011.09.094 [46] CONG Y, BANTA G T, SELCK H, et al. Toxic effects and bioaccumulation of nano-, micron- and ionic-Ag in the polychaete, Nereis diversicolor[J]. Aquatic Toxicology, 2011, 105(3/4): 403-411. [47] ATES M, ARSLAN Z, DEMIR V, et al. Accumulation and toxicity of CuO and ZnO nanoparticles through waterborne and dietary exposure of goldfish (Carassius auratus)[J]. Environmental Toxicology, 2015, 30(1): 119-128. doi: 10.1002/tox.22002 [48] BEN BRAHIM A, LIMAM M. Ensemble feature selection for high dimensional data: A new method and a comparative study[J]. Advances in Data Analysis and Classification, 2018, 12(4): 937-952. doi: 10.1007/s11634-017-0285-y [49] JIANG C S, ZHAO P P, LI W H, et al. In silico prediction of chemical neurotoxicity using machine learning[J]. Toxicology Research, 2020, 9(3): 164-172. doi: 10.1093/toxres/tfaa016 [50] MURATOV E N, BAJORATH J, SHERIDAN R P, et al. Correction: QSAR without borders[J]. Chemical Society Reviews, 2020, 49(11): 3716. doi: 10.1039/D0CS90041A [51] 丁蕊, 陈景文, 于洋, 等. 基于集成学习算法构建有机化学品鱼体生物富集因子的QSAR预测模型[J]. 环境化学, 2021, 40(5): 1295-1304. doi: 10.7524/j.issn.0254-6108.2021011304 DING R, CHEN J W, YU Y, et al. Using ensemble learning algorithms to develop QSAR models on bioconcentration factors of organic chemicals in multispecies fish[J]. Environmental Chemistry, 2021, 40(5): 1295-1304 (in Chinese). doi: 10.7524/j.issn.0254-6108.2021011304
[52] OUASSIL N, PINALS R L, del BONIS-O’DONNELL J T, et al. Supervised learning model predicts protein adsorption to carbon nanotubes[J]. Science Advances, 2022, 8(1): eabm0898. doi: 10.1126/sciadv.abm0898 [53] JIANG D J, WU Z X, HSIEH C Y, et al. Could graph neural networks learn better molecular representation for drug discovery? A comparison study of descriptor-based and graph-based models[J]. Journal of Cheminformatics, 2021, 13(1): 1-23. doi: 10.1186/s13321-020-00477-w [54] SVETNIK V, WANG T, TONG C, et al. Boosting: an ensemble learning tool for compound classification and QSAR modeling[J]. Journal of Chemical Information and Modeling, 2005, 45(3): 786-799. doi: 10.1021/ci0500379 [55] SHERIDAN R P, WANG W M, LIAW A, et al. Extreme gradient boosting as a method for quantitative structure–activity relationships[J]. Journal of Chemical Information and Modeling, 2016, 56(12): 2353-2360. doi: 10.1021/acs.jcim.6b00591 [56] WU Z X, ZHU M F, KANG Y, et al. Do we need different machine learning algorithms for QSAR modeling?A comprehensive assessment of 16 machine learning algorithms on 14 QSAR data sets[J]. Briefings in Bioinformatics, 2021, 22(4): bbaa321. doi: 10.1093/bib/bbaa321 [57] HUANG Y, LI X H, XU S J, et al. Quantitative structure–activity relationship models for predicting inflammatory potential of metal oxide nanoparticles[J]. Environmental Health Perspectives, 2020, 128(6): 67010. doi: 10.1289/EHP6508 [58] JAWORSKA J, NIKOLOVA-JELIAZKOVA N, ALDENBERG T. QSAR applicability domain estimation by projection of the training set in descriptor space: A review[J]. Alternatives to Laboratory Animals, 2005, 33(5): 445-459. doi: 10.1177/026119290503300508 [59] GAO Y F, XIE Z C, FENG J F, et al. Different factors determined the toxicokinetics of organic chemicals and nanomaterials exposure to zebrafish (Danio Rerio)[J]. Ecotoxicology and Environmental Safety, 2019, 186: 109810. doi: 10.1016/j.ecoenv.2019.109810 [60] ZHU X S, TIAN S Y, CAI Z H. Toxicity assessment of iron oxide nanoparticles in zebrafish (Danio rerio) early life stages[J]. PLoS One, 2012, 7(9): e46286. doi: 10.1371/journal.pone.0046286 [61] 李宁静, 许喆, 姚烘烨, 等. 粒径对纳米氧化锌的生物累积和氧化应激的影响[J]. 科学通报, 2021, 66(24): 3219-3226. doi: 10.1360/TB-2021-0186 LI N J, XU Z, YAO H Y, et al. Impact of particle size of zinc oxide nanoparticles on its bioaccumulation and oxidative stress responses[J]. Chinese Science Bulletin, 2021, 66(24): 3219-3226 (in Chinese). doi: 10.1360/TB-2021-0186
[62] KLEANDROVA V V, LUAN F, GONZÁLEZ-DÍAZ H, et al. Computational ecotoxicology: Simultaneous prediction of ecotoxic effects of nanoparticles under different experimental conditions[J]. Environment International, 2014, 73: 288-294. doi: 10.1016/j.envint.2014.08.009 [63] ROBINSON R L M, SARIMVEIS H, DOGANIS P, et al. Identifying diverse metal oxide nanomaterials with lethal effects on embryonic zebrafish using machine learning[J]. Beilstein Journal of Nanotechnology, 2021, 12: 1297-1325. doi: 10.3762/bjnano.12.97 [64] ZHU X S, WANG J X, ZHANG X Z, et al. Trophic transfer of TiO2 nanoparticles from daphnia to zebrafish in a simplified freshwater food chain[J]. Chemosphere, 2010, 79(9): 928-933. doi: 10.1016/j.chemosphere.2010.03.022 [65] YUN X Y, LEWIS A J, STEVENS-KING G, et al. Bioaccumulation of per- and polyfluoroalkyl substances by freshwater benthic macroinvertebrates: Impact of species and sediment organic carbon content[J]. Science of the Total Environment, 2023, 866: 161208. doi: 10.1016/j.scitotenv.2022.161208 [66] ROSENKRANZ P, CHAUDHRY Q, STONE V, et al. A comparison of nanoparticle and fine particle uptake by Daphnia magna[J]. Environmental Toxicology and Chemistry, 2009, 28(10): 2142-2149. doi: 10.1897/08-559.1 [67] ABDOLAHPUR MONIKH F, CHUPANI L, ARENAS-LAGO D, et al. Particle number-based trophic transfer of gold nanomaterials in an aquatic food chain[J]. Nature Communications, 2021, 12: 899. doi: 10.1038/s41467-021-21164-w [68] ATES M, DEMIR V, ARSLAN Z, et al. Chronic exposure of tilapia (Oreochromis niloticus) to iron oxide nanoparticles: Effects of particle morphology on accumulation, elimination, hematology and immune responses[J]. Aquatic Toxicology, 2016, 177: 22-32. doi: 10.1016/j.aquatox.2016.05.005 -


 下载:
下载: