-
油基钻屑是油气开采过程中产生的附着大量油基钻井液的钻屑。在油气开采过程中,油基钻屑的无害化处理也是必不可少的环节[1]。油基钻屑中主要的污染物包括石油烃、多氯联苯和重金属[2]。目前,针对油基钻屑的石油烃的处理方法主要包括物理、化学以及生物三大类。其中,利用生物技术尤其是微生物降解油基钻屑中的石油烃因其处理步骤简单、成本低、环境友好等优点,越来越受到研究者的关注[3]。
微生物降解油基钻屑中石油烃主要是利用自然界中以石油烃为营养碳源的细菌、真菌或酵母,通过其体内代谢过程将石油烃分解为无机物质。该方法是一种从根本上降解油基钻屑中石油烃不产生二次污染的环境友好型技术,但受限于油基钻屑中石油烃类组成及性质不统一、单一微生物无法完成复杂石油烃的降解、处置周期长等[1, 4]。相关研究多集中在以石油污泥为微生物来源,利用筛选培养基筛选其中可降解石油烃的微生物。研究者从油基钻屑中分离出了苏云金芽孢杆菌、嗜盐芽孢杆菌和其他一些嗜盐细菌[5],但这些菌株处理油基钻屑的能力却未被探究。单海霞等[6]利用收集到的3种石油降解菌处理油基钻屑,发现在添加除油菌OBT 180 d后,钻屑中油含量从134.7 g·kg−1降至34.6 g·kg−1。陈政阳等[7]从四川省珙县油基泥浆钻井岩屑中筛选获得2株可高效降解油基钻屑的细菌 (球形芽孢杆菌和枯草芽孢杆菌) ,对石油的降解率分别为46.08%、48.94%,但也发现部分长链烷烃、多环芳烃等仍未被有效降解。此外,研究发现添加能降解污染物的微生物群落可通过群落中不同个体之间的协同作用增强微生物法处理油基钻屑的效果。REZAEI等[8]通过添加从石油污染的盐渍土中富集的嗜盐微生物群落,可有效降解油基钻屑中的总石油烃。OKPARANMA等[9]发现枯草芽孢杆菌和绿脓杆菌可以有效降解油基钻屑中的芳香化合物。蔡静等[10]使用可降解石油的芽孢杆菌和能恢复生态的固氮菌等菌群组成的微生物复合菌剂处理油基钻屑,48 d后最高的降解率可分别达到86.7%。
微生物正常的生长和代谢均需依赖碳源、氮源、无机盐、生长因子和水。其中,最主要的是碳源和氮源,碳源主要参与微生物代谢产物的合成,氮源为微生物生长提供蛋白和核酸,对其生长发酵具有重要的意义,也可用于合成含氮代谢产物。微生物处理油基钻屑主要是依赖微生物以石油烃类化合物为主要碳源,同时需要补充合适的氮源促进微生物生长,从而取得良好的降解效果。KALANTARY等[11]通过研究添加3种常量营养素 (N、P和K) 对微生物降解污染土壤中多环芳烃的影响,发现3种常量营养素对降解率影响大小为:N > K > P。也有大量研究结果表明,添加氮磷钾营养液,可有效促进石油烃的降解,显著提高微生物对石油污染土壤的修复效果[12-14]。因此,本研究拟通过探究添加不同的氮源对细菌群落及其降解油基钻屑中石油烃的影响,获得油基钻屑降解过程中的最适氮源,以期为生物法高效处理油基钻屑提供参考。
-
试剂:(NH4)2HPO4、氯化铵NH4Cl、硝酸钾KNO3、硫酸铵(NH4)2SO4购自国药集团化学试剂有限公司;酵母粉和蛋白胨购自Oxoid。海底泥取自南海海泥 (取样深度为25 m) 。油基钻屑为白油基钻井液离心分离后的固相,取自于海上某钻井平台,并保藏于-20 ℃。油基钻屑样品的含水量、含油量及有机物组分参考已有文献报道的方法测定[15-16]。该油基钻屑的含水量为12.51%,含油量为5.34%,有机物质量分数如表1所示。
-
取10.00 g油基钻屑样品置于三角瓶中,按照质量分数20∶1添加不同的氮源。将5.00 g南海海泥样品用50 mL生理盐水重悬,37 ℃孵育1 h后,菌液按2%的添加量,加入对照和含有不同氮源的油基钻屑中。25 ℃,200 r·min−1 培养,间隔7 d取样,整个过程中共计取样5次。样品存于无菌取样袋中,并保存于−20 ℃,待使用。样品具体信息见表2。
-
样本中微生物群落总DNA提取采用试剂盒完成 (E.Z.N.A.® soil DNA kit,Omega Bio-tek) ,检测质量合格后,使用338F (5’-ACTCCTACGGGAGGCAGCAG-3’) 和806R (5'-GGACTACHVGGGTWTCTAAT-3’) 对16S rRNA基因V3-V4区进行PCR扩增。在PCR产物纯化、检测和定量后,使用NEXTFLEX Rapid DNA-Seq Kit进行建库,并利用Illumina Miseq PE300平台进行测序[17]。
-
测序得到的原始数据,使用Fastp软件进行质控后,利用Flash软件进行拼接,最后使用Uparse软件,根据97%的相似度阈值对序列进行OTU聚类并剔除嵌合体。
样本中细菌群落测序后,物种注释与评估、细菌群落多样性分析、物种差异分析和细菌群落功能预测均使用美吉生物云平台完成 (www.majorbio.com) [18]。29个样本中细菌群落测序原始数据已提交至NCBI数据库,登录号为:PRJNA1030500。
-
样品中石油烃含量测定参考文献[19]完成。即1.00 g样品中加入20 mL二氯甲烷后超声处理15 min,接着机械振荡萃取30 min (250 r·min−1) ,重复3次后,合并有机相;有机相利用无水Na2SO4干燥后,采用GC-MS分析样品中石油烃的种类,采用GC对样品中的各石油烃的含量进行定量分析。测试条件为:色谱柱Agilgent HP-5、进样口温度300 ℃、升温程序 (40 ℃下保持0.5 min,然后以15 ℃·min−1的速率升至150 ℃,以10 ℃·min−1的速率升至290 ℃保持5 min) 、检测器FID、检测器温度290 ℃;每个样品测定3次,结果表示为平均值±标准方差。
-
含有细菌群落的油基钻屑中添加不同氮源处理不同时间后,样本中细菌群落在门水平和属水平上的物种组成如图1所示,主要物种在门水平和属水平上的相对丰度如表3所示。在门水平上,添加无机氮源 ((NH4)2HPO4、NH4Cl、KNO3和(NH4)2SO4) 后,细菌主要是变形菌门 (Proteobacteria) 和放线菌门 (Actinobacteriota) 的菌株,其总相对丰度在95%以上;添加有机氮源 (酵母粉和蛋白胨) 后,细菌主要是变形菌门 (Proteobacteria) 、放线菌门 (Actinobacteriota) 和厚壁菌门 (Firmicutes) 的菌株,其总相对丰度在95%以上;此外,除添加(NH4)2HPO4样本中细菌主要属于变形菌门 (Proteobacteria) ,添加酵母粉的样本中细菌主要属于变形菌门 (Proteobacteria) 和放线菌门 (Actinobacteriota) ,其他氮源添加的样品中细菌均主要属于放线菌门 (Actinobacteriota) (图1(a)) 。在属水平上,添加(NH4)2HPO4的样品中细菌主要归属于不动杆菌属、红球菌属、短波单胞菌属和其他根瘤菌属 (Allorhizobium) ,总丰度在90%以上;NH4Cl的样品中细菌主要归属于不动杆菌属、红球菌属、新鞘氨醇杆菌属、短波单胞菌属、其他根瘤菌属和柄杆菌属,总丰度在95%以上;添加KNO3的样品中细菌主要归属于不动杆菌属、红球菌属、新鞘氨醇杆菌属、其他根瘤菌属、德沃斯氏菌属 (Devosia) 和鞘氨醇单胞菌,总丰度在90%以上;添加(NH4)2SO4的样品中细菌主要归属于不动杆菌属、红球菌属、新鞘氨醇杆菌属、短波单胞菌属、柄杆菌属和鞘氨醇单胞菌,总丰度在90%以上;添加酵母粉的样品中细菌主要归属于不动杆菌属、红球菌属、短波单胞菌属和芽孢杆菌属,总丰度在90%以上;添加蛋白胨的样品中细菌主要归属于不动杆菌属、短波单胞菌属、其他根瘤菌属和芽孢杆菌属,总丰度在90%以上 (图1(b)) 。
此外,由图1和表3可知,添加不同氮源的油基钻屑在处理过程中主要细菌群落是处于演替变化的。在属水平上,添加不同氮源的油基钻屑样本中不动杆菌属的细菌均随着处理时间的增加而减少。添加(NH4)2HPO4的样品由前期主要细菌群落是不动杆菌属,逐渐演变为后期的红球菌属、不动杆菌属和短波单胞菌属;添加NH4Cl的样品由前期主要细菌群落是不动杆菌属、红球菌属、新鞘氨醇杆菌属和短波单胞菌属,逐渐演变为后期的不动杆菌属、短波单胞菌属、新鞘氨醇杆菌属、红球菌属、其他根瘤菌属、柄杆菌属和德沃斯氏菌属;添加KNO3的样品由前期主要细菌群落是不动杆菌属、新鞘氨醇杆菌属、红球菌属和德沃斯氏菌属,逐渐演变为后期的新鞘氨醇杆菌属、其他根瘤菌属、德沃斯氏菌属、不动杆菌属和红球菌属柄杆菌属;添加(NH4)2SO4的样品由前期主要细菌群落是不动杆菌属、红球菌属和新鞘氨醇杆菌属,逐渐丰富为后期的短波单胞菌属、不动杆菌属、红球菌属、柄杆菌属、新鞘氨醇杆菌属和其他根瘤菌属;添加酵母粉的样品由前期主要细菌群落是不动杆菌属和芽孢杆菌属,逐渐丰富为后期的红球菌属、不动杆菌属、短波单胞菌属和芽孢杆菌属;添加蛋白胨的样品中细菌群落的组成变化不大,主要变化表现在不动杆菌属细菌减少,而短波单胞菌属增加。
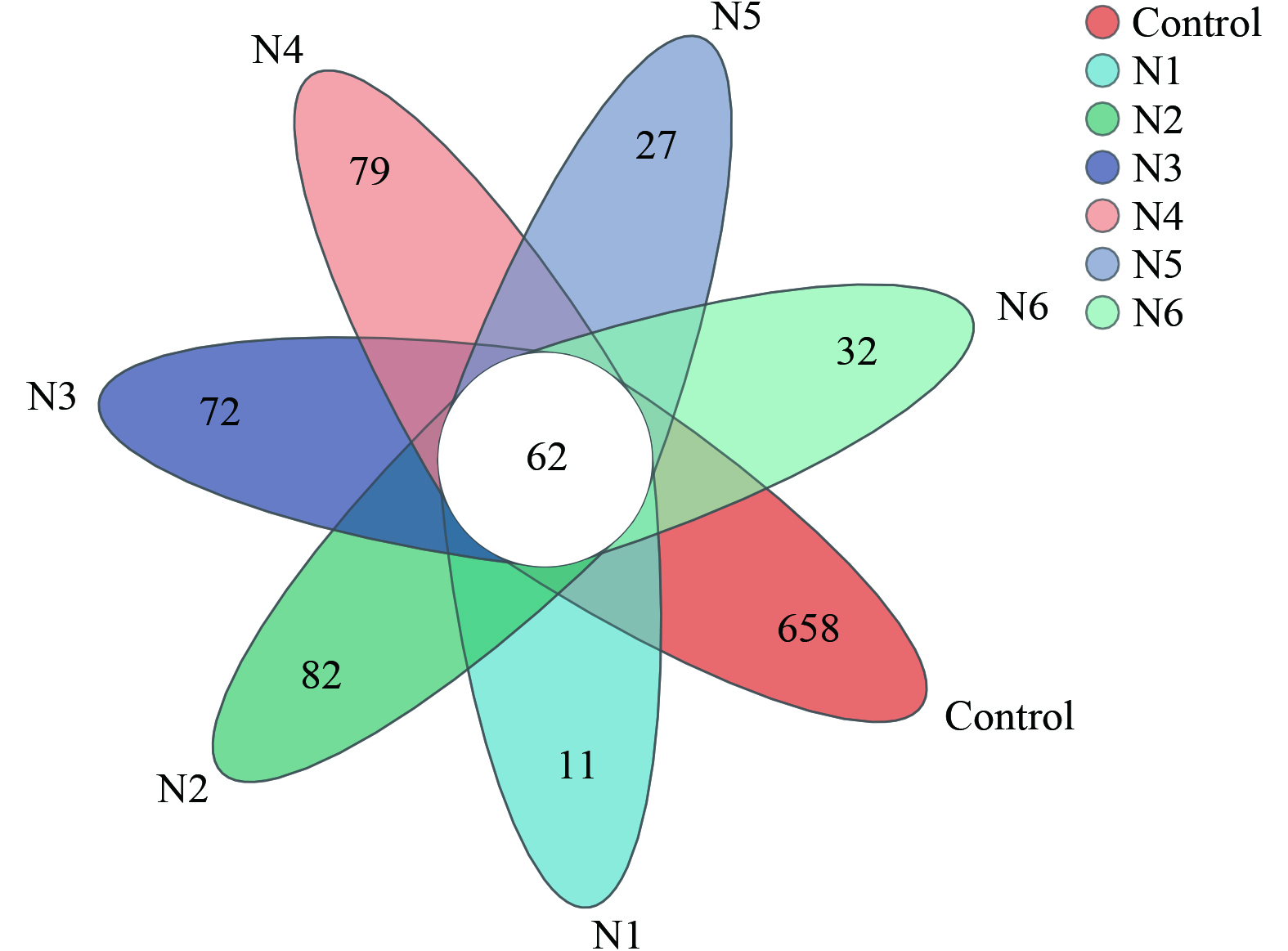
添加不同氮源后油基钻屑样本中细菌群落文氏图见图2。不同样本中所共有的物种 (OTU) 数目为62;无机氮源添加的样本中细菌群落的独有物种数目均超过有机氮源的样本。除此之外,添加(NH4)2HPO4 (N1)、NH4Cl (N2)、KNO3 (N3)、(NH4)2SO4 (N4)、酵母粉 (N5) 和蛋白胨 (N6) 的6种氮源处理28 d后样品中细菌群落的OTU数分别为485、406、436、218和207,表明不同氮源的添加对样本中细菌群落影响较大,其中无机氮源添加微生物群落丰富度高于有机氮源的添加,也说明氮源可能会在功能细菌的生长和代谢中发挥重要作用。
-
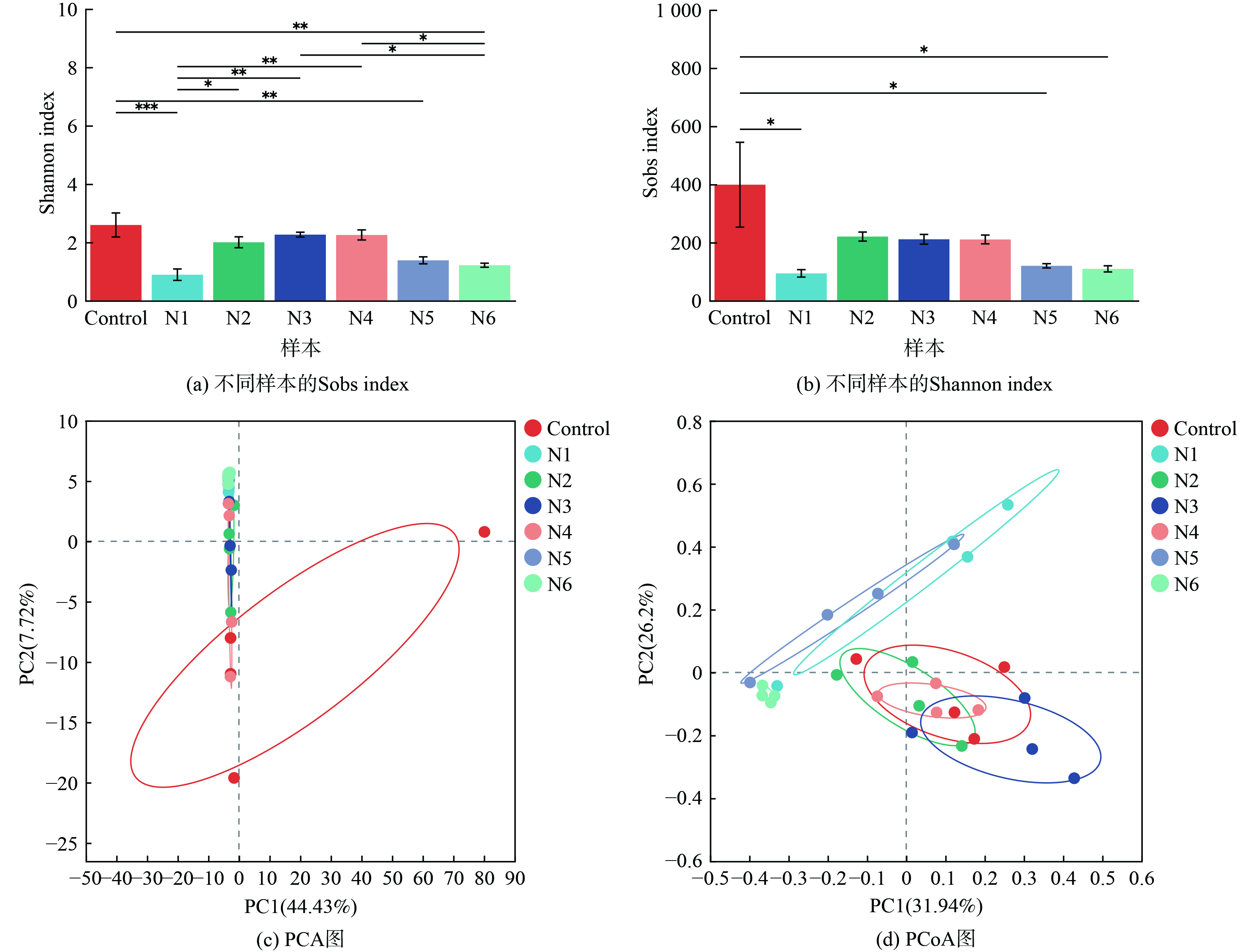
1) Alpha多样性分析结果。不同样本中细菌群落的丰富度和多样性可通过Alpha多样性指数来反映。Sobs和Shannon指数分别用于分析细菌群落的丰富度 (richness) 和多样性 (diversity) 。本研究中不同处理的样本中微生物群落的Alpha多样性指数见表4。添加(NH4)2HPO4、NH4Cl、酵母粉和蛋白胨的样本中微生物群落多样性和丰富度均随着处理时间的增加而呈现先降低后上升趋势;而在添加KNO3和(NH4)2SO4的样本中,微生物群落多样性和丰富度均随着处理时间的增加而呈现先升高后降低的趋势,这表明外加氮源对微生物群落多样性和丰富度的影响较大。
图3(a)和图3(b)进一步列出了在油基钻屑中不添加氮源和添加不同氮源处理28 d后样本中细菌群落Sob指数和Shannon指数的差异。总体来看,不同样本中细菌群落丰富度由高到低是Control > N2 > N4 > N3 > N5 > N6 > N1,且添加不同氮源的样品中微生物群落丰富度无差异;不同样本中细菌群落多样性由高到低是Control > N3 > N4 > N2 > N5 > N6 > N1,且添加无机氮源 (N3、N4) 和有机氮源 (N5、N6) 的样品中微生物群落多样性存在显著差异。
2) Beta多样性分析结果。Beta多样性分析可表征不同样本中细菌群落组成差异。图3(c)和图3(d)列出了本研究中的原始 (Control) 、不添加氮源 (N1) 和添加不同氮源 (N2~N6) 的7个样本在属水平上的主成分分析图 (Principal Component Analysis,PCA分析) 和主坐标分析图 (PCoA分析,Principal co-ordinates analysis) 。样本物种组成越相似,反映在PCA和PCoA图中的距离越近。因此,本研究中Control与添加有机氮源的样本 (N5、N6) 中细菌群落组成差异最大;分别添加有机氮源样本和无机氮源样本的细菌群落组成差异最小;N1与N5样本的细菌群落组成最为相似,这表明酵母粉和(NH4)2HPO4的添加更大程度地改变了原始的细菌群落。
-
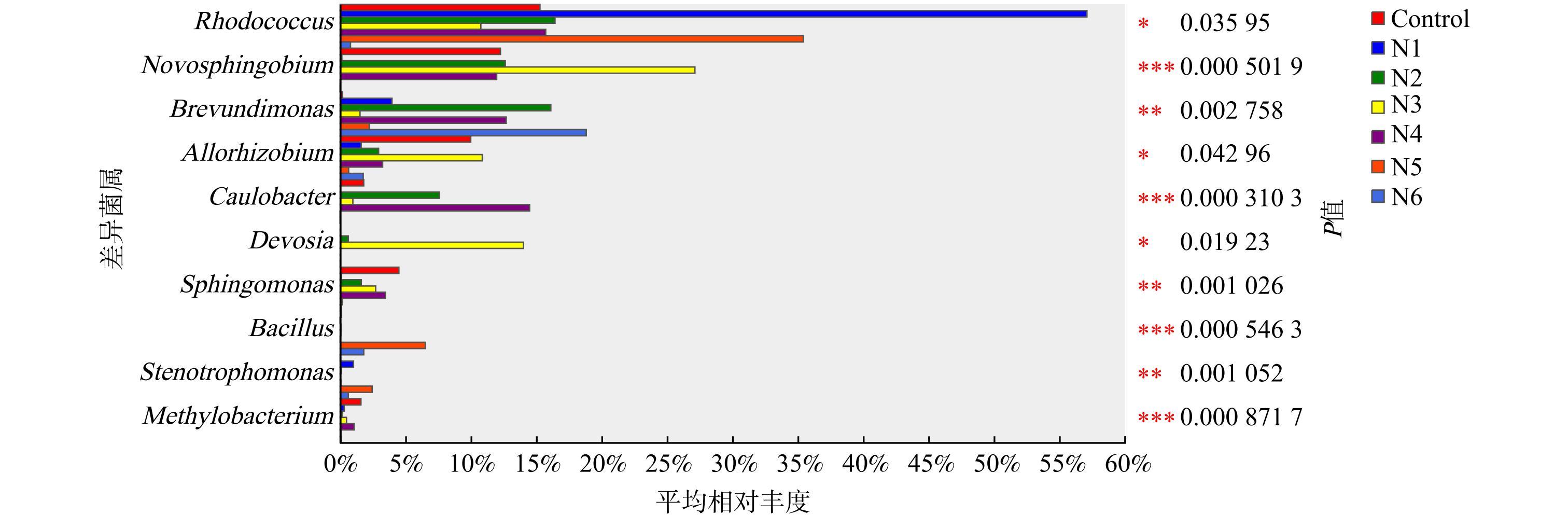
添加不同氮源的样本中细菌群落在属水平上的差异结果如图4所示。不同样本中差异菌株按丰度排序主要是红球菌属 (0.01 ≤ P ≤ 0.05) 、新鞘氨醇杆菌属 (P ≤ 0.001) 、短波单胞菌属 (0.001≤ P ≤ 0.01) 、其他根瘤菌属 (0.01 ≤ P ≤ 0.05) 、柄杆菌属 (P ≤ 0.001) 、德沃斯氏菌属 (0.01 ≤ P ≤ 0.05) 、鞘氨醇单胞菌 (0.001≤ P ≤ 0.01) 、芽孢杆菌属 (P ≤ 0.001) 、寡养单胞菌属 (0.001≤ P ≤ 0.01) 和甲基杆菌属 (P ≤ 0.001) 。其中,添加(NH4)2HPO4有利于红球菌属的生长;添加KNO3有利于新鞘氨醇杆菌属、其他根瘤菌属和柄杆菌属菌株的生长;(NH4)2SO4的添加可促进柄杆菌属和鞘氨醇单胞菌属细菌的生长;有机氮源酵母粉的添加可促进红球菌属、芽孢杆菌属和寡养单胞菌属的生长。
-
FAPROTAX功能预测是基于人工构建的FAPROTAX数据库,可将原核生物映射到代谢或其他生态相关功能,其功能集中在海洋和湖泊生物地球化学,特别是硫、氮、氢和碳循环。因此,该方法更适合环境样本中微生物群落功能的预测[20]。本研究中微生物群落主要功能包括化能异养 (Chemoheterotrophy) 、需氧化能异养 (Aerobic chemoheterotrophy) 、芳香族化合物降解 (Aromatic compound degradation) 、尿素分解 (Ureolysis) 、芳烃降解 (Aromatic hydrocarbon degradation) 、碳氢化合物降解 (Hydrocarbon degradation) 、脂肪族非甲烷碳氢化合物降解(Aliphatic non-methane hydrocarbon degradation)等65个功能。其中,排名前30的功能丰度占总功能的95%以上。图5为丰度排名前30名的微生物群落功能Heatmap图。化能异养和需氧化能异养2个功能的在所有样本中均丰度较高,这说明油基钻屑的细菌群落处理过程中以好氧菌和异养菌为主。与未加氮源的处理样品相比,6种氮源的添加抑制了细菌群落对含硫化合物分解代谢;外加蛋白胨作为氮源可促进芳香化合物的降解,但其对碳氢化合物的降解能力降低;添加(NH4)2HPO4和酵母粉更有利于碳氢化合物降解,同时也保留了其降解芳香化合物的能力;除添加(NH4)2HPO4外,其余5种氮源的添加均增加了细菌群落对含氮化物的作用。此外,与细菌群落多样性变化不同,添加不同氮源的样本中细菌群落的功能随着处理时间的增加而呈不同变化趋势。外加6种不同氮源处理的样品中细菌群落对含硫化合物分解代谢能力随着处理时间的增加而呈现降低趋势,且大多数在7 d后达到稳定;除了外加蛋白胨作为氮源的样本中细菌群落对芳香化合物的降解随着处理时间的增加保持不变,其他外加氮源的样本中细菌群落对芳香化合物的降解均随着处理时间的增加略有下降;外加6种不同氮源处理的样品中细菌群落对碳氢化合物的降解能力随着处理时间的增加而呈现先增后减的趋势。总体来看,多数无机氮源的添加可促进氢和碳循环,而有机氮源 (特别是蛋白胨) 的添加可促进硫、氮的循环。
-
利用GC/MS分析油基钻屑中的石油烃种类,初步确定在本次样本中,原始油基钻屑样本中主要的石油烃种类包括:C15H32、C16H34、C17H36、C18H38、C19H40、C20H42 (图6(a)) 。通过向钻屑中添加不同氮源,测定不同处理下样本中主要烷烃的降解情况,发现添加有机氮源,尤其在添加酵母粉处理组中,6种烷烃含量均有降低 (图6(b)) ;在添加无机氮源处理组中,(NH4)2HPO4和NH4Cl处理下烷烃的降解效果明显,尤其添加(NH4)2HPO4处理组中,对C17H36的降解效果比其余处理组效果明显。通过图6(b)结果可知,添加不同氮源,均可以对钻屑中的石油烃有降解效果,但为进一步明确2组效果较好处理组的详细处理时间,以及在处理过程中,对应不同处理时间下微生物种类的变化,对(NH4)2HPO4和酵母粉两组处理下的详细变化进一步分析。
-
在(NH4)2HPO4和酵母粉两组处理下,加入对应的外源氮源后,间隔7 d取样,测定处理样本中不同取样时期下样本中石油烃浓度的变化,结果如图7所示。图7(a)表明,在添加(NH4)2HPO4样本中,石油烃降解明显,且随着时间的增加,降解率逐步提升;图7(b)表明,在添加酵母粉样本中,石油烃浓度随着时间的增加而增大。然而,添加(NH4)2HPO4的样本中细菌群落的降解效率明显高于添加酵母粉的样本组,这表明无机氮源(NH4)2HPO4可有效促进细菌群落对石油烃类化合物的降解。这与细菌群落功能预测结果一致。
-
图8为本研究中样本、主要细菌菌属和主要化学成分 (6种石油烃) 之间的关系。其主要用来反映菌群与环境因子之间关系,图中化学成分箭头的长短代表化学成分对于物种数据影响程度的大小。本研究中对于添加(NH4)2HPO4和酵母粉2种氮源的油基钻屑微生物处理过程中,6种石油烃浓度变化与样本中细菌群落分布和细菌菌属丰度变化均密切相关。图中从样本点和主要细菌菌属点向数量型化学成分的箭头做投影,投影点距离原点的距离代表化学成分对样本群落分布和细菌菌属相对影响的大小。因此,添加(NH4)2HPO4样本中,样本中细菌群落分布在处理28 d时 (ZX4_2样本) 对C15H32、C16H34、C17H36、C18H38和C19H40浓度影响最大,其中短波单胞菌属和寡养单胞菌属细菌的影响最为明显 (图8(a)和图8(b)) 。而添加酵母粉样本中,样本中细菌群落分布在处理28 d时 (ZX4_6样本) 对C15H32、C16H34、C17H36、C18H38和C19H40浓度影响最大,其中叶杆菌属及短波单胞菌属细菌的影响最大 (图8(c)和图8(d)) 。而添加2种氮源的样本中均是Control样本中细菌群落分布和叶杆菌属细菌对C20H42浓度影响最大 (图8(b)和图8(d)) 。此外,添加无机氮源 ((NH4)2HPO4) 有利于短波单胞菌属的细菌在多数石油烃类化合物的代谢中发挥作用;添加有机氮源 (酵母粉) 可强化叶杆菌属细菌对石油烃类化合物的降解。综上所述,细菌群落降解油基钻屑中石油烃主要影响处理最后阶段细菌群落的分布和叶杆菌属及短波单胞菌属细菌。
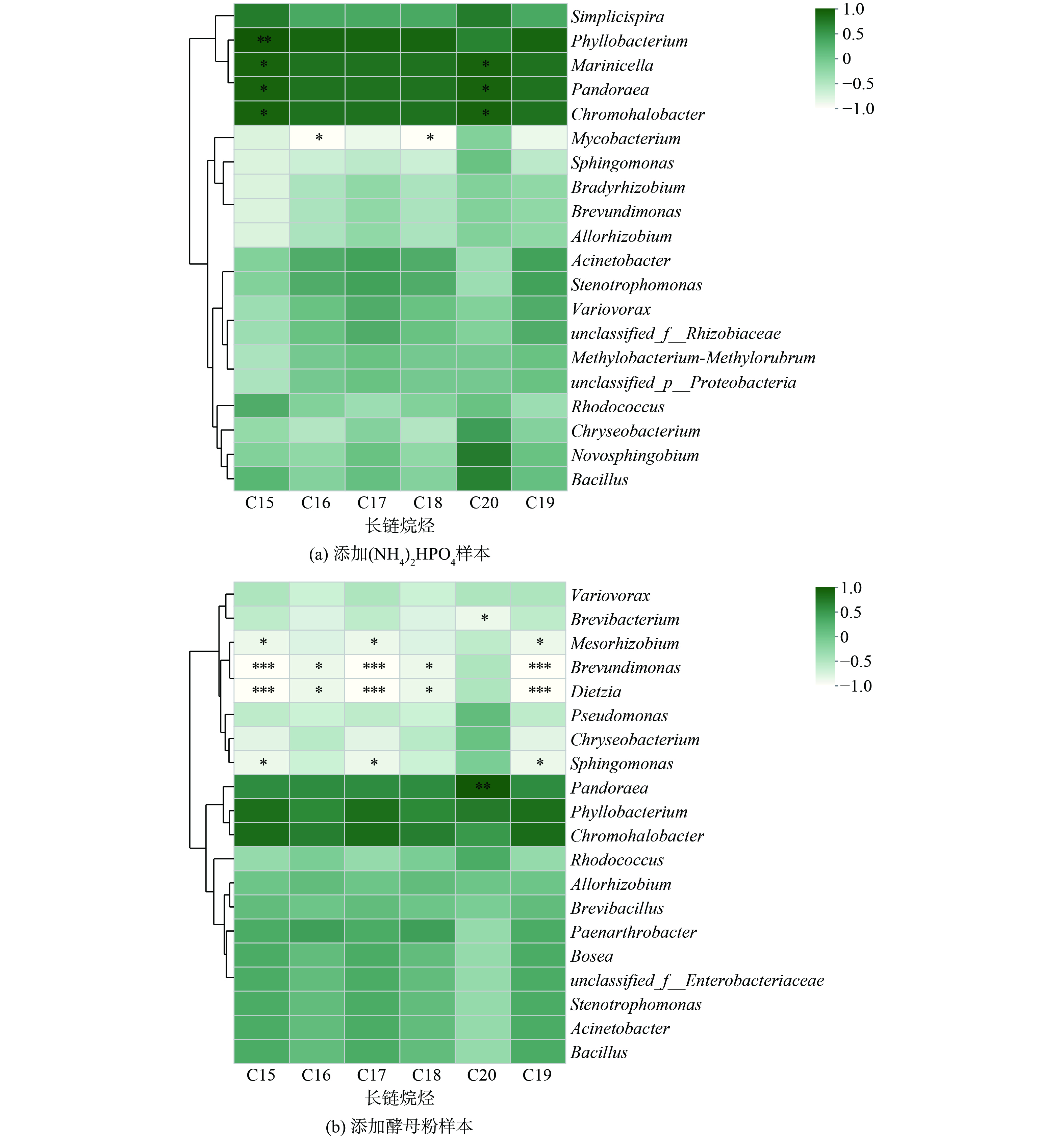
为进一步深入了解细菌菌属与化学成分变化之间的关系,本研究分析了油基钻屑处理过程中主要细菌菌属 (丰度前20的细菌) 与所测6种长链烷烃含量降低率的相关性,结果如图9所示。添加(NH4)2HPO4样本中,与C15H32和C20H42显著正相关的菌株是叶杆菌属 (0.001≤ P ≤ 0.01) 、杆状海胞菌属 (Marinicella,0.01≤P≤0.05) 、潘多拉菌属 (Pandoraea,0.01≤P≤0.05) 和色盐杆菌属 (Chromohalobacter,0.01≤P≤0.05) ;与C16H34和C18H38显著负相关的菌株是分枝杆菌 (Mycobacterium,0.01≤P≤0.05) 。添加酵母粉的样本中细菌菌属与化学成分变化之间具有更加丰富的相关性,与C15H32、C17H36和C19H40显著负相关的菌属是中慢生根瘤菌属 (Mesorhizobium,0.01 ≤ P ≤ 0.05) 和鞘氨醇单胞菌 (0.01 ≤ P ≤ 0.05) ,极显著负相关的菌属是短波单胞菌属 (P ≤ 0.001) 和迪茨氏菌属 (Dietzia,P ≤ 0.001) ;C16H34和C18H38显著负相关的菌属是短波单胞菌属 (0.01 ≤ P ≤ 0.05) 和迪茨氏菌属 (0.01 ≤ P ≤ 0.05) ;与C20H42显著负相关的菌属是短杆菌属 (0.01 ≤ P ≤ 0.05) ,显著正相关的菌属是潘多拉菌属 (0.001≤P≤0.01) 。结合化学成分变化结果,推测短波单胞菌属、寡养单胞菌属、鞘氨醇单胞菌属、迪茨氏菌属和短杆菌属的细菌对油基钻屑中石油烃的降解起着重要作用。
-
1) 不同氮源添加对油基钻屑中细菌多样性的影响。土壤类样本中微生物群落动态的变化是由于样本本身理化特性及外界环境施加的选择性力造成的。本研究中通过在油基钻屑样本中添加不同氮源后,发现不同氮源的添加一定程度上降低了油基钻屑中细菌群落的多样性和丰富度。这与孙思路[21]的研究结果基本一致。该研究发现施加氮素 (铵态氮和硝态氮) 会减少土壤细菌生物量,影响土壤微生物群落结构,从而影响16S rRNA基因丰度。主要原因可能是本研究中探究的细菌群落不仅包括油基钻屑本身含有的细菌还包括外加的南海海泥样本中的细菌,而这些外加的细菌只有适应了所在环境才能生长和代谢。此外,本研究中添加NH4Cl、KNO3、(NH4)2SO4的样本中细菌群落多样性和丰富度均高于添加(NH4)2HPO4、酵母粉和蛋白胨的样本。目前,针对不同氮源氮源对污染土壤特别是油基钻屑中细菌群落的多样性和丰富度研究较少,但研究者发现草地土壤细菌随着外源氮输入量的增加,其多样性细菌多样性明显下降,群落结构也发生改变[22]。进一步不仅可研究外加氮源量对污染土壤微生物群落组成、多样性和丰富度的影响,还可将不同氮源与磷、钾等营养物复配探究其对样本中微生物群落结构和修复功能的影响。
2) 细菌群落对石油烃降解的影响。生物法修复石油污染土壤主要依赖的是微生物。已有研究[23]和本实验室初步探究结果发现南海海泥样品中细菌群落的多样性较为丰富且其代谢过程主要涉及“氮循环”、“硫循环”和“碳循环”,可在环境修复中发挥重要作用;而且海洋来源的微生物更有利其在海上油田产生的油基钻屑中发挥作用,故本研究中使用南海海泥样本作为本研究中功能微生物的来源。目前,多数研究也集中在从特殊环境中筛选获得高效降解石油主要成分的菌株。已获得的具有高效降解石油烃的细菌,主要是不动杆菌属、红球菌属、寡养单胞菌属和芽孢杆菌属等。本研究中微生物群落分析发现,油基钻屑生物处理过程中主要的细菌菌属不动杆菌属和红球菌属,已有研究表明这2个菌属在石油污染土壤修复可发挥重要的作用[24-25]。此外,包木太等[26]从石油污染的土壤中分离获得一株寡养单胞菌 (Stenotrophomonas sp.) ,其对原油去除率可以超过50%,且该菌株对C9~C38化合物均具有较好的降解效果,正构烷烃的去除率最高。本研究还得到寡养单胞菌属的细菌在不同样本中具有显著差异,其在酵母粉添加样本中含量最为丰富 (图4) ,且该样本中细菌群落对长链烷烃的降解效果较好 (图6和图7) ;而且本研究结果还表明C15H32、C16H34和C17H36对寡养单胞菌属的细菌具有明显的影响 (图8(a)) ,这说明寡养单胞菌属在长链烷烃的降解中可发挥重要的作用。芽孢杆菌属细菌是报道较多的可以高效降解油基钻屑和石油污染土壤中石油烃的微生物[7, 27-28]。本研究中添加酵母粉的样本中细菌群落的芽孢杆菌属较为丰富 (图1) ,且与添加其他氮源的组别中芽孢杆菌具有显著差异 (图4) ,其对6种长链烷烃的降解率也较高 (图6和图7) ,这说明本研究中芽孢杆菌属的细菌在油基钻屑清理中长链烷烃的降解中起着重要的作用。
此外,石油类化合物主要是饱和烃、芳香烃、非烃和沥青质。单菌株很难实现这4类化合物的同时降解,而利用混合菌株或者细菌群落可使各类菌株发挥出协同作用完成石油污染土壤的高效修复。YAN等[2]利用包括Pseudomonas、Acinetobacter、Alcaligenes、Agrobacterium、Comamonas在内的6种细菌的混合菌群降解清洗过的油基钻屑,发现混合菌群处理120 d后可将清洗过的油基钻屑中总油质量分数从(16 900±530) mg·kg−1降低至(5 470±370) mg·kg−1,降解率可高达67.3%。本研究采用从南海获得的海泥样本来源的细菌群落作为外源混合功能菌株,也可有效降低海上油基钻屑中长链烷烃的质量分数。因此,微生物群落替代单菌落可成为生物法处理油基钻屑的有效手段。
3) 添加不同氮源对细菌降解石油烃的影响。在污染土壤中额外补充氮源可增加土壤样本中营养物质的含量,为微生物生长代谢和行使其降解功能提供能量,从而提高微生物降解效率。其主要机制是添加的物质会增强微生物体内酶的活性,以提高微生物对营养物质的分配和转运速率,最终促使原油在微生物高效快速的分解[29]。此外,多数研究还指出,在有机层土壤中,低N添加显著增加了微生物碳利用效率 (45.12%) ,高N添加却显著减少了微生物碳利用效率 (27.84%) 。而在矿质层土壤中,低N添加处理中土壤微生物微生物碳利用效率没有发生变化,但在高N添加处理中微生物碳利用效率显著增加了133.18%[30]。这说明合适氮源的添加可通过促进微生物的生长和代谢促进微生物对石油烃类化合物的降解。本研究还发现,添加无机氮源和有机氮源可影响样本中细菌群落的结构、组成和功能,进一步不同程度的促进微生物群落对油基钻屑中长链烷烃的降解,特别是KH2PO4和酵母粉的添加,对本研究中探究的6种长链烷烃的降解效果显著。已有研究也证明无机氮源的添加有利于微生物降解柴油和石油烃等化合物,提高其在污染物降解中的应用。王鑫等[31]研究了外加氮源 (NH4Cl、NH4NO3和NaNO3) 对筛选获得3株石油降解菌 (T4、R4和D3) 降解柴油的影响,结果发现添加NaNO3可使R4菌株对柴油的降解效率从48%提高至77%,D3菌株对柴油的降解效率从44%提高至88%;而NH4NO3的添加使得T4菌株对柴油的降解率从42%提高至59%。WU等[32]在利用细菌处理含有污染土壤时,通过添加(NH4)2SO4和KH2PO4可使细菌降解石油烃类物质的效率增加到60%,效率提升76% (对照组的降解率为34%) 。而王艳杰等[33]通过在石油污染土壤中添加适量的NH4NO3和K2HPO4,可使样本中总石油烃类物质的去除率提高11.29%,其中芳香烃和饱和烃的降解效率有明显的提升。RAMADASS等[13]通过添加无机氮磷钾营养,在生物修复210 d后,总石油烃的去除效率为33%。XU等[14]在石油污染土壤中添加氮磷营养元素,8周的修复后石油降解率达到30%。然而,有机氮源,如酵母粉、蛋白胨等促进微生物对石油烃降解的相关研究较少,进一步的研究可细化有机氮源对微生物生长及其降解污染物功能的影响,为油基钻屑生物降解技术的现场应用提供参考。
-
1) 添加不同氮源的油基钻屑中细菌结构组成具有明显的差异,但其主要优势菌属均是不动杆菌属和红球菌属;且细菌群落结构随着处理时间的变化而变化。与未添加氮源的对照组相比,添加不同氮源的样品中微生物群落多样性和丰富度均降低,且添加不同氮源的样品中微生物群落丰富度无差异,添加无机氮源 (KNO3、(NH4)2SO4) 和有机氮源 (酵母粉、蛋白胨) 的样品中微生物群落多样性存在显著差异。添加不同氮源的样本中细菌群落在属水平上的差异菌属主要是是红球菌属 (0.01 ≤ P ≤ 0.05) 、新鞘氨醇杆菌属 (P ≤ 0.001) 、短波单胞菌属 (0.001≤ P ≤ 0.01) 、其他根瘤菌属 (0.01 ≤ P ≤ 0.05) 、柄杆菌属 (P ≤ 0.001) 、德沃斯氏菌属 (0.01 ≤ P ≤ 0.05) 、鞘氨醇单胞菌 (0.001≤ P ≤ 0.01) 、芽孢杆菌属 (P ≤ 0.001) 、寡养单胞菌属 (0.001≤ P ≤ 0.01) 和甲基杆菌属 (P ≤ 0.001) 。2) FAPROTAX功能预测结果说明,油基钻屑处理过程中以好氧菌和异养菌为主,而添加(NH4)2HPO4和酵母粉更有利于碳氢化合物降解,同时降解效果实验结果也证明添加(NH4)2HPO4和酵母粉可有效促进细菌群落对石油烃类化合物的降解,分析可能(NH4)2HPO4和酵母粉2种氮源在提供氮源的同时能提供其他营养元素,后续可以进一步分析磷、钾对石油烃降解细菌群落的促进作用。3) 通过环境因子分析,发现与油基钻屑中主要石油烃成分降解相关的主要菌株是不动杆菌属、红球菌属、芽孢杆菌属、寡养单胞菌属、鞘氨醇单胞菌和短波单胞菌属的细菌。
外加氮源对细菌群落及其降解油基钻屑中石油烃的影响
Effects of nitrogen sources addition on bacterial communities and their degradation of petroleum hydrocarbons in oil-based drilling cuttings
-
摘要: 为探究外加氮源种类对细菌群落及其降解油基钻屑中石油烃降解的影响,基于高通量测序技术和石油烃含量测定常规方法,研究了添加6种不同氮源后油基钻屑生物法处理过程细菌群落多样性、组成及主要基因功能的差异,并分析细菌群落变化与石油烃化合物降解之间的相关性。结果发现,不同氮源的添加使油基钻屑中细菌群落结构出现明显差异,但不动杆菌属 (Acinetobacter) 和红球菌属 (Rhodococcus) 仍是每个样本中的优势菌属,而不同氮源的添加降低了油基钻屑中细菌群落的多样性和丰富度;添加不同氮源的油基钻屑中细菌群落的主要差异 (P≤0.001) 菌属是新鞘氨醇杆菌属 (Novosphingobium) 、柄杆菌属 (Caulobacter) 、芽孢杆菌属 (Bacillus) 和甲基杆菌属 (Methylobacterium) ;磷酸氢二铵和酵母粉加入有效促进油基钻屑中主要石油烃成分的降解,多数降解率可达到60%以上;与油基钻屑中主要石油烃成分降解相关的主要菌株是不动杆菌属、红球菌属、芽孢杆菌属、寡养单胞菌属 (Stenotrophomonas) 、鞘氨醇单胞菌 (Sphingomonas) 和短波单胞菌属 (Brevundimonas) 的细菌。通过实验发现外加氮源有利于油基钻屑中主要石油烃主要成分的降解,以期为油基钻屑生物降解技术的现场应用提供参考。Abstract: To investigate the impact of different types of nitrogen sources addition on bacterial communities and their degradation of petroleum hydrocarbons in oil-based drilling cuttings.The differences in bacterial community diversity, composition, and functions during the biological treatment of oil-based drilling cuttings with six different nitrogen sources were studied, and the correlation between bacterial community changes and petroleum hydrocarbon degradation was analyzed by high-throughput sequencing technology and conventional methods for determining petroleum hydrocarbon.The results showed that nitrogen sources addition caused significant differences in the bacterial community structure in oil-based drilling cuttings, but Acinetobacter and Rhodococcus were still the dominant genera in each sample, and nitrogen sources addition reduced the diversity and richness of the bacterial community in oil-based drilling cuttings; The main differences (P ≤ 0.001) of bacterial communities in oil-based drilling cuttings were Novosphingobium, Caulobacter, Bacillus and Methylobacterium among the six nitrogen sources addition; The addition of (NH4)2HPO4 and yeast extract effectively promoted the degradation of the main petroleum hydrocarbons in oil-based drilling cuttings, with most degradation rates reaching over 60%; The main bacteria related to the degradation of major petroleum hydrocarbon components in oil-based drilling cuttings were Acinetobacter, Rhodococcus, Bacillus, Stenotrophomonas, Sphingomonas and Brevundimonas. It was found that nitrogen sources addition could be beneficial for the degradation of the main components of petroleum hydrocarbons in oil-based drilling cuttings, which could provide a research basis for the on-site application of oil-based drilling cuttings biodegradation technology.
-
-
表 1 油基钻屑中有机物主要成分及其质量分数
Table 1. Main components and mass proportion of organic compounds in oil-based drilling cuttings
直链烷烃 支链烷烃 环烷烃 环氧烷烃 卤代烃 烯烃 脂肪醇 脂肪酸 醚类 酮类 酯类 其他 51.29% 21.37% 7.54% 0.37% 4.07% 0.36% 3.13% 1.26% 0.53% 2.25% 7.38% 0.45% 表 2 本研究中样本的具体信息
Table 2. The detailed information of the samples in this study
编号 油基钻屑 海泥样本 添加氮源 备注 ZX0_1 + + − 油基钻屑 (0 d) ZX1_1 + + − 7 d (Control) ZX1_2 + + (NH4)2HPO4 7 d ZX1_3 + + NH4Cl 7 d ZX1_4 + + KNO3 7 d ZX1_5 + + (NH4)2SO4 7 d ZX1_6 + + Yeast extract 7 d ZX1_7 + + Peptone 7 d ZX2_1 + + − 14 d (Control) ZX2_2 + + (NH4)2HPO4 14 d ZX2_3 + + NH4Cl 14 d ZX2_4 + + KNO3 14 d ZX2_5 + + (NH4)2SO4 14 d ZX2_6 + + Yeast extract 14 d ZX2_7 + + Peptone 14 d ZX3_1 + + − 21 d (Control) ZX3_2 + + (NH4)2HPO4 21 d ZX3_3 + + NH4Cl 21 d ZX3_4 + + KNO3 21 d ZX3_5 + + (NH4)2SO4 21 d ZX3_6 + + Yeast extract 21 d ZX3_7 + + Peptone 21 d ZX4_1 + + − 28 d (Control) ZX4_2 + + (NH4)2HPO4 28 d (N1) ZX4_3 + + NH4Cl 28 d (N2) ZX4_4 + + KNO3 28 d (N3) ZX4_5 + + (NH4)2SO4 28 d (N4) ZX4_6 + + Yeast extract 28 d (N5) ZX4_7 + + Peptone 28 d (N6) 表 3 油基钻屑处理过程中主要细菌群落在门水平和属水平的相对丰度
Table 3. The relative abundance of main bacterial communities at genus level and phyla level during the bioprocessing of oil-based drilling cuttings
添加氮源 样品
编号变形菌门
(Proteobacteria)放线菌门
(Actinobacteriota)其他
(Others)不动杆菌属
(Acinetobacter)红球菌属
(Rhodococcus)新鞘氨醇杆菌属
(Novosphingobium)短波单胞菌属
(Brevundimonas)其他根瘤菌属
(Allorhizobium)柄杆菌属
(Caulobacter)德沃斯氏菌属
(Devosia)芽孢杆菌属
(Bacillus)其他
(Others)Raw ZX0_1 16.80% 81.28% 1.91% 0.17% 80.71% 0.02% 0.04% 0.01% 0.01% 0.01% 0.27% 18.76% Control ZX1_1 75.91% 23.81% 0.28% 53.41% 23.72% 6.84% 0.03% 1.57% 1.33% 0.00% 0.11% 12.97% ZX2_1 31.77% 12.45% 55.78% 11.03% 11.16% 3.06% 0.11% 4.34% 0.45% 0.06% 0.12% 69.66% ZX3_1 82.77% 16.88% 0.35% 34.33% 16.81% 19.94% 0.16% 13.53% 2.19% 0.00% 0.12% 12.91% ZX4_1 87.91% 9.50% 2.59% 28.59% 9.44% 19.19% 0.42% 20.49% 3.24% 0.00% 0.08% 18.53% (NH4)2HPO4 ZX1_2 94.14% 5.68% 0.18% 83.60% 5.65% 0.03% 1.45% 2.80% 0.03% 0.00% 0.00% 6.44% ZX2_2 28.12% 71.87% 0.01% 26.72% 71.83% 0.00% 0.35% 0.42% 0.01% 0.00% 0.00% 0.66% ZX3_2 10.66% 89.33% 0.01% 9.00% 89.13% 0.02% 0.64% 0.48% 0.01% 0.00% 0.00% 0.73% ZX4_2 35.73% 63.02% 1.26% 16.55% 61.81% 0.48% 13.42% 2.74% 0.02% 0.05% 0.00% 4.94% NH4Cl ZX1_3 81.12% 18.69% 0.19% 59.93% 18.61% 7.21% 9.24% 0.13% 1.98% 0.00% 0.01% 2.88% ZX2_3 73.08% 26.64% 0.27% 37.08% 26.54% 10.20% 11.22% 2.76% 7.22% 0.02% 0.19% 4.77% ZX3_3 84.49% 15.34% 0.17% 34.09% 15.22% 12.61% 16.79% 3.85% 11.91% 0.28% 0.02% 5.23% ZX4_3 94.07% 5.45% 0.48% 22.84% 5.37% 20.49% 27.19% 5.02% 9.30% 2.19% 0.01% 7.59% KNO3 ZX1_4 88.46% 11.42% 0.12% 44.51% 10.97% 23.94% 0.78% 2.83% 0.32% 9.18% 0.02% 7.45% ZX2_4 78.22% 21.37% 0.41% 19.65% 20.27% 27.10% 1.58% 10.21% 0.88% 11.15% 0.16% 8.99% ZX3_4 90.35% 9.49% 0.15% 18.61% 7.83% 29.53% 1.03% 11.01% 1.05% 20.89% 0.03% 10.02% ZX4_4 88.53% 9.21% 2.26% 5.93% 3.95% 27.96% 2.68% 19.43% 1.64% 14.84% 0.01% 23.56% (NH4)2SO4 ZX1_5 81.94% 17.92% 0.14% 50.74% 17.76% 16.21% 3.88% 0.01% 5.71% 0.00% 0.01% 5.68% ZX2_5 79.00% 20.87% 0.12% 31.26% 20.82% 14.38% 6.37% 2.00% 14.18% 0.00% 0.02% 10.97% ZX3_5 89.70% 10.28% 0.02% 25.90% 10.26% 7.96% 18.15% 3.76% 24.09% 0.00% 0.00% 9.87% ZX4_5 82.35% 14.06% 3.59% 17.38% 14.04% 9.33% 22.39% 7.22% 13.98% 0.06% 0.00% 15.60% 酵母粉 (Yeast extract) ZX1_6 77.51% 4.77% 17.73% 67.53% 1.46% 0.00% 0.00% 1.05% 0.00% 0.00% 17.18% 12.78% ZX2_6 63.43% 32.48% 4.09% 59.30% 31.74% 0.00% 0.75% 0.32% 0.00% 0.00% 3.98% 3.91% ZX3_6 28.78% 69.28% 1.94% 22.96% 66.65% 0.00% 3.45% 0.49% 0.00% 0.01% 1.76% 4.67% ZX4_6 51.44% 42.63% 5.93% 41.10% 41.83% 0.01% 4.69% 0.76% 0.00% 0.09% 3.14% 8.39% 蛋白胨 (Peptone) ZX1_7 94.09% 0.40% 5.50% 78.56% 0.22% 0.00% 12.90% 0.75% 0.00% 0.00% 2.03% 5.54% ZX2_7 91.44% 3.00% 5.56% 62.87% 2.60% 0.00% 24.31% 1.56% 0.00% 0.00% 2.18% 6.46% ZX3_7 94.25% 0.47% 5.27% 71.79% 0.27% 0.00% 18.66% 1.64% 0.00% 0.00% 1.87% 5.77% ZX4_7 96.31% 0.23% 3.45% 70.35% 0.07% 0.00% 19.39% 3.10% 0.00% 0.04% 1.14% 5.91% 表 4 本研究不同样品中细菌群落的Alpha多样性指数
Table 4. The alpha diversity index of bacterial community at the samples in this study
添加氮源 样品编号 sobs指数 shannon指数 覆盖率 coverage Raw ZX0_1 514 0.866 4 0.999 8 Control ZX1_1 246 1.763 6 0.999 5 ZX2_1 836 3.719 3 0.999 3 ZX3_1 247 2.310 1 0.999 3 ZX4_1 264 2.564 8 0.999 5 (NH4)2HPO4 ZX1_2 98 0.987 3 0.999 7 ZX2_2 72 0.720 4 0.999 8 ZX3_2 76 0.453 3 0.999 8 ZX4_2 127 1.375 6 0.999 7 NH4Cl ZX1_3 213 1.489 0 0.999 4 ZX2_3 194 1.963 7 0.999 6 ZX3_3 207 2.174 5 0.999 5 ZX4_3 265 2.356 9 0.999 3 KNO3 ZX1_4 198 2.033 6 0.999 5 ZX2_4 256 2.292 3 0.999 5 ZX3_4 211 2.267 3 0.999 6 ZX4_4 177 2.437 7 0.999 7 (NH4)2SO4 ZX1_5 216 1.791 0 0.999 5 ZX2_5 248 2.244 1 0.999 6 ZX3_5 176 2.330 0 0.999 7 ZX4_5 199 2.623 5 0.999 5 酵母粉 (Yeast extract) ZX1_6 111 1.607 2 0.999 8 ZX2_6 107 1.182 4 0.999 7 ZX3_6 119 1.155 0 0.999 7 ZX4_6 140 1.562 0 0.999 7 蛋白胨 (Peptone) ZX1_7 80 1.032 1 0.999 7 ZX2_7 129 1.370 2 0.999 7 ZX3_7 108 1.186 9 0.999 8 ZX4_7 118 1.249 6 0.999 8 -
[1] 左京杰, 张鑫, 杨勇. 含油钻屑处理技术现状及发展趋势[J]. 油气田环境保护, 2019, 29(6): 11-15. doi: 10.3969/j.issn.1005-3158.2019.06.003 [2] YAN P, LU M, GUAN Y, et al. Remediation of oil-based drill cuttings through a biosurfactant-based washing followed by a biodegradation treatment[J]. Bioresource Technology, 2011, 102(22): 10252-10259. doi: 10.1016/j.biortech.2011.08.074 [3] VARJANI S J, UPASANI V N. A new look on factors affecting microbial degradation of petroleum hydrocarbon pollutants[J]. International Biodeterioration & Biodegradation, 2017, 120: 71-83. [4] 郁丽萍, 吕静, 章诗辞, 等. 页岩气油基钻屑降解菌群的构建及其降解特性[J]. 环境污染与防治, 2018, 40(10): 1101-1105. [5] TURNER KP. Bioremediation of drill cuttings from oil based muds[D]. Nottingham: University of Nottingham, 2002. [6] 单海霞, 何焕杰, 刘晓宇, 等. 油基钻屑的生物处理[J]. 江南大学学报(自然科学版), 2013, 12(4): 470-474. [7] 陈政阳, 刘国, 唐彬彬, 等. 油基泥浆钻井岩屑中高效石油降解菌的筛选及其降解特性[J]. 环境工程, 2018, 36(2): 48-53. [8] REZAEI S M, SHAVANDI M, DASTGHEIB S M M, et al. Bioremediation of oil-based drill cuttings by a halophilic consortium isolated from oil-contaminated saline soil[J]. 3 Biotech, 2018, 8: 229. [9] OKPARANMA R, AYOTAMUNO J, ARAKA P. Bioremediation of hydrocarbon contaminated-oil field drill-cuttings with bacterial isolates[J]. African Journal of Environmental Science and Technology, 2009, 3(5): 131-140. [10] 蔡静, 杨国兴, 张胜利, 等. 四川工区秋冬季微生物降解油基钻屑可行性研究[C]. 2015年度钻井液完井液学组工作会议暨技术交流研讨会论文集, 广东湛江, 2015, 681-684. [11] KALANTARY R R, MOHSENI-BANDPI A, ESRAFILI A, et al. Effectiveness of biostimulation through nutrient content on the bioremediation of phenanthrene contaminated soil[J]. Journal of Environmental Health Science & Engineering, 2014, 12: 143. [12] 杨茜, 吴蔓莉, 聂麦茜, 等. 石油污染土壤的生物修复技术及微生物生态效应[J]. 环境科学, 2015, 36(5): 1856-1863. [13] RAMADASS K, MEGHARARJ M, VENKATESWARLU K, et al. Bioavailability of weathered hydrocarbons in engine oil-contaminated soil: Impact of bioaugmentation mediated by Pseudomonas spp. on bioremediation[J]. The Science of the total environment, 2018, 636: 968-974. [14] XU J, ZHANG J, HUANG T, et al. Comparative bioremediation of oil contaminated soil by natural attenuation, biostimulation and bioaugmentation[J]. Advanced Materials Research, 2013, 777: 258-262. [15] 白超峰, 吴洪特, 岳前升, 等. 废弃油基钻井液钻屑含油量室内测定方法探讨[J]. 石油天然气学报, 2013, 35(12): 4. [16] 陈则良, 陈忠, 陈乔, 等. 典型页岩气油基钻屑的组成分析及危害评价[J]. 环境工程, 2017, 35(8): 125-129. [17] WANG F, ZHAO H W, XIANG H Y, et al. Species diversity and functional prediction of surface bacterial communities on aging flue-cured tobaccos[J]. Current Microbiology, 2018, 75: 1306-1315. doi: 10.1007/s00284-018-1525-x [18] REN Y, YU G, SHI C P, et al. Majorbio Cloud: A one-stop, comprehensive bioinformatic platform for multiomics analyses[J]. iMeta, 2022, 1: e12. doi: 10.1002/imt2.12 [19] 徐金兰, 田桂永, 师启航. Fenton预氧化促进土壤微生物均衡降解烷烃[J]. 环境工程, 2023, 41(2): 131-139. [20] 程玉, 王继华. 污染土壤中植物根际细菌群落功能差异研究[J]. 环境科学与技术, 2022, 45(5): 84-91. [21] 孙思路. 施用不同氮素对土壤中抗生素抗性基因丰度和细菌群落的影响[D]. 南京: 南京农业大学, 2020. [22] LIU W X, JIANG L, YANG S, et al. Critical transition of soil bacterial diversity and composition triggered by nitrogen enrichment[J]. Ecology, 2020, 101: e03053. doi: 10.1002/ecy.3053 [23] 李涛. 南海沉积物微生物多样性及群落结构研究[D]. 上海: 同济大学, 2008. [24] 张光军, 方萍. 红球菌在石油烃类物质降解中的作用[J]. 环境科学与技术, 2013, 36(2): 7. [25] 刘玉华, 王慧, 胡晓珂. 不动杆菌属(Acinetobacter)细菌降解石油烃的研究进展[J]. 微生物学通报, 2016, 43(7):1579-1589. [26] 包木太, 王丽娜, 李一鸣, 等. 一株石油烃降解菌D2-1解烃性能研究[J]. 深圳大学学报理工版, 2012, 29(6): 515-520. [27] 龚汉意, 邓春萍, 柯强, 等. 一株耐盐石油烃降解菌的分离、鉴定及其降解特性研究[J]. 山东化工, 2021, 50(4): 39-42. doi: 10.3969/j.issn.1008-021X.2021.07.015 [28] 李慧恩, 陈冠雄, 张颖, 等. 高效石油烃降解菌的分离鉴定及降解特性[J]. 哈尔滨工业大学学报, 2007, 39(10): 1664-1669. [29] DU Z, CHEN Y, LI X. Quantitative proteomic analyses of the microbial degradation of estrone under various background nitrogen and carbon conditions[J]. Water Research, 2017, 123: 361-368. [30] LI J, SANG C P, YANG J Y, et al. Stoichiometric imbalance and microbial community regulate microbial elements use efficiencies under nitrogen addition[J]. Soil Biology and Biochemistry, 2021, 156: 108207. doi: 10.1016/j.soilbio.2021.108207 [31] 王鑫, 王学江, 卜云洁, 等. 外加氮源强化石油降解菌降解性能[J]. 同济大学学报(自然科学版), 2014, 42(6): 924-929. [32] WU M L, DICK W A, LI W H, et al. Bioaugmentation and biostimulation of hydrocarbon degradation and the microbial community in a petroleum-contaminated soil[J]. International Biodeterioration & Biodegradation, 2016, 107: 158-164. [33] 王艳杰, 李法云, 荣湘民, 等. 生物质材料与营养物配施对石油污染土壤的修复[J]. 农业环境科学学报, 2018, 37(2): 232-238. doi: 10.11654/jaes.2017-1058 -



 下载:
下载: