-
氯霉素(chloramphenicol,CAP)是一种广谱抗生素,已被广泛用于人类医学、水产和动物养殖等[1]。由于药物滥用,CAP在水环境中被广泛检测到[2]。CAP对人类的血液系统、消化系统均具有毒性和副作用,并具有潜在的致癌性和基因毒性[3]。此外,大量使用抗生素会增加抗生素耐药细菌(antibiotic-resistant bacteria,ARB)和抗生素抗性基因(antibiotic resistance genes,ARG)发生和传播的风险,这已成为严重的全球公共卫生问题。高浓度的抗生素对环境中的微生物产生巨大的选择压力,并通过水平基因转移加速ARG的大量增殖[4]。氯霉素耐药的主要机制被认为是核糖体靶标的修饰[5],而其抗菌活性主要与CAP分子中的丙二醇结构有关[6]。氯霉素乙酰转移酶(chloramphenicol acetyltransferase,CAT)使丙二醇上的羟基与来自乙酰辅酶A的乙酰基发生氯霉素乙酰化[7]。在氯霉素耐药性产生的过程中菌体内带有耐药遗传基因的质粒通过介导产生氯霉素乙酰转换酶,使氯霉素中丙二醇基团的3-羟位乙酰化,从而使氯霉素不能与细菌核糖体的50S亚基结合而失去活性[8]。
近年来,厌氧膜生物反应器(anaerobic membrane bioreactor,AnMBR)已经成为处理抗生素废水的具有前景的技术。AnMBR将厌氧生物处理技术和膜技术相结合,使得污泥停留时间和水力停留时间相分离,具有更好的出水水质和较低的污泥产量等优势[9-10]。AnMBR较长的污泥停留时间更有利于抗生素的去除,完全的生物质截留有助于富集降解难降解有机物质的细菌[11],在高效处理制药废水方面具有潜力[12]。之前有研究表明,氯霉素可以通过厌氧生物作用转化为不具有抗菌活性的副产物[13]。但CAP降解路径的研究并不完全,需要进一步探究CAP的降解中间产物,证明CAP在AnMBR中降解过程的有效性和可行性。
然而,高浓度的氯霉素会抑制AnMBR中水解酸化菌和产甲烷古菌的活性[14]。导电材料已被广泛用于强化直接种间电子转移过程(direct interspecies electron transfer, DIET),这有利于缓解挥发性脂肪酸的累积且可促进产甲烷过程。碳基导电材料包括活性炭、生物炭、石墨烯等,其具有大的比表面积可使微生物在其表面定殖,并且通过表面的含氧官能团或者碳基材料的高电导率强化DIET过程[15]。与生物炭相比,纳米洋葱碳(carbon nano-onions,CNOs)热重损失低且具有极少的含氧官能团,因此,不足以通过含氧官能团传递电子[16]。CNOs作为一种新型的零维碳纳米材料,具有高比表面积、高电导率和更好的生物相容性[17]。在之前的研究中发现将CNOs投加到AnMBR中,CNOs可以作为导电鞭毛促进微生物之间的直接种间电子转移[16]。然而,CNOs强化厌氧消化去除CAP的机理还需要进一步研究。此外,CNOs对于抗生素抗性基因的影响仍然是未知的。
基于以上研究结果,本研究将CNOs应用于AnMBR处理氯霉素废水,分析了AnMBR中氯霉素的降解路径和抗生素抗性基因的分布,并采用续批实验解析CNOs强化厌氧消化去除CAP的相关机理,为AnMBR在氯霉素废水处理的应用提供新的见解。
-
将纳米金刚石在氮气氛围中逐步加热到1 700 ℃制备得CNOs。采用透射电子显微镜(transmission electron microscope, TEM)(JEOL JEM 2100F,日本电子株式会社)观察CNOs形貌;采用X射线衍射(X-ray diffraction, XRD)(D8 Advance,德国布鲁克公司)确定CNOs的晶格特征;采用傅里叶红外光谱仪((Nicolet5700,美国Thermo公司)表征CNOs的官能团。
本实验使用模拟的制药废水,由于缺乏用于CAP厌氧降解的电子供体,蔗糖被用作碳源来促进CAP的厌氧共消化,具体组成与质量浓度按照之前研究所述[15]。
-
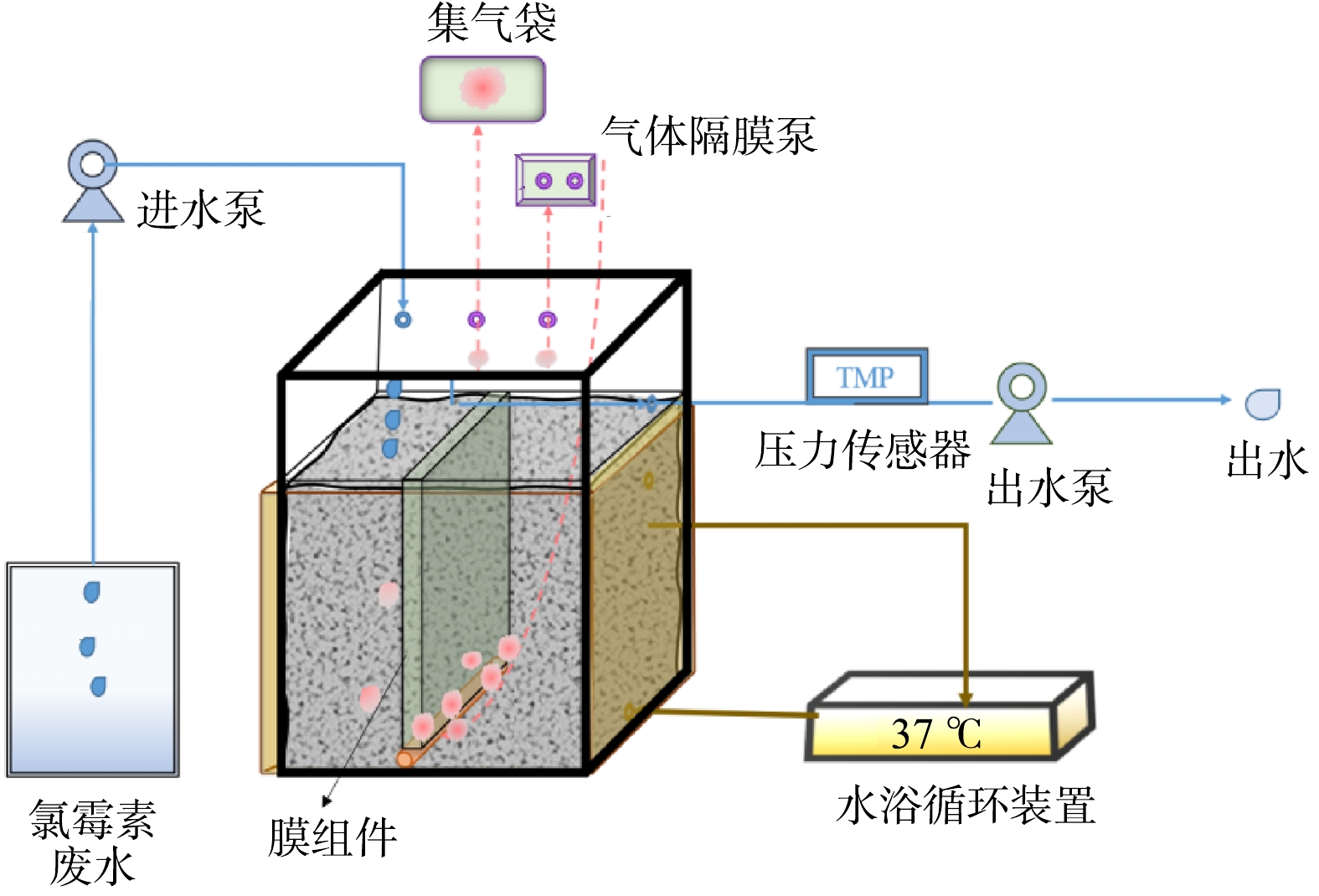
本研究中使用的反应器如图1所示,该反应器的工作容积为20 L,配备了膜孔径为0.1 µm、过滤面积为0.102 9 m2的微滤膜。反应器的HRT为48 h,过滤通量为4.04 L·(m2·h)−1。
-
1) 续批实验。分3组进行续批实验,实验条件见表1。A组为空白组,B组为对照组,C组为实验组。所有组的工作体积为150 mL(20 mL接种物和130 mL底物)。接种了污泥和底物之后,血清瓶内曝氮气30 min以保证厌氧条件。所有续批实验在38 ℃的恒温振荡培养箱中进行,转速为120 r·min−1,定时取样检测其CAP质量浓度、化学需氧量(chemical oxygen demand,COD)和甲烷体积,所有实验设置3个平行样。
2) AnMBR实验。AnMBR用于处理模拟CAP废水,第1阶段用于驯化污泥,以适应10 mg·(L·d)−1的模拟CAP废水,有机负荷(以COD计)为1.12 g·(L·d)−1;第2阶段的CAP负荷提高至30 mg·(L·d)−1,有机负荷降至0.56 g·(L·d)−1;第3阶段的CAP负荷维持在30 mg·(L·d)−1,有机负荷升至1.12 g·(L·d)−1;与第3阶段相比,第4阶段将CAP负荷提升至50 mg·(L·d)−1,有机负荷维持在1.12 g·(L·d)−1,并添加100 mg·L−1的CNOs,用于探索CNOs在高负荷下的CAP和有机负荷对CAP废水处理的影响,其厌氧消化性能和膜过滤性能较为稳定[15]。本研究分析了AnMBR处理CAP的中间产物鉴定与抗生素抗性基因的分布。
-
1)氯霉素及其副产物的鉴定。通过高效液相色谱-质谱分析和鉴定CAP以及副产物。高效液相色谱系统配备了C18色谱柱(4.6 mm×250 mm×5 μm)和波长为280 nm的紫外检测器。流动相是甲醇和水的混合物(50:50,v:v),流量为0.8 mL·min−1,温度为35 ℃。质谱的流动相为45%甲酸水溶液(0.1%)和55%甲醇。流速为0.3 mL·min−1。质谱质荷比扫描范围为50~500。
2)抗生素抗性基因的测定及常规分析。对外排泵基因(cmlA、floR和tetC)、第一类整合子的整合酶(intI1)、intI1相关的sul1基因进行了定量PCR,使用的引物列于表2。不同阶段和初始污泥之间ARGs的显著差异(P)通过t检验进行分析,P<0.05为统计学意义。用SPSS24.0评估ARGs和CAP之间的相关性[4]。气体中的甲烷体积按照描述的方法测定[15]。
3) PICRUSt2预测。PICRUSt2是针对16S rDNA扩增子测序结果进行功能预测。根据京都基因和基因组百科全书(KEGG)数据库的信息,可以获得代谢通路信息,并根据分类单元丰度计算出各功能类别的丰度。在Majorbio云平台(www. majorbio.com)上进行分析。
-
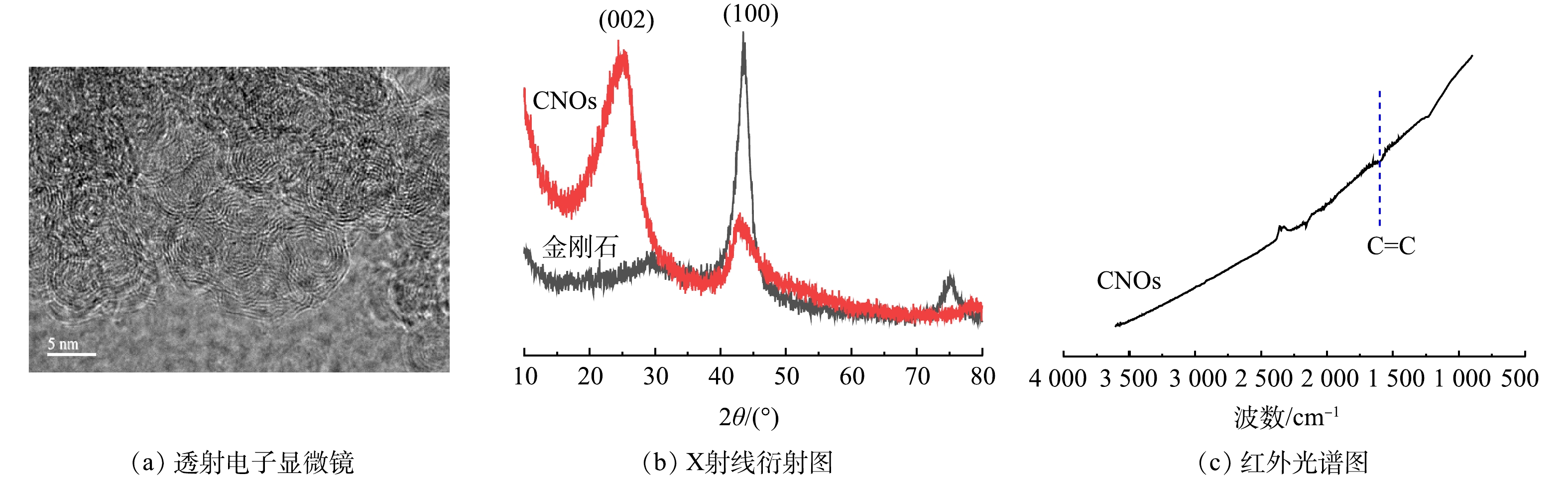
图2(a)为CNOs的TEM图。由图2(a)中可以看出,所制备的CNOs为纳米尺寸的洋葱碳。金刚石和CNOs的XRD图谱如图2(b)所示。可见,在CNOs的图谱中出现了2θ为25.3°和43.4°的2个石墨峰,分别对应002晶面和100晶面,表明CNOs的成功制备[18]。图2(c)为CNOs的红外谱图。可见,1 583 cm−1处的特征峰归因于共轭碳基团的C=C伸缩振动,红外谱图以及之前的研究中热重基本没有损失表明CNOs具有较少的含氧官能团[18]。
-
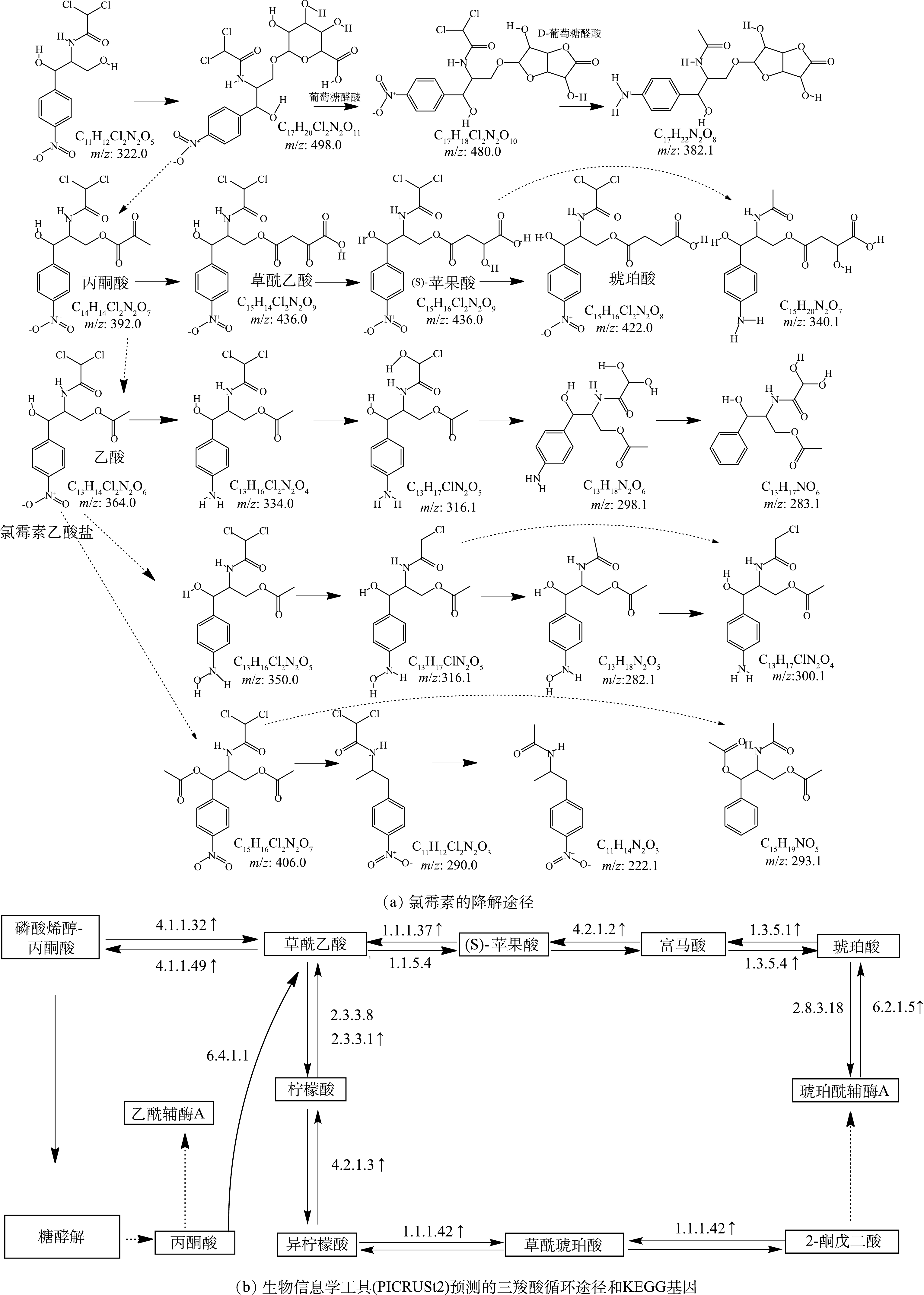
有研究表明,50 mg·L−1 CAP可以完全抑制厌氧消化,但CNOs强化的AnMBR取得了稳定且优异的CAP去除效率[16,19],这表明CNOs强化的AnMBR中CAP的抑菌性能减缓,因此,需要进一步探究CAP的厌氧降解产物。如图3显示,已知的CAP降解产物包括硝基还原产物α-N-二氯乙酰基-P-氨基苯基塞里诺(AMCl2,m/z=293),随后的脱氯产物N-(1-(4-氨基苯基)-1、3-二羟基丙-2-基)-氯乙酰胺(AMCl,m/z=257),最后被脱氯降解为N-(1-(4-氨基苯基)-1,3-二羟基丙-2-基)乙酰胺(AM,m/z=224)。这些已知的CAP降解产物没有抗菌活性[13]。而在本研究中,质谱结果(图3)表明这些中间产物在总丰度中不占优势,还存在其他的中间产物。
有研究[19]表明,CAP可以转化为氯霉素乙酸盐从污水处理厂中排出。本研究中的质谱检测结果表明氯霉素乙酸盐(m/z=362.1923)显示出较高丰度(图3),氯霉素乙酸盐是CAT作为抗生素耐药酶介导的产物,氯霉素中的丙二醇结构与乙酸发生酯化反应可降低CAP的毒性[20]。此外,由图4(a)所示的降解路径中,氯霉素乙酸盐和氯霉素1,3-二乙酸盐还可以进一步发生脱氯和硝基还原反应。
HPLC/MS中还检测到高丰度的m/z值为499.2、481.3、437.2、438.2和393.2的物质。图4(a)显示了可能的降解路径,中间产物为氯霉素葡萄糖醛酸酯(m/z=499.2)、氯霉素葡萄糖醛酸内酯(m/z=480.0)、氯霉素丙酮酸盐(m/z=392.0)、草酸氯霉素(m/z=436.0)、琥珀酸氯霉素(m/z =423.2)和苹果酸氯霉素(m/z=438.0)。葡萄糖苷、丙酮酸、琥珀酸和苹果酸,包括乙酰辅酶A(介导CAT),都参与了以葡萄糖为底物的三羧酸(tricarboxylic acid cycle, TCA)循环。由图4可见,CAP上的丙二醇基团和参与TCA的物质发生了酯化反应,与CAT的作用机理相一致,酯化反应使得丙二醇基团产生的抗菌活性消失[21-22]。这一途径可能与以下2点有关:首先,含羧酸的TCA产物会代谢成酰基辅酶A加合物,这些加合物可能与氯霉素具有更高的反应性,从而产生抗药性[23];其次,脂肪酶也能与氯霉素发生互补酯化和水解反应,合成氯霉素衍生物[24]。在动物体内也检测葡萄糖醛酸以及葡萄糖醛酸内酯通常用于将有毒物质结合成无毒的结合剂,这有利于降低毒性[25-26]。MA等[27]发现了一个新的氯霉素降解途径,其中包含一个相对分子质量为335.0的未知产物,这个未知产物可能推测为氯霉素草酰乙酸的硝基还原和脱氯产物。
图4(b)反映了当CAP的质量浓度从第1阶段的10 mg·L−1增加到第3阶段的30 mg·L−1时,TCA循环酶的变化。CAP的增加导致参与TCA循环的大多数酶的丰度上调,这刺激了低毒性的氯霉素降解产物的生成。与第3阶段相比,添加CNOs后,苹果酸脱氢酶、琥珀酰转移酶、丙酮酸合成酶、柠檬酸合成酶、磷酸烯醇丙酮酸羧激酶、富马酸水合酶、丙酮酸羧化酶相关酶的活性得到提高(表3),所有这些酶都参与了TCA循环过程。因此,CNOs通过加强TCA过程可降低CAP的生物毒性。
-
微生物可以通过流出泵蛋白将进入细胞的抗生素主动泵出细胞而产生抗生素耐药性[28]。因此,本研究调查了编码流出泵的典型ARG,包括cmlA、floR、sul1和tetC[4]。不同阶段ARG的相对丰度如图5(a)所示。使用CNOs后,I类整合酶基因(class 1 integron integrase encoding gene, intI1)的相对丰度明显高于上一阶段,intI1是抗性基因发生水平转移的指标基因[29]。细菌通过水平基因转移获得ARG,这是微生物获得抗性的主要途径,表明CNOs增强了ARG的水平基因转移。
为了评估水平基因转移和CAP质量浓度对ARGs改变的影响,在ARGs和intI1、CAP进行了相关分析(图5(b))。cmlA的相对丰度随着CAP质量浓度的增加而明显增加,并与intI1呈正相关,表明cmlA通过水平基因转移。值得注意的是,sul1的相对丰度远远高于其他ARGs,可能和sul1在最初的污泥中的相对丰度较高有关。这可能与sul1的底物特异性较弱有关,其广泛存在于污水处理厂中,可以运输各种抗生素[29]。
此外,在第3阶段,膜表面泥饼层的intl1比污泥混合物中的高。之前的研究表明AnMBR可以有效去除ARG(至少1.9~3.9个对数单位),去除率与膜污染程度成正相关[29]。适当的膜污染通过孔堵塞来减小膜的有效孔径,从而截留较小尺寸的ARG。此外,膜污染层的主要成分胞外聚合物与AnMBR中ARG的去除表现出显著的相关性[30]。
-
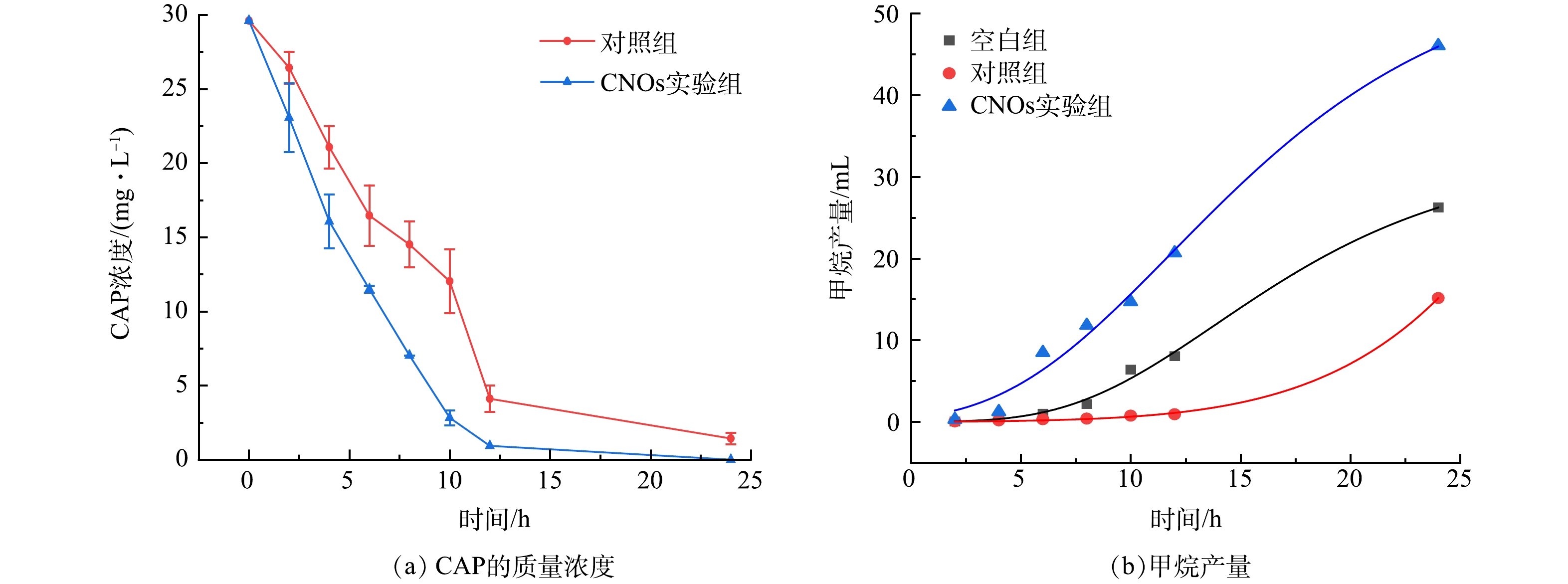
之前的研究表明,CNOs缓解了AnMBR中VFA的累积和增加了甲烷菌的丰度[16]。为了进一步探究CNOs对于厌氧消化的作用,在3组厌氧生物体系处理CAP废水过程中探究了CNOs对于强化厌氧消化各过程的作用。由图6(a)可知,10 h后,添加CNOs的实验组中,30 mg·L−1的氯霉素在对照组降解到2.8 mg·L−1,相应的CAP去除率为90.4%,反应10 h后,在对照组降解到12.0 mg·L−1,相应的CAP去除率为59.4%。24 h后实验组的CAP完全去除,而对照组的CAP去除率仅为95.2%。添加CNOs后,CAP的降解速率得到提高,这可能归因于CNOs增强了TCA过程,从而促进了CAP的生物降解。24 h内空白组、对照组和实验组甲烷产量分别为26.3、15.2和46.1 mL,相应的产甲烷滞后时间分别为7.3、41.7 h和4.3 h,对照组产甲烷滞后时间远高于空白组(表4),表明氯霉素的添加抑制了甲烷的生成。而添加CNOs后,通过空白组和对照组实验证实了CNOs在氯霉素抑制的情况下产甲烷的强化作用,这可以归因于高电导率高生物相容性的CNOs可以促进直接种间电子转移[16]。
与其他技术相比,CNOs强化的AnMBR技术具有以下优势。首先,在AnMBR中添加了100 mg·L−1的CNOs,对50 mg·L−1的CAP平均去除率可以高达98.6%[16],而生物电芬顿工艺处理50 mg·L−1 CAP的去除率仅为36.38%[31]。其次,改性Ti4O7阳极可以高效电氧化降解废水中的氯霉素[32]。相较而言,CNOs的制备相对简单,并且可以长期强化AnMBR处理氯霉素废水的效果且无需后续额外的成本消耗。此外,相较于吸附法的相转移[33],CNOs可以通过强化TCA过程有助于CAP的生物毒性的降低。
另一方面,从废刀具中回收的金刚石经过高温退火可制备得到CNOs[34]。厌氧消化的产物——甲烷可作为碳源通过催化裂解制备CNOs,有效减少温室气体的排放[35]。因此,通过绿色环保工艺制备CNOs并将其用于厌氧生物技术具有显著的经济效益和环境效益。
-
1)通过质谱分析解析了AnMBR中CAP可能的降解途径,发现除了已知的脱氯和硝基还原过程,CAP和参与TCA循环的物质之间的酯化反应,这一定程度上降低CAP的生物毒性。
2) CNOs显著提高了AnMBR中intI1的相对丰度,增强ARG的水平基因转移。此外,AnMBR的膜组件可以有效截留ARG。
3)续批实验证实了CNOs在厌氧消化中的强化效果:CNOs的添加显著提高了氯霉素的去除效率,CAP在10 h内去除率高达90.4%,24 h后对照组的CAP去除率为95.2%。此外,CNOs还增强了甲烷产量,产甲烷滞后时间被大大缩短。
纳米洋葱碳强化厌氧膜生物反应器处理氯霉素废水
Carbon nano-onions enhancing anaerobic membrane bioreactor for chloramphenicol wastewater treatment
-
摘要: 针对氯霉素难以生物降解的问题,本研究将纳米洋葱碳(carbon nano-onions,CNOs)作为导电材料投加到厌氧膜生物反应器(anaerobic membrane bioreactor,AnMBR)去除废水中的氯霉素(chloramphenicol,CAP),探讨了CAP的厌氧降解途径和抗生素抗性基因(ARG)分布的情况,解析了CNOs对氯霉素去除的强化作用。质谱检测结果表明CAP和参与三羧酸循环的物质之间发生了酯化反应,这些降解产物的毒性低于CAP。此外,CNOs的添加促进了部分三羧酸循环酶的上调,进一步降低了CAP的生物毒性。研究还发现CNOs可以增强ARG的水平基因转移,其中intI1基因的相对丰度显著增加。另一方面,AnMBR的膜组件有效截留了ARG。采用序批实验进一步研究CNOs对氯霉素去除的强化作用,结果表明,添加CNOs的实验组中,反应10 h时 CAP去除率为90.4%,远高于未添加CNOs的对照组(59.4%)。此外,添加CNOs的厌氧生物体系24 h内甲烷产量为46.1 mL,远高于对照组,且产甲烷滞后时间更短。以上结果表明CNOs强化了厌氧生物体系对氯霉素废水的去除效果,降低了CAP的生物毒性,可为AnMBR处理抗生素废水提供参考。Abstract: To address the problem of difficult biodegradation of chloramphenicol (CAP), carbon nano-onions (CNOs) were used as a conductive material for the removal of CAP from wastewater by an anaerobic membrane bioreactor (AnMBR). The anaerobic degradation pathways of CAP and the distribution of antibiotic resistance genes (ARGs) were investigated, and the enhanced effect of CNOs on CAP removal was analyzed. Mass spectrometry data indicated that the esterification reactions occurred between CAP and substances involved in the tricarboxylic acid cycle, and these degradation products were less toxic than CAP. Moreover, the addition of CNOs promoted the up-regulation of some tricarboxylic acid cycle enzymes, which further reduced the biotoxicity of CAP. It was also found that CNOs enhanced horizontal gene transfer of ARGs, of which the relative abundance of intI1 gene significantly increased. On the other hand, the membrane module of AnMBR effectively sequestered ARGs. Sequential batch experiments were used to further investigate the enhanced effect of CNOs on CAP removal, and the results showed that the CAP removal rate in the experimental group with the addition of CNOs was 90.4% (10 h), which was much higher than that in the control group without CNOs (59.4%). Besides, the methane production of the anaerobic biological system with the addition of CNOs was 46.1 mL in 24 h, which was much higher than that of the control group, and the lag time for methane production was shorter. These results indicated that CNOs strengthened the removal effect of anaerobic biotic system on CAP wastewater and reduced the biotoxicity of CAP, which provided a certain theoretical basis for the treatment of antibiotic wastewater by AnMBR.
-
-
表 1 续批实验的运行条件
Table 1. Operating conditions for batch experiments
组别 COD/
(mg·L−1)CAP质量浓度/
(mg·L−1)CNOs质量浓度/
(mg·L−1)A组 1 120 — — B组 1 120 30 — C组 1 120 30 100 表 2 本研究中检测的目标基因的PCR引物
Table 2. PCR primers of target genes detected in this study
基因 引物 上游引物 下游引物 退火温度/ ℃ 尺寸/碱基对 16S K90_K94 GCGGTGGAGCATGTGGTTTA GATAAGGGTTGCGCTCGTTG 58 181 cmlA cmlA-F_cmlA-R GCTGCTACTCCCCGTTAAGT TGCCTGCCCATCATTAGTCC 58 117 floR flo-F_flo-R GAGGGTGTCGTCATCTACGG GAGCATCGCCAGTATAGCCA 58 138 tetC tetC-F_tetC-R GCGGGATATCGTCCATTCCG GCGTAGAGGATCCACAGGACG 58 207 sul1 sul1-F_sul1-R CGCACCGGAAACATCGCTGCAC TGAAGTTCCGCCGCAAGGCTCG 58 163 intI1 intI2-F_intI1-R GGCTTCGTGATGCCTGCTT CATTCCTGGCCGTGGTTCT 60 146 表 3 PICRUSt2 功能预测的酶丰度
Table 3. Enzyme abundance predicted by PICRUSt2 function
酶序号 酶名称 初始 阶段1 阶段2 阶段3 阶段4 1.1.1.37 苹果酸脱氢酶 22 485.8 21755 29 120.04 32 928.95 32 668.61 1.1.1.41 异柠檬酸脱氢酶 1 949.75 2153 6 011.45 5 568.33 5 777.17 1.1.1.42 异柠檬酸脱氢酶 23 654.42 19 995.08 22 770.24 26 236.43 22 162.41 1.1.5.4 苹果酸脱氢酶(醌) 532.17 863.67 349.2 174.18 810.75 1.2.4.1 丙酮酸脱氢酶(乙酰转移型) 49 138.99 49 643.84 78 275.85 98 050.02 78 024.77 1.2.4.2 氧戊二酸脱氢酶(琥珀酰转移型) 9 122.5 7 602.17 5 147.71 3 644.06 5 785.25 1.2.7.1 丙酮酸合成酶 53 731.7 49 164.13 57 612.78 54 615.72 69 220.49 1.2.7.11 2- 氧代酸氧化还原酶(铁氧还原酶) 76 601.3 86 884.3 105667 117092 112 967.3 1.2.7.3 2-氧代戊二酸合成酶 117 248.2 131 264.3 152 519.3 164 382.5 157 703.1 1.3.5.1 琥珀酸脱氢酶(醌类) 65 107.02 62 133.5 64 476.58 67 416.56 54 060.32 1.3.5.4 富马酸还原酶(醌) 73 792.76 68 295.33 67 900.07 68 827.86 57 654.34 1.8.1.4 二氢脂酰脱氢酶 45 618.39 41 709.74 42 906.71 43 004.54 40 243.58 2.3.1.12 二氢脂酰赖氨酸-残基乙酰转移酶 27 940.3 25 631.5 38 049.53 46 427.03 39 255.27 2.3.1.61 二氢脂酰赖氨酸残基琥珀酰转移酶 15 497.7 14 852.17 13 348.71 13 464.95 17 538.25 2.3.3.1 柠檬酸合成酶 31 585.42 29 917.75 31 589.53 34 533.27 34 564.67 2.3.3.8 ATP 柠檬酸合成酶 12 813.84 8328 4 902.84 708.84 1 391.16 2.8.3.18 琥珀酰-CoA:醋酸 CoA-转移酶 558.12 809.17 632.21 765.65 665.39 4.1.1.32 磷酸烯醇丙酮酸羧激酶(GTP) 6 730.7 4 300.5 4 883.37 5 973.64 8 353.92 4.1.1.49 磷酸烯醇丙酮酸羧激酶(ATP) 23 010.55 23 612.75 23129 24 138.81 18 987.66 4.2.1.2 富马酸氢化酶 56 123.85 52 683.65 65 858.57 70 662.65 71 269.52 4.2.1.3 乌头酸水解酶 30 773.66 24 833.91 30 700.54 33 739.99 31 124.31 6.2.1.4 琥珀酸--CoA 连接酶(GDP 形成型) 1 5 0.01 0.01 0.01 6.2.1.5 琥珀酸--CoA 连接酶(ADP 形成型) 58 020.84 58 084.84 65 898.08 71 190.12 61 970.32 表 4 产甲烷动力学建模数据
Table 4. Kinetic modeling data of methane production
组别 甲烷潜势/(mL·(g·h)−1) 甲烷最大产生速率/(mL·g−1) 滞后时间/h R2 空白组 32.51 1.82 7.32 0.996 46 对照组 2 345.88 30.16 41.72 0.999 62 CNOs实验组 56.09 2.74 4.31 0.991 56 -
[1] AMELINE A, TAQUET M C, TERRADE J E, et al. Identification of chloramphenicol in human hair leading to a diagnosis of factitious disorder[J]. Clinical Toxicology, 2020, 58(9): 926-930. [2] ELDER F C T, PROCTOR K, BARDEN R, et al. Spatiotemporal profiling of antibiotics and resistance genes in a river catchment: Human population as the main driver of antibiotic and antibiotic resistance gene presence in the environment[J]. Water Research, 2021, 203: 117533. [3] AMBEKAR C S, CHEUNG B, LEE J, et al. Metabolism of chloramphenicol succinate in human bone marrow[J]. European Journal of Clinical Pharmacology, 2000, 56(5): 405-409. [4] GUO N, MA X F, REN S J, et al. Mechanisms of metabolic performance enhancement during electrically assisted anaerobic treatment of chloramphenicol wastewater[J]. Water Research, 2019, 156: 199-207. [5] LEVY S B, MARSHALL B. Antibacterial resistance worldwide: Causes, challenges and responses[J]. Nature Medicine, 2004, 10(12): S122-S129. [6] DING Y C, JIANG W L, LIANG B, et al. UV photolysis as an efficient pretreatment method for antibiotics decomposition and their antibacterial activity elimination[J]. Journal of Hazardous Materials, 2020, 392: 122321. [7] MURRAY I A, SHAW W V. O-Acetyltransferases for chloramphenicol and other natural products[J]. Antimicrobial Agents and Chemotherapy, 1997, 41(1): 1-6. [8] ALCALA A, RAMIREZ G, SOLIS A, et al. Structural and functional characterization of three Type B and C chloramphenicol acetyltransferases from species[J]. Protein Science, 2020, 29(3): 695-710. [9] LI N, HU Y, LU Y Z, et al. In-situ biogas sparging enhances the performance of an anaerobic membrane bioreactor (AnMBR) with mesh filter in low-strength wastewater treatment[J]. Applied Microbiology and Biotechnology, 2016, 100(13): 6081-6089. [10] ZHU M, LI N, LU Y, et al. The performance and microbial communities of an anaerobic membrane bioreactor for treating fluctuating 2-chlorophenol wastewater[J]. Bioresource Technology, 2020, 317: 124001. [11] GOLOVKO O, KUMAR V, FEDOROVA G, et al. Seasonal changes in antibiotics, antidepressants/psychiatric drugs, antihistamines and lipid regulators in a wastewater treatment plant[J]. Chemosphere, 2014, 111: 418-426. [12] KAYA Y, BACAKSIZ A M, BAYRAK H, et al. Treatment of chemical synthesis-based pharmaceutical wastewater in an ozonation-anaerobic membrane bioreactor (AnMBR) system[J]. Chemical Engineering Journal, 2017, 322: 293-301. [13] LIANG B, CHENG H Y, KONG D Y, et al. Accelerated Reduction of Chlorinated Nitroaromatic Antibiotic Chloramphenicol by Biocathode[J]. Environmental Science & Technology, 2013, 47(10): 5353-5361. [14] 马清佳, 田哲, 员建, 等. 9种抗生素对污泥高温厌氧消化的急性抑制[J]. 环境工程学报, 2018, 12(7): 2084-2093. doi: 10.12030/j.cjee.201712098 [15] LU J S, CHANG J S, LEE D J. Adding carbon-based materials on anaerobic digestion performance: A mini-review[J]. Bioresource Technology, 2020, 300: 122696. [16] ZHU M C, LU Y Z, CHEN S W, et al. Carbon nano-onions acting as artificial pili enhance chloramphenicol degradation in an anaerobic membrane bioreactor[J]. Chemical Engineering Journal, 2023, 475: 146110. [17] D’ AMORA M, RODIO M, BARTELMESS J, et al. Biocompatibility and biodistribution of functionalized carbon nano-onions (f-CNOs) in a vertebrate model[J]. Scientific Reports, 2016, 6(1): 33923. [18] LIU L, LU Y, PU Y, et al. Highly sulfonated carbon nano-onions as an excellent nanofiller for the fabrication of composite proton exchange membranes with enhanced water retention and durability[J]. Journal of Membrane Science, 2021, 640: 119823. [19] YIN X J, ZHAI J, HU W, et al. A fast start-up of the organotrophic anammox process inoculated with constructed wetland sediment[J]. Ecological Engineering, 2019, 138: 454-460. [20] ELDER F C T, PASCOE B, WELLS S, et al. Stereoselective metabolism of chloramphenicol by bacteria isolated from wastewater, and the importance of stereochemistry in environmental risk assessments for antibiotics[J]. Water Research, 2022, 217: 118415. [21] GUI C, CHEN J, XIE Q, et al. CytA, a reductase in the cytorhodin biosynthesis pathway, inactivates anthracycline drugs in[J]. Communications Biology, 2019, 2(1): 454. [22] WEN W H, ZHANG Y, ZHANG Y Y, et al. Reductive inactivation of the hemiaminal pharmacophore for resistance against tetrahydroisoquinoline antibiotics[J]. Nature Communications, 2021, 12(1): 7085. [23] LASSILA T, HOKKANEN J, AATSINK S M, et al. Toxicity of carboxylic acid-containing drugs: The role of acyl migration and CoA conjugation investigated[J]. Chemical Research in Toxicology, 2015, 28(12): 2292-2303. [24] BIZERRA A M C, MONTENEGRO T G C, LEMOS T L G, et al. Enzymatic regioselective production of chloramphenicol esters[J]. Tetrahedron, 2011, 67(16): 2858-2862. [25] GAUGAIN M, CHOTARD M P, HURTAUD P D, et al. Comprehensive validation of a liquid chromatography–tandem mass spectrometry method for the confirmation of chloramphenicol in urine including stability of the glucuronide conjugate and efficiency of deconjugation[J]. Journal of Chromatography B, 2016, 1011: 145-150. [26] RONG Y, TU Y F, YIN T J, et al. Rapid intestinal glucuronidation and hepatic glucuronide recycling contributes significantly to the enterohepatic circulation of icaritin and its glucuronides in vivo[J]. Archives of Toxicology, 2020, 94(11): 3737-3749. [27] MA X D, LIANG B, QI M Y, et al. Novel pathway for chloramphenicol catabolism in the activated sludge bacterial isolate sp. CAP-1[J]. Environmental Science & Technology, 2020, 54(12): 7591-7600. [28] VON WINTERSDORFF C J H, PENDERS J, VAN NIEKERK J M, et al. Dissemination of antimicrobial resistance in microbial ecosystems through horizontal gene transfer[J]. Frontiers in Microbiology, 2016, 7: 173. [29] GüNDOĞDU A, LONG Y B, VOLLMERHAUSEN T L, et al. Antimicrobial resistance and distribution of sul genes and integron-associated intI genes among uropathogenic Escherichia coli in Queensland, Australia[J]. Journal of Medical Microbiology, 2011, 60(11): 1633-1642. [30] CHENG D L, NGO H H, GUO W S, et al. Anaerobic membrane bioreactors for antibiotic wastewater treatment: Performance and membrane fouling issues[J]. Bioresource Technology, 2018, 267: 714-724. [31] 吉运, 邓宇, 杨爱江, 等. 氮氧自掺杂生物质多孔炭修饰阴极的生物电芬顿产电及其对氯霉素的降解性能[J]. 环境工程学报, 2022, 16(11): 3587-3595. doi: 10.12030/j.cjee.202201050 [32] 刘星鑫, 王安祺, 卓琼芳, 等. 改性Ti4O7阳极对氯霉素的高效电氧化降解[J]. 环境工程学报, 2023, 17(8): 2534-2543. doi: 10.12030/j.cjee.202303033 [33] 霍朝飞, 陈文燕, 张金凤, 等. 磁性山竹壳炭的制备及对氯霉素吸附性能[J]. 环境工程学报, 2023, 17(7): 2145-2157. doi: 10.12030/j.cjee.202302064 [34] 郑小清, 蒙德仲, 周新木. 废刀具中金刚石等有价物质的综合回收[J]. 江西化工, 2008(4): 197-199. doi: 10.3969/j.issn.1008-3103.2008.04.062 [35] 张卫珂, 付俊杰, 常杰, 等. 纳米洋葱碳的制备及其纯化研究[J]. 新型炭材料, 2014, 29(5): 398-403. -


 下载:
下载: