-
由于抗生素在农业[1]和医疗[2]环境的频繁使用,使得抗生素抗性成为21世纪6大难题之一[3]。抗生素的持续性选择压力,使得抗生素抗性基因(Antibiotic resistance genes,ARGs)不仅出现在人类活动频繁的环境[1-2, 4-5],在远离人类的地区也有报道[6-7],因此人类对ARGs的不断扩散非常担忧。冰川环境因抗生素选择压力比较小,能反应微生物对抗生素的固有抗性,因此是抗生素抗性风险评估的重要环境。该环境ARGs的研究有2种不同的结果:VAN GOETHEM et al[8]使用宏基因组测序技术,在南极Mackay冰川表层土壤中检测到的ARGs序列与其他环境具有不同的聚类分枝,这些ARGs是其进化的结果。然而,WANG et al[9]对南极冈瓦纳研究站的研究结果表明,人类的长期和短期活动都会影响ARGs在南极的传播,但长期影响和短期影响的结果差不多。OKUBO et al[10]在1200~1400年的南极冰芯检测到sul2,虽然赋予人工合成的磺胺甲恶唑抗性的基因在抗生素时代之前就已经存在,但sul2-strA-strB基因簇与当今细菌具有非常高的序列相似性,该结果与VAN GOETHEM et al[8]的报道相反。
ARGs不仅会受到抗生素的选择压力,重金属同样也会对其产生影响[11–12],如Cu会选择临床上重要的抗生素—万古霉素的耐药性[13]。重金属污染具有持久性,能在环境中存在数百年,其对ARGs的选择压力甚至比抗生素自身还要严重。重金属同样也会选择重金属抗性基因(Heavy metal resistance genes,HMRGs),因此ARGs和HMRGs可同时出现,使得微生物产生多重抗性。这类研究多集中在人类活动频繁的环境,如污水处理厂[14]、活性污泥[15]、农业土壤[1]、医院废水[2]、肥料[16]、城市污水和饮用水[17]、富营养化的地表水中[18],但尚未见到冰川环境HMRGs的研究。而且上述人类活动频繁的环境HMRGs的研究多集中在Cu、Zn、Hg、Ag、As、Ni和Cd等的抗性基因,关于Te和Mn等的抗性基因尚未见报道。
冰川雪冰中重金属的浓度非常低,有时候甚至低于pg/g(10−12 g/g)水平[19],给其研究带来诸多困难。由于HMRGs的丰度与重金属浓度的密切相关性[20],以及荧光定量PCR(Real-time Quantitative PCR,qPCR)的敏感性,HMRGs可代替重金属,从侧面反应冰川中重金属的存在状况。作者之前通过宏基因组测序技术,在青藏高原西昆仑山的崇测冰帽冰川沉积物检测出Zn(9.13%)、Cu(22.64%)、Fe(14.76%)、As(7.35%)、Ni(0.61%)、Mo(9.86%)、Mn(9.16%)、Co(5.38%)、Hg(2.40%)、Cr(6.36%)、Cd(5.50%)、W(0.75%)和Te(6.09%)这13种HMRGs,其中Te和Mn所占百分比并不低,位于第4和第8位。本研究以距离崇测冰帽比较近的青藏高原喀喇昆仑山脉德普昌达克冰川,以及南极中山站为研究位点,通过qPCR,检测代表Hg、Cu、Zn、Cr、Te、Mn和Cd等金属的抗性基因,人工合成的氟喹诺酮类、磺胺类的抗性基因,天然的四环素和红霉素类抗性基因,以及在耐药性传递方面起重要作用的I型整合子(class I integron,intI),一方面初步判断ARGs与HMRGs在不同研究位点存在与否及相对丰度,另一方面了解ARGs、HMRGs与intI之间的相关性。该研究有助于了解冰川环境ARGs的存在状况,以及重金属和intI对其影响。
全文HTML
-
2016年7月,采集德普昌达克冰川不同海拔高度的3个雪样:德普昌1(34°16'15.79″N,81°10'55.76″E,5433.78 ma.s.l)、德普昌2(34°16'16.42″N,81°10'55.76″E,5422.40 ma.s.l)和德普昌3(34°16'17.81″N,81°10'55.80″E,5411.75 ma.s.l),运输至商丘师范学院河南省特色微生物资源开发与应用工程研究中心,在洁净室进行雪样融化与过滤及DNA提取。
2016年2月,中国第32次南极科考队在中山站采集3个雪样:中山1(69°22′22.93″S,76°22′05.94″E,11 m a.s.l,站区莫愁湖畔)、中山2(69°22′17.74″S,76°22′11.18″E,20 m a.s.l,正北方六角楼下)和中山3(69°22′24.03″S,76°21′30.72″E,25 m a.s.l,站区西边丘陵顶部)。所采样品经雪龙号运输至上海。由上海运输至南京大学海岸与海岛开发教育部重点实验室的低温室进行保存。2016年7月,在该重点实验室万级洁净室进行雪样融化与过滤及DNA提取。再将提取的DNA用冰袋和塑料泡沫盒运输至商丘师范学院,进行后续的qPCR分析。
为保证所采雪样不受人为污染,采样者在采样过程中一律穿洁净服、戴口罩和PE手套。用无菌不锈钢铲去掉表层雪样1 cm,5点梅花取样法采取1 cm以下的表层样品。所采雪样装入无菌采样瓶,将每个采样瓶装入克林莱密实袋,以避免外部污染。所有样品置于冰柜中,以低温(≤–5 ℃)方式运回实验室。
-
雪样置于4 ℃缓慢融化后,在万级洁净室内的超净工作台上,将约400~700 mL融水用小型切向流超滤系统(Labscale,Millipore)配套0.22 μm无菌Millipore微孔滤膜进行过滤。用灭菌剪刀将收集雪样的滤膜剪碎,置于2 mL离心管,按照DNA提取试剂盒(FastDNA Spin Kit for Soil,MP)说明书进行DNA提取。提取的DNA用微量紫外-可见光分光光度计(Thermo Scientific™ NanoDrop™ One)测OD值。所有样品DNA的A260/A280均介于1.8~2.0。
-
所涉及目标基因包括8个ARGs、和3个intI,16S rDNA作为分析时的内参基因。引物名称、序列、片段长度和退火温度等,见表1。
qPCR反应体系为:SYBRR Premix Ex TaqTMⅡ(TliRNaseH Plus, Takara)10 µL,上下游引物(10 µmol/L)各0.8 µL,10 ng/µL DNA模板1.0 μL,ROX Reference Dye(50×)0.4 µL,无菌ddH2O7 µL。反应程序:95 ℃预变性1 min;95 ℃变性10 s,不同引物退火温度下40 s,72 ℃延伸30 s,共40个循环。用无菌ddH2O代替DNA模板作阴性对照,每个样品3个重复。
-
试验数据以3次重复的平均值表示,目标基因的相对丰度为其浓度与16S rDNA浓度的比值。用SPSS 22.0软件的Spearman对ARGs、HMRGs和intI的相对丰度进行相关性分析。
1.1. 样品采集
1.2. DNA提取
1.3. qPCR
1.4. 数据分析
-
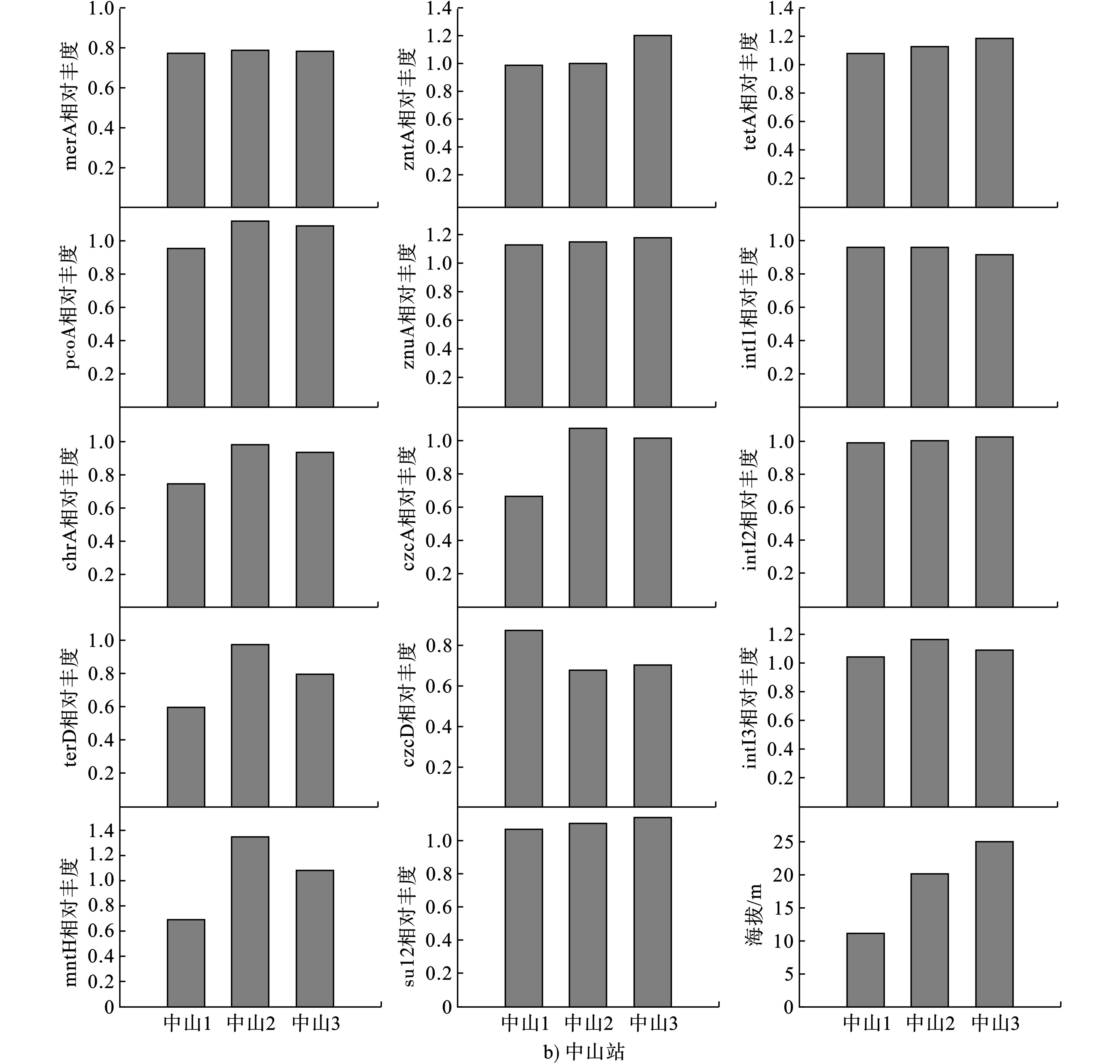
在8种ARGs中,aac(6')-Ib-cr仅在德普昌达克冰川被检出,sul1和ermB仅在中山站被检出,而qnrA、tetY和tetM在2个位点均未被检出。仅sul2和tetA在德普昌达克冰川和中山站2个位点都能被检出。这2种ARGs在2个位点的分布,见图1。
图1可见,sul2的相对丰度在德普昌达克冰川不随海拔增加而递增或递减,tetA的相对丰度在德普昌达克冰川随海拔升高而降低,sul2和tetA的相对丰度在中山站随海拔升高而增加。
aac(6')-Ib-cr的相对丰度在德普昌达克冰川的分布与sul2相同,不随海拔增加而递增或递减。sul1的相对丰度在中山站也未因海拔的增加而递增或递减。ermB的相对丰度在中山站的分布与sul2和tetA相同,随海拔升高而增加。
-
9个HMRGs在德普昌达克冰川和中山站均能被检测到,但在2个研究位点随海拔高度的变化,相对丰度的分布趋势有所差异。图1可见,znuA、czcD和pcoA的相对丰度在德普昌达克冰川随海拔升高而降低。然而,merA、chrA和mntH的相对丰度则与海拔高度呈正比。在南极,仅merA、znuA和zntA的相对丰度随海拔变化呈现出规律性变化,即随海拔的升高而增加。因此,merA在德普昌达克冰川随海拔的变化趋势与中山站相一致,而znuA随海拔的变化趋势却与中山站相反。
HMRGs的相对丰度在德普昌达克冰川和中山站这2个位点之间也有差异。如,terD、mntH、pcoA、znuA、chrA、zntA和czcA在德普昌达克冰川的相对丰度平均值低于中山站,而merA和czcD则相反。
-
3个intI在2个研究位点均被检出。图1可见,3个intI在德普昌达克冰川均低于中山站。intI3在2个位点不因海拔的增加而递增或递减,intI1在德普昌达克冰川不随海拔的增加而递增或递减,但在中山站随海拔升高而递减。intI2在德普昌达克冰川随海拔升高而降低,但在中山站则相反,即随海拔升高而递增。
对2种ARGs(sul2和tetA)、9种HMRGs和3种intI进行相关性分析,结果见表2。
表2可知,由于每个位点只有3个样品,因此相关系数为1的2个因数才具有显著相关性。在德普昌达克冰川,sul2和tetA分别与不同的HMRGs和intI相关联。sul2与terD、czcA、intI1和intI3显著相关;tetA与czcD、pcoA、znuA和intI2显著相关。在中山站,sul2与tetA正相关,与二者密切相关的HMRGs和intI也相同。如merA、zntA、znuA和intI2均与sul2和tetA显著相关,intI2还同时与merA、zntA和znuA显著相关。
2.1. ARGs
2.2. HMRGs
2.3. ARGs、HMRGs和intI的相关性
-
自然环境中重金属对ARGs影响的研究较少,尤其是冰川这种环境,重金属元素的自然本底值很低,给其研究带来一定难度。由于HMRGs与重金属之间密切相关性,可以借助HMRGs来反应冰川环境重金属的水平。本文通过对青藏高原德普昌达克冰川和南极中山站雪样ARGs、HMRGs和intI的相对丰度进行分析,来判断远离人类的冰川环境重金属和intI如何影响ARGs。
德普昌达克冰川的2个ARGs中,仅tetA随海拔的变化呈现规律性变化,与之变化相一致的是czcD、pcoA、znuA、intI1和intI2,这些基因均随海拔高度的增加而降低。YANG et al[39]对青藏高原湿地中ARGs的研究结果也证明了海拔越高,ARGs的总丰度越低,sul等基因的丰度越低。这些结果说明高海拔的地区因人类活动强度低,因而这些ARGs、HMRGs和intI含量通常较低,因此,ARGs、HMRGs和intI可以看作是人类活动强度或人为污染的代表。
南极中山站的merA、zntA、znuA、sul2、tetA和intI2均随海拔升高而升高。由于3个样品海拔高度在11~25 m之间,差别不大,因此其差异并非海拔所引起。但从样品1到样品3,距离中山站建筑区域的距离降低,人类活动就会越来越频繁。因此,上述6个基因的相对丰度就会随人类活动的增加而提升。方精云[40]对北极的研究也说明人类活动较为密集的区域周边的Mn含量会上升。
虽然2个研究位点ARGs、HMRGs和intI的分布有差异,但也有共性。比如,ARGs均受Zn抗性基因znuA的影响,而且znuA与intI之间显著相关,说明代表Zn的抗性基因与intI一起会影响ARGs在冰川上的分布。Zn与农业及水产养殖关系密切[41–42],这说明影响ARGs分布的是与人类活动密切相关的重金属。znuA对tetA和sul2的影响大于pcoA,这与Zn对ARGs的影响大于Cu的报道[30]相一致。
但是有些HMRGs在冰川上的分布可能与其他因素有关,因此不能仅依赖相关性来判断重金属对ARGs的影响。如merA在德普昌达克冰川和中山站均随海拔升高而增加,这与Hg具有“海拔效应”的报道[43]相一致。即Hg和其相对应的抗性基因merA在冰川雪样中会因海拔升高引起的温度降低而提高其沉积效率。Cr分布在较大的微粒上[44],而微粒的含量在冰川表层雪中随海拔升高,总体上呈明显的递减趋势[45]。因此,chrA在德普昌达克冰川随海拔的增加而降低。merA和chrA在冰川上的分布或许可以反映其他环境因子的变化。
ARGs、HMRGs和intI不仅具有海拔上的差异,还有空间上的区别。sul2、tetA、3个intI、zntA、pcoA、znuA、terD、chrA、czcA和mntH在德普昌达克冰川的相对丰度平均值低于中山站。这与青藏高原冰川表层雪的Sb浓度[46]、可培养细菌菌落形成单位[47]比南极高的报道均相反。这可能是由于中山站采样位点距离科考站比较近,科考队员的频繁活动增加了ARGs、HMRGs和intI的丰度。人为影响程度越深的地方,ARGs浓度和多样性就越高,这种现象在垃圾填埋场[48]、河流、河口和海岸[49]等地均有报道。
在远离人类的环境,HMRGs与ARGs的相关性说明了重金属对ARGs的影响。冰川环境的选择压力包括永久性的低温、高剂量的紫外线照射、每日冻结以及通常累积并存储在冰晶洞中的重金属和放射性核素[50],所有这些都可能增强ARGs。因此,要想降低ARGs的产生和扩散,必须从抗生素和重金属等多方面去考虑。tetA和sul2的抗性机制分别为外排泵和靶标替代,二者在中山站均与intI2相关。intI不仅可以影响tetA和sul2的扩散,还可以使2种抗性机制不相同的ARGs共同出现在同一种细菌,从而给抗生素药物的治疗带来一定难度。
-
本研究通过qPCR,分析了青藏高原德普昌达克冰川和南极中山站不同海拔及不同研究位点ARGs、HMRGs和intI相对丰度的差异。结果表明,德普昌达克冰川ARGs、HMRGs和intI的相对丰度并不比南极中山站高。2个冰川的研究都说明,虽然冰川环境远离人类活动,但ARGs受intI的影响,同时还与人类活动有关的重金属Zn有关。然而,上述研究仅基于qPCR及相关性分析,还需用高通量qPCR以及重金属分析的结果,判断重金属对ARGs的影响,以及从序列相似性角度判断冰川环境的ARGs、HMRGs和intI是原有的还是人类活动输入的。


 下载:
下载:

