-
细菌耐药性感染日益影响人类健康和经济发展[1-2]。英国卫生部报告显示,若不采取有效措施,到2050年,细菌耐药性将导致全球每年出现1 000万人死亡[3]。耐药基因 (antibiotic resistance genes,ARGs) 是细菌获得耐药表型的关键遗传载体,故被列为全球重要的新兴环境污染物之一[4-6]。
城市水系统中的污水处理系统被证明是ARGs的重要储存库之一,同时也是ARGs污染传播至受纳环境的主要来源[7-8]。在污水处理系统中,ARGs的产生和增殖与污水中选择压密切相关[8-9]。已有研究报道了纳米银[10-11]、纳米铜/氧化铜[12-14]、纳米氧化锌 (nZnO) [15]和纳米零价铁 (nZVI) [16-17]等纳米金属及氧化物对污水处理系统活性污泥中总体ARGs分布与归趋的影响。MILLER等[10]通过实时荧光定量聚合酶链反应 (qPCR) 证明,环境浓度纳米银 (0.01~1.0 mg·L−1) 对厌氧消化器中四环素和磺胺类ARGs的丰度无显著影响。MA等[11]通过宏基因组测序分析发现,经过56 d的纳米银 (0.1~20 mg·L−1) 驯化,活性污泥中绝大多数类型ARGs的分布无明显变化,但可促使氨基糖苷类ARGs (如strA) 和吖啶黄类ARGs (acrB) 分别提高300%、50%。此外,ZHANG等[15]发现,nZnO颗粒导致污泥厌氧消化系统中可移动遗传元件 (mobile genetic elements,MGEs) 绝对丰度增加了354.70%,因此可能会增加ARGs的传播风险。最近两项研究基于相关性分析得出,nZVI主要通过改变ARGs潜在微生物寄主降低氯霉素废水处理系统[16]和厌氧消化系统[17]中ARGs的丰度。需要明确的是,污水处理系统中ARGs根据其形态可分为污泥细胞内ARGs (iARGs) 、污泥细胞外附着态ARGs (aeARGs) 和污水细胞外游离态ARGs (feARGs) [18-19]。ARGs的赋存形态不同,其增殖与传播方式亦不相同[19-20]。iARGs主要通过细胞分裂或水平接合进行增殖,而aeARGs和feARGs主要来源于细胞死亡裂解或主动分泌,并能借助转化和转导机制传播[19,21-22]。然而,金属纳米粒子对污水处理系统中不同形态ARGs增殖与传播能力的影响及其机制尚未探明。
本研究采用抗生素敏感性测验和qPCR考察不同质量浓度nZVI和nZnO暴露对污水处理系统中典型磺胺类耐药菌 (ARB) 浓度和不同形态ARGs丰度的影响,并通过检测细胞膜通透性、ARGs表达水平及典型MGEs (intI1) 分布特征阐明nZVI和nZnO暴露下ARGs的转变机制,有助于揭示金属纳米粒子和细菌耐药性的双重新兴环境污染效应,以期为研发高效控制污水处理系统中耐药污染的策略提供参考。
-
活性污泥取自某主体工艺为厌氧/缺氧/好氧-膜生物反应器的污水处理厂。活性污泥混合液挥发性悬浮物浓度 (MLVSS) 为2.2 g·L−1。nZVI和nZnO等试剂购自阿拉丁 (Aladdin,中国) 。试剂级别均为分析纯。
-
将活性污泥静置后去上清液,采用质量分数为0.85%的NaCl清洗1次。用量筒量取300 mL活性污泥混合液至500 mL锥形瓶中。参照某污水处理厂进水基质水平添加CH3COONa 171.4 mg·L−1 (相当于COD为200 mg·L−1) 、NH4+-N 20 mg·L−1、NO3−-N 10 mg·L−1和PO43--P 2 mg·L−1。然后,投加适量预先超声分散 (5 min) 的nZVI和nZnO储备液至锥形瓶中,使其最终质量浓度维持为1和50 mg·L−1 (分别代表污水处理厂中环境检出水平和污水生物处理胁迫实验质量浓度) [23-25]。最后,将其置于培养箱 (ZQZY-70BF,中国) 中恒温振荡 (20 °C,150 r·min−1) 反应12 h。运行模式参照某实际污水处理厂采用6 h厌氧+6 h好氧的曝气方式,曝气量控制为500 L·(m3·min)−1。分别于0.5、2、4、6、8、10和12 h取污泥混合液用于ARB浓度数检测及细胞膜完整性检测;同时,取污泥混合液进行离心,获得的污泥沉淀和上清液保存于-20 °C,用于RNA和不同形态DNA提取。
-
采用琼脂稀释法[26]检测ARB相对浓度。采用无菌0.85% (质量分数) NaCl溶液10倍梯度稀释活性污泥混合液至适当细菌浓度。取1 mL稀释后的样品涂布于含有磺胺 (终浓度为512 mg·L−1) 的MH琼脂上。抗生素浓度遵循临床和实验室标准协会指南[27]的最低抑制浓度。同时,取1 mL 0.85% (质量分数) 无菌NaCl溶液涂布在无菌MH琼脂平板上用作空白对照。最后,将平板倒置于(36±1) °C生化培养箱培养(48±2) h。选取菌落数为30~300菌落形成单位的平板计数菌落总数。ARB相对浓度为添加抗生素的实验组菌落数与抗生素空白组菌落数 (即异养菌浓度) 的比值。
-
取5 mL污泥混合液离心 (4 °C、4 000 r·min−1) 处理5 min,保留污泥沉淀,并将上清液转移至新的离心管中。采用离子交换树脂法[28]提取污泥沉淀中的细胞外附着态DNA (aeDNA) 。首先,向污泥沉淀加入适量离子交换树脂 (70 g·g−1 MLVSS) ,补充磷酸盐缓冲液至5 mL;置于4 °C磁力水浴槽内以600 r·min−1处理8 h后,并于4 °C、以10 000 r·min−1离心5 min;取上清液过0.22 μm滤膜,即得aeDNA粗提取液。接着,取污泥沉淀,采用十二烷基硫酸钠 (SDS) -高盐法提取胞内DNA (iDNA) [29]。向污泥沉淀中加入810 μL iDNA抽提液和2 μL蛋白酶K (20 mg·mL−1标准品) ,并于37 °C、以225 r·min−1水平振荡30 min;加入60 μL 20% (质量分数) SDS,置于65 °C水浴锅中水浴2 h;室温离心 (10 000 r·min−1、10 min) 后,取上清液转移至新的2 mL离心管中;向剩余沉淀加入180 μL iDNA抽提缓冲液和20 μL 20% (质量分数) SDS,重复上述处理2次;合并3次处理获得的上清液,即为iDNA粗提取液。最后,采用苯酚-氯仿法[30]进一步纯化aeDNA和iDNA粗提取液。采用超微量分光光度计 (Nanodrop 2 000,美国) 测定DNA浓度与纯度[31-32]。纯化后aeDNA和iDNA样品保存于-20 °C冰箱。采用0.22 μm滤膜过滤上述污泥混合液离心获得的上清液。取4 mL滤液至50 mL离心管中,采用乙醇沉淀法[33]分离游离态DNA (feDNA) 。首先,向离心管中添加2 mL醋酸铵 (终浓度7.5 mol·L−1) 和12 mL 4 °C预冷无水乙醇,颠倒混匀后于冰上静置1 h。接着,于4 °C、以14 000 r·min−1离心30 min后,小心移除上清液;加入1 mL 4 °C预冷的70% (体积分数) 乙醇,并将乙醇与沉淀混合液转移到一个新的2 mL离心管中;重复此操作1次,使feDNA完全转移至2 mL离心管中。最后,将上述DNA悬浮液离心 (4 °C、14 000 r·min−1、10 min) 后,用70% (体积分数) 乙醇洗涤,并将feDNA沉淀悬浮于50 μL无菌无酶水中。利用PicoGreen dsDNA Quantitation Kit (Invitrogen,中国) 和多功能酶标仪 (BioTek,美国) 测定feDNA 浓度。
-
通过TRIzol试剂 (Invitrogen,中国) 抽提活性污泥中的RNA。取2 mL活性污泥离心 (4 °C、8 000 r·min−1、5 min) 后弃上清液,加入1 mL TRIzol试剂涡旋后,室温静置1 h。于4 °C、12 000 r·min−1离心10 min,将上清液转移至新离心管;加入0.2 mL氯仿后,室温静置2 min。将上述混合液离心 (4 °C、12 000 r·min−1、5 min) 后,取0.5 mL上层水相至新离心管;加入0.5 mL异丙醇,于−20 °C放置20 min。离心 (4 °C、12 000 r·min−1、20 min) 后弃上清液,加入1 mL 75% (体积分数) 预冷 (4 °C) 乙醇;颠倒混匀后离心 (4 °C、8 000 r·min−1、5 min) 后弃上清液,并于室温风干。最后,加入30 μL无RNA酶去离子水溶解RNA沉淀。采用超微量分光光度计 (Nanodrop 2 000,美国) 测定RNA初始浓度及纯度。采用PrimeScriptTM RT reagent Kit with gDNA Eraser试剂盒 (Takara,中国) 对RNA进行反转录。先将2 μL RNA和2 μL 5×gDNA Eraser Buffer、1 μL gDNA Eraser及适量RNase Free ddH2O (总体积10 μL) 添至200 μL PCR反应管1内,吸打混匀。置于PCR仪 (Eppendorf,德国) 中42 °C反应2 min后,将混合液转至反应管2中。再加入4 μL 5×PrimeScript®Buffer2、1 μL PrimeScript®RT Enzyme MixI、4 μL RNase Free ddH2O和1 μL RT Primer Mix,吸打混匀后将PCR反应管2置于PCR仪,于37 °C下反应15 min。最后在85 °C反应5 s进行反转录,生成cDNA于−20 °C保存。
-
采用qPCR定量分析污水处理系统典型磺胺类ARGs (sul1和sul2) 和intI1的丰度和表达水平。反应体系 (20 μL) 为:10 μL SuperReal PreMix Plus (TIANGEN,中国) ,0.6 μL正引物和反引物,2 μL DNA模板和6.8 μL ddH2O。反应条件为:95 °C预变性 1~2 min,95 °C变性 30 s,退火30 s,72 °C延伸 30 s,共40个循环。sul1、sul2、intI1和16S rDNA的引物序列和退火温度参见文献[34-36]。所有样品一式3份。
-
采用LIVE/DEAD BacLight Bacterial Viability Kits (Invitrogen,中国) 检测细菌细胞膜完整性。取0.5 mL污泥混合液离心 (4 °C、8 000 r·min−1、5 min) 弃上清液后,加入1 mL质量分数为0.85%的NaCl缓冲液,于10 000 r·min−1下离心15 min。污泥沉淀重悬于适量0.85% (质量分数) NaCl缓冲液 (OD600约为0.05) 后,取100 μL上述污泥混合液与等体积染料,吸打混匀后室温孵育15 min。采用多功能荧光酶标仪 (BioTek,美国) 测定样品的荧光值。激发波长设置为485 nm,发射波长为530 nm和630 nm。所有样品一式3份。
-
ARGs相对丰度为单位质量 (ng) DNA中ARGs拷贝数与16S rDNA拷贝数之比。所有图形均使用Origin 20.0进行绘制。图中误差棒表示2次或3次实验数据的标准差。
-
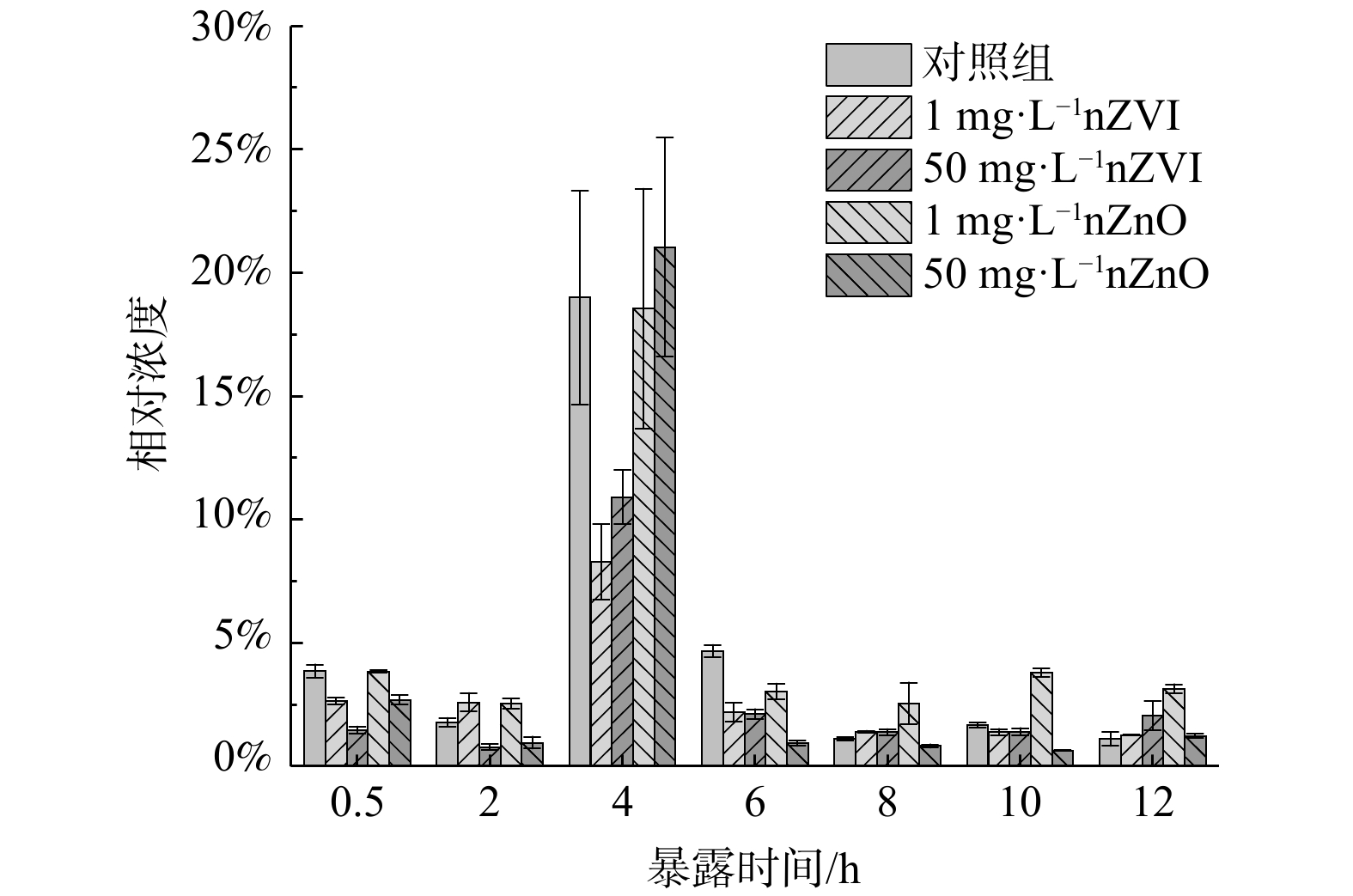
nZVI和nZnO暴露已被证明能有效抑制鲍曼不动杆菌、金黄色葡萄球菌、拟杆菌、芽孢杆菌、棒状杆菌、硫假单胞菌等ARB生长[37-39]。然而,nZVI和nZnO暴露对污水处理系统中ARB的影响尚未可知。因此,将活性污泥暴露于不同浓度 (1和50 mg·L−1) nZVI和nZnO,于不同时间 (0.5、2、4、6、8、10和12 h) 进行取样检测。如图1所示,不同浓度nZVI和nZnO暴露下污泥中磺胺类ARB的相对浓度为0.65%~21.04%,且经nZVI和nZnO暴露后有所下降。具体而言,经过4 h nZVI和nZnO暴露后ARB相对浓度的水平出现大幅上升,至5.65%~18.34%,随后下降至初始水平并保持稳定。对比nZVI和nZnO暴露组与对照组数据发现,ARB变化可能主要由异养菌浓度的波动所致。进一步分析不同浓度nZVI和nZnO对ARB的影响发现,在0~6 h (除4 h),1 mg·L−1 nZVI暴露下ARB相对浓度明显高于50 mg·L−1 nZVI;而在8~12 h,50 mg·L−1 nZVI 暴露下的ARB相对浓度逐渐超过1 mg·L−1 nZVI。这可能是因为nZVI在好氧处理阶段 (6~12 h) 更易于氧化,从而降低了对ARB的抑制作用[40]。此外,1 mg·L−1 nZnO在暴露期间对ARB无明显影响,而50 mg·L−1 nZnO在暴露期 (除4 h外) 会明显抑制ARB增殖。
-
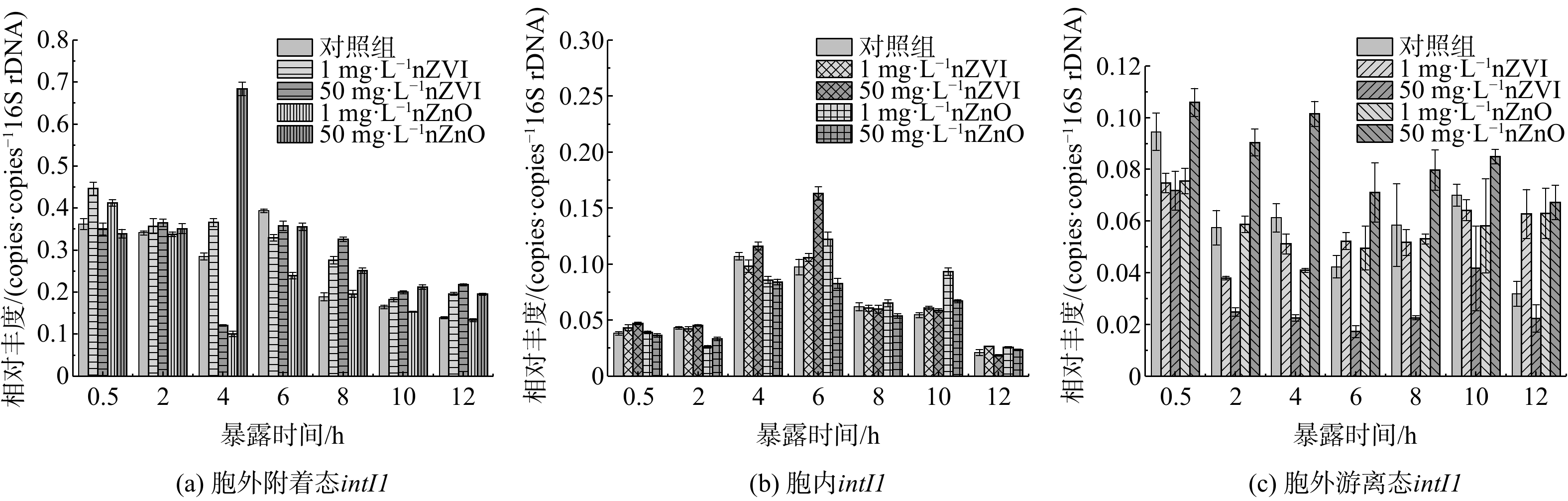
nZVI和nZnO暴露下3种不同形态ARGs的分布情况如图2所示。污泥中aeARGs和iARGs的相对丰度分别为3.34×10−2~2.83×10−1 copies·copies−1 16S rDNA和2.31×10−2~9.12×10−2 copies·copies−1 16S rDNA,且在暴露于nZVI和nZnO (除6 h外) 后持续降低。经过12 h nZVI和nZnO暴露,磺胺类aeARGs和iARGs的相对丰度分别降低了32.02%~71.69%和5.29%~20.55%。显然,nZVI和nZnO暴露对aeARGs分布的扰动作用明显强于iARGs。需要指出的是,nZVI和nZnO暴露后sul1相对丰度削减率低于对照组0.25%~16.21% (除1 mg·L−1 nZnO外) ,而sul2相对丰度的则高于对照组1.51%~15.47%。这说明纳米金属及其氧化物暴露对aeARGs和iARGs的削减能力可能取决于ARGs的类型。污泥厌氧消化过程中添加nZVI[17,41-42]或nZnO[43]有利于提高sul1、sul2、tetX和gryA的相对丰度,但同时会削减tetM、ermX、ermf和tet36的相对丰度。此外,相较对照组而言,nZVI和nZnO暴露12 h后的iARGs相对丰度下降了6.42%~10.47%,而aeARGs仅在1 mg·L−1 nZVI和nZnO暴露组分别下降5.31%和11.15%。进一步对比发现,50 mg·L−1 nZVI暴露下aeARGs丰度的削减比例比1 mg·L−1 nZVI低5.51%~33.15%;相反,50 mg·L−1 nZVI暴露下iARGs丰度的削减幅度明显高于1 mg·L−1 nZVI (图2(a)~(b)) 。此外,50 mg·L−1 nZnO暴露对aeARGs和iARGs相对丰度的削减比例均小于1 mg·L−1 nZnO暴露 (图2(a)~(d)) 。
由图2(e)和(f)可知,nZVI和nZnO暴露后feARGs相对丰度为1.53×10−2~3.32×10−1 copies·copies−1 16S rDNA,且随暴露时间增加逐渐降低。此外,与对照组相比,nZVI和nZnO暴露明显促进了feARGs的增殖。在河水[44]或海水[45]中添加nZnO已被证明有利于sul1富集。进一步对比不同浓度nZVI和nZnO暴露下feARGs的变化可知,nZVI暴露对feARGs的影响呈现低促高抑规律;而50 mg·L−1 nZnO暴露会持续刺激feARGs增殖 (图2(e)~(f)) 。这可能是由于50 mg·L−1 nZnO能导致ARB大量裂解 (图1) ,进而使iARGs释放,转变成feARGs。
-
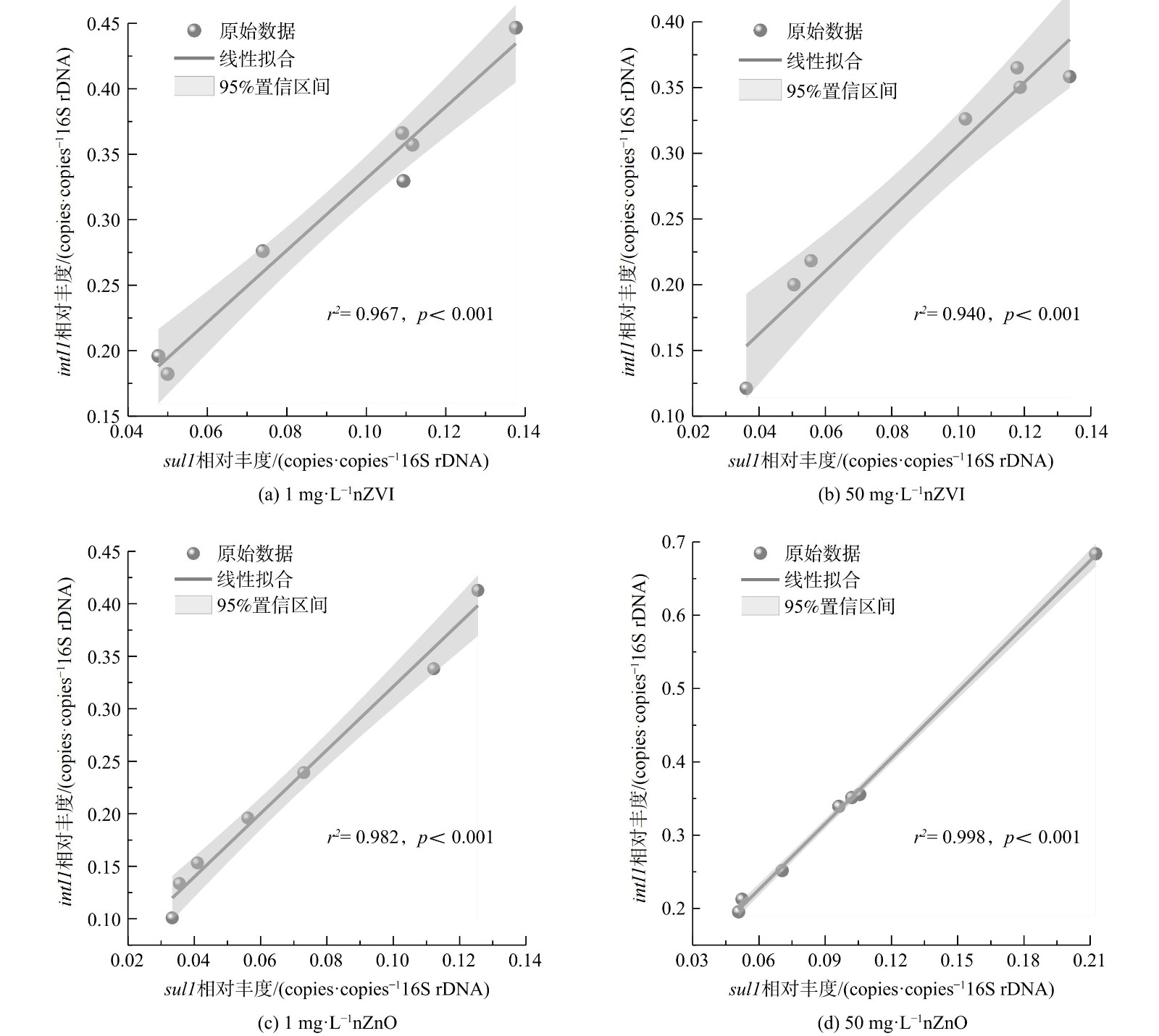
1) ARGs可移动性。磺胺类ARGs的动态转变通常与可移动遗传元件——intI1密切相关[19,46]。因此,重点探究了nZVI和nZnO暴露下intI1的分布特征。如图3所示,nZVI和nZnO暴露12 h后,胞外附着态、胞内和胞外游离态intI1的相对丰度分别降低了37.70%~67.65%、34.06%~60.30%和16.01%~68.87%。然而,nZVI和nZnO暴露12 h后intI1的相对丰度高于对照组。同时,胞外附着态sul1的相对丰度与intI1相对丰度呈现显著相关性 (图4) 。这说明nZVI和nZnO胁迫可能通过富集intI1促进sul1增殖。在粪便堆肥过程中,nZVI和nZnO暴露下intI1与ARGs具有明显的正相关性[42]。
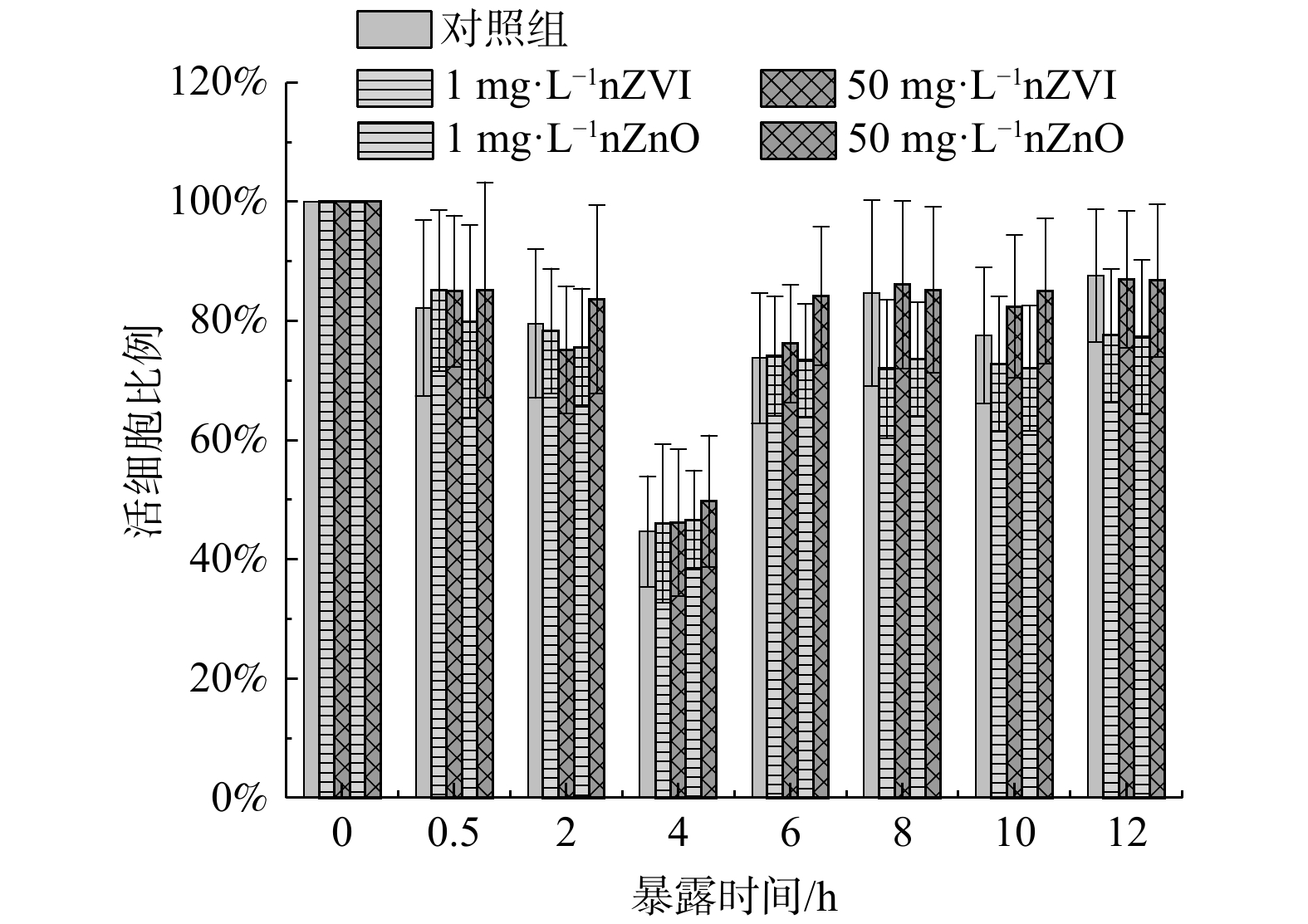
2) 细胞膜通透性。nZVI和nZnO暴露下污泥中细菌的活细胞比例如图5所示。在4 h时,污泥细菌活细胞比例发生急剧下降 (50.22%~55.30%) ,这在一定程度上解释了同时段发生的污泥aeARGs丰度增加的现象 (图2 (a)~(b) ) 。值得注意的是,在8~12 h暴露期间 (好氧阶段) ,1 mg·L−1 nZVI使污泥中细菌的活细胞比例分别降低了22.38%~28.00%,而50 mg·L−1 nZVI暴露组活细胞比例无明显变化。这可能是因为nZVI在有氧状态下更易于氧化,从而失去了抑菌能力[40]。
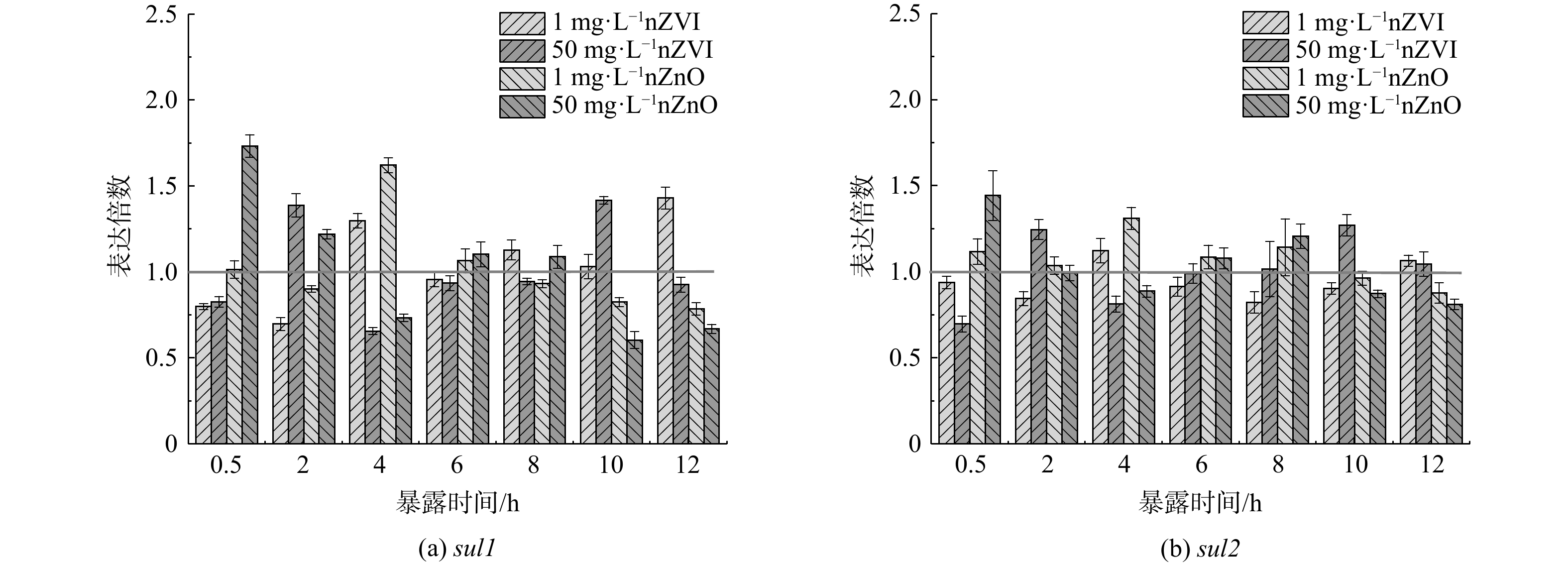
3) ARGs转录水平。转录可能是病原菌调控ARGs丰度水平以抵御抗生素攻击的方式之一[47-48]。由图6可知,nZVI和nZnO暴露会显著改变sul1和sul2的表达水平。如1 mg·L−1 nZVI与nZnO暴露4 h后sul1表达水平分别是对照组的1.30和1.62倍,而50 mg·L−1 nZVI和nZnO暴露导致sul1表达水平急剧下降为对照组的0.66和0.73倍。此外,nZVI和nZnO对sul1和sul2表达水平的影响会随时间发生波动。再者,nZVI和nZnO对sul1表达水平的扰动幅度也明显高于sul2。因此,nZVI和nZnO暴露能调控污泥中细菌内ARGs的转录过程,且因ARGs类型不同而迥异。
-
nZVI和nZnO暴露有效降低了污水处理系统中磺胺类ARB相对浓度,且nZVI表现出更强的削减效果。nZVI和nZnO暴露可有效削减磺胺类aeARGs和feARGs的相对丰度,对iARGs的影响较小。nZVI和nZnO暴露可能通过诱导污泥中的intI1增殖、改变细胞膜通透性和调节细菌转录水平加剧ARGs的传播与扩散风险。典型纳米金属对污水处理系统中ARB和不同形态ARGs消长的影响及其机制可为制定有效调控和全面削减污水处理系统中耐药污染策略提供参考。
纳米金属对污水处理系统中磺胺耐药基因和耐药菌的影响及其机理
Effects and mechanisms of metallic nanoparticles on the fate of sulfanilamide antibiotic resistance genes and resistant bacteria in wastewater treatment system
-
摘要: 细菌耐药性和纳米材料污染为新兴环境问题。细菌耐药性遗传载体——耐药基因 (ARGs) 广泛存在于污水处理系统中,且已被证明与金属纳米材料密切相关。采用实时荧光定量PCR技术,探究了纳米零价铁 (nZVI) 和纳米氧化锌 (nZnO) 对污水处理系统中磺胺类耐药菌 (ARB) 和ARGs的分布特征的影响。结果表明,50 mg·L−1 nZVI和nZnO暴露均有利于削减磺胺ARB浓度数。与对照组相比,nZVI和nZnO暴露后污泥中sul1丰度增加了0.25%~16.21%,而sul2出现明显削减 (1.51%~15.47%) 。此外,50 mg·L−1 nZVI会导致游离态胞外ARGs大幅削减。nZVI和nZnO暴露通过富集污水处理系统中的intI1、改变细胞膜通透性和调节细菌转录能力促进sul1的增殖。本研究结果阐明了典型纳米金属对污水处理系统中ARB和不同形态ARGs消长的影响及其机制,可为制定有效调控和全面削减污水处理系统中耐药污染策略提供参考。
-
关键词:
- 耐药基因(ARGs) /
- 耐药菌(ARB) /
- 纳米零价铁(nZVI) /
- 纳米氧化锌(nZnO) /
- 污水处理系统
Abstract: Antibiotic resistance and nanomaterials contaminants are important emerging environmental issues worldwide. Antibiotic resistance vectors—antibiotic resistance genes (ARGs) widely exist in wastewater treatment systems and have been proved to be closely related to metal nanoparticles. The influences of nano zero valent iron (nZVI) and nano zinc oxide (nZnO) on the distribution of antibiotic resistant bacteria (ARB) and different forms of ARGs in wastewater treatment system were investigated by quantitative real-time PCR. The results showed that 50 mg·L−1 nZVI and nZnO exposure was beneficial to the reduction of ARB concentration. Compared with the control group, the abundance of sul1 in the sludge after exposure to nZVI and nZnO increased by 0.25%~16.21%, while sul2 decreased significantly (1.51%~15.47%). In addition, 50 mg·L−1 nZVI led to a significant reduction in free extracellular ARGs. nZVI and nZnO exposure can promote the proliferation of sul1 by enriching intI1, changing the permeability of cell membrane and regulating the transcription ability of bacteria. The present study was helpful for clarifying the effects and mechanisms of metal nanoparticles on the fate of ARB and different forms of ARGs in the wastewater treatment system, which may lay the foundation for effective regulation and mitigation strategies of antibiotic resistance in the wastewater treatment system. -
-
-
[1] Science/AAAS Custom Publishing Office and Shanghai Jiao Tong University. 125 questions: Exploration and discovery [EB/OL]. [2021-05-14].https://www.science.org/content/resource/125-questions-exploration-and-discovery. [2] PEHRSSON E C, TSUKAYAMA P, PATEL S, et al. Interconnected microbiomes and resistomes in low-income human habitats[J]. Nature, 2016, 533(7602): 212-216. doi: 10.1038/nature17672 [3] O'NEILL J, Tackling drug-resistant infections globally: final report and recommendations [R]. 2016. O'Neill J. [4] PRUDEN A, PEI R, STORTEBOOM H, et al. Antibiotic resistance genes as emerging contaminants: studies in northern Colorado[J]. Environmental Science & Technology, 2006, 40(23): 7445-7450. [5] SANDERSON H, FRICKER C, BROWN R S, et al. Antibiotic resistance genes as an emerging environmental contaminant[J]. Environmental Reviews, 2016, 24(2): 205-218. doi: 10.1139/er-2015-0069 [6] UNEP, Frontiers 2017: Emerging Issues of Environmental Concern [R]. Kenya: United Nations Environment Programme, 2017. [7] 王荣昌, 王超颖, 曾旭. 污水处理过程中抗生素抗性基因的检测及其水平转移机制的研究进展[J]. 环境化学, 2017, 36(12): 2567-2573. doi: 10.7524/j.issn.0254-6108.2017042605 [8] ULUSEKER C, KASTER K M, THORSEN K, et al. A review on occurrence and spread of antibiotic resistance in wastewaters and in wastewater treatment plants: mechanisms and perspectives[J]. Frontiers in Microbiology, 2021, 12: 717809. doi: 10.3389/fmicb.2021.717809 [9] 李超, 鲁建江, 童延斌, 等. 喹诺酮抗性基因在城市污水处理系统中的分布及去除[J]. 环境工程学报, 2016, 10(3): 1177-1183. doi: 10.12030/j.cjee.20160328 [10] MILLER J H, NOVAK J T, KNOCKE W R, et al. Effect of silver nanoparticles and antibiotics on antibiotic resistance genes in anaerobic digestion[J]. Water Environment Research, 2013, 85(5): 411-421. doi: 10.2175/106143012X13373575831394 [11] MA Y, METCH J W, YANG Y, et al. Shift in antibiotic resistance gene profiles associated with nanosilver during wastewater treatment [J]. FEMS Microbiology Ecology, 2016, 92(3). [12] FU J J, HUANG D Q, LU Z Y, et al. Comparison of the dynamic responses of different anammox granules to copper nanoparticle stress: Antibiotic exposure history made a difference[J]. Bioresource Technology, 2021, 333: 125186. doi: 10.1016/j.biortech.2021.125186 [13] HUANG H, ZHENG X, YANG S, et al. More than sulfidation: Roles of biogenic sulfide in attenuating the impacts of CuO nanoparticle on antibiotic resistance genes during sludge anaerobic digestion[J]. Water Research, 2019, 158: 1-10. doi: 10.1016/j.watres.2019.04.019 [14] ZHANG Y, WANG L, XIONG Z, et al. Removal of antibiotic resistance genes from post-treated swine wastewater by mFe/nCu system[J]. Chemical Engineering Journal, 2020, 400: 125953. doi: 10.1016/j.cej.2020.125953 [15] ZHANG Y, XU R, XIANG Y, et al. Addition of nanoparticles increases the abundance of mobile genetic elements and changes microbial community in the sludge anaerobic digestion system[J]. Journal of Hazardous Materials, 2021, 405: 124206. doi: 10.1016/j.jhazmat.2020.124206 [16] LI J, GUO N, ZHAO S, et al. Mechanisms of metabolic performance enhancement and ARGs attenuation during nZVI-assisted anaerobic chloramphenicol wastewater treatment[J]. Journal of Hazardous Materials, 2021, 419: 126508. doi: 10.1016/j.jhazmat.2021.126508 [17] ZHANG Y, YANG Z, XIANG Y, et al. Evolutions of antibiotic resistance genes (ARGs), class 1 integron-integrase (intI1) and potential hosts of ARGs during sludge anaerobic digestion with the iron nanoparticles addition[J]. Science of the Total Environment, 2020, 724: 138248. doi: 10.1016/j.scitotenv.2020.138248 [18] ZAREI-BAYGI A, SMITH A L. Intracellular versus extracellular antibiotic resistance genes in the environment: Prevalence, horizontal transfer, and mitigation strategies[J]. Bioresource Technology, 2021, 319: 124181. doi: 10.1016/j.biortech.2020.124181 [19] ZHOU S, ZHU Y, YAN Y, et al. Deciphering extracellular antibiotic resistance genes (eARGs) in activated sludge by metagenome[J]. Water Research, 2019, 161: 610-620. doi: 10.1016/j.watres.2019.06.048 [20] MAO D, LUO Y, MATHIEU J, et al. Persistence of extracellular DNA in river sediment facilitates antibiotic resistance gene propagation[J]. Environmental Science & Technology, 2014, 48(1): 71-78. [21] DONG P, WANG H, FANG T, et al. Assessment of extracellular antibiotic resistance genes (eARGs) in typical environmental samples and the transforming ability of eARG[J]. Environment International, 2019, 125: 90-96. doi: 10.1016/j.envint.2019.01.050 [22] DOMINIAK D M, NIELSEN J L, NIELSEN P H. Extracellular DNA is abundant and important for microcolony strength in mixed microbial biofilms[J]. Environmental Microbiology, 2011, 13(3): 710-721. doi: 10.1111/j.1462-2920.2010.02375.x [23] ZHENG X, WU R, CHEN Y. Effects of ZnO nanoparticles on wastewater biological nitrogen and phosphorus removal[J]. Environmental Science & Technology, 2011, 45(7): 2826-2832. [24] ZHOU L, ZHUANG W, WANG X, et al. Potential effects of loading nano zero valent iron discharged on membrane fouling in an anoxic/oxic membrane bioreactor[J]. Water Research, 2017, 111: 140-146. doi: 10.1016/j.watres.2017.01.007 [25] CHEN P J, TAN S W, WU W L. Stabilization or oxidation of nanoscale zerovalent iron at environmentally relevant exposure changes bioavailability and toxicity in medaka fish[J]. Environmental Science & Technology, 2012, 46(15): 8431-8439. [26] WIEGAND I, HILPERT K, HANCOCK R E W. Agar and broth dilution methods to determine the minimal inhibitory concentration (MIC) of antimicrobial substances[J]. Nature Protocols, 2008, 3(2): 163-175. doi: 10.1038/nprot.2007.521 [27] PATEL J B, COCKERILL F, BRADFORD P, et al. Clinical and laboratory standards institute[J]. Performance standards for Antimicrobial Susceptibility Testing, 2017: 42-45. [28] PALMGREN R, NIELSEN P H. Accumulation of DNA in the exopolymeric matrix of activated sludge and bacterial cultures[J]. Water Science and Technology, 1996, 34(5-6): 233-240. doi: 10.2166/wst.1996.0555 [29] ZHOU J, BRUNS M A, TIEDJE J M. DNA recovery from soils of diverse composition[J]. Applied and Environmental Microbiology, 1996, 62(2): 316-322. doi: 10.1128/aem.62.2.316-322.1996 [30] CORINALDESI C, DANOVARO R, DELL'ANNO A. Simultaneous recovery of extracellular and intracellular DNA suitable for molecular studies from marine sediments[J]. Applied and Environmental Microbiology, 2005, 71(1): 46-50. doi: 10.1128/AEM.71.1.46-50.2005 [31] 萨姆布鲁克, 分子克隆实验指南: 下册 [M]. 第三版. 北京: 科学出版社, 2002. [32] ZHANG Y, NIU Z, ZHANG Y, et al. Occurrence of intracellular and extracellular antibiotic resistance genes in coastal areas of Bohai Bay (China) and the factors affecting them[J]. Environmental Pollution, 2018, 236: 126-136. doi: 10.1016/j.envpol.2018.01.033 [33] GREEN M R, SAMBROOK J. Precipitation of DNA with ethanol[J]. Cold Spring Harbor Protocols, 2016, 2016(12): pdb.prot093377. doi: 10.1101/pdb.prot093377 [34] PEI R, KIM S C, CARLSON K H, et al. Effect of river landscape on the sediment concentrations of antibiotics and corresponding antibiotic resistance genes (ARG)[J]. Water research, 2006, 40(12): 2427-2435. doi: 10.1016/j.watres.2006.04.017 [35] LUO Y I, MAO D, RYSZ M, et al. Trends in antibiotic resistance genes occurrence in the Haihe River, China[J]. Environmental Science & Technology, 2010, 44(19): 7220-7225. [36] FIERER N, JACKSON J A, VILGALYS R, et al. Assessment of soil microbial community structure by use of taxon-specific quantitative PCR assays[J]. Applied and environmental microbiology, 2005, 71(7): 4117-4120. doi: 10.1128/AEM.71.7.4117-4120.2005 [37] SHAHBAZI S, SHIVAEE A, NASIRI M, et al. Zinc oxide nanoparticles impact the expression of the genes involved in toxin-antitoxin systems in multidrug-resistant Acinetobacter baumannii[J]. Journal of Basic Microbiology, 2022, 11: 1. [38] ABDELGHAFAR A, YOUSEF N, ASKOURA M. Zinc oxide nanoparticles reduce biofilm formation, synergize antibiotics action and attenuate Staphylococcus aureus virulence in host; an important message to clinicians[J]. BMC Microbiology, 2022, 22(1): 1-17. doi: 10.1186/s12866-021-02409-6 [39] QIU X, ZHOU G, WANG H. Nanoscale zero-valent iron inhibits the horizontal gene transfer of antibiotic resistance genes in chicken manure compost[J]. Journal of Hazardous Materials, 2022, 422: 126883. doi: 10.1016/j.jhazmat.2021.126883 [40] 陆贤, 郭美婷, 张伟贤. 纳米零价铁对耐四环素菌耐药特性的影响[J]. 中国环境科学, 2017, 37(1): 381-385. doi: 10.3969/j.issn.1000-6923.2017.01.047 [41] WANG P, CHEN X, LIANG X, et al. Effects of nanoscale zero-valent iron on the performance and the fate of antibiotic resistance genes during thermophilic and mesophilic anaerobic digestion of food waste[J]. Bioresource Technology, 2019, 293: 122092. doi: 10.1016/j.biortech.2019.122092 [42] WANG Q, GU J, WANG X, et al. Effects of nano-zerovalent iron on antibiotic resistance genes and mobile genetic elements during swine manure composting[J]. Environmental Pollution, 2020, 258: 113654. doi: 10.1016/j.envpol.2019.113654 [43] HUANG H, CHEN Y, YANG S, et al. CuO and ZnO nanoparticles drive the propagation of antibiotic resistance genes during sludge anaerobic digestion: possible role of stimulated signal transduction[J]. Environmental Science:Nano, 2019, 6(2): 528-539. doi: 10.1039/C8EN00370J [44] 陈芋如. 纳米颗粒物对河口水环境中微生物群落及抗生素抗性基因的影响 [D]. 上海: 华东师范大学, 2020. [45] CHEN Y, GUO X, FENG J, et al. Impact of ZnO nanoparticles on the antibiotic resistance genes (ARGs) in estuarine water: ARG variations and their association with the microbial community[J]. Environmental Science:Nano, 2019, 6(8): 2405-2419. doi: 10.1039/C9EN00338J [46] NG C, TAY M, TAN B, et al. Characterization of metagenomes in urban aquatic compartments reveals high prevalence of clinically relevant antibiotic resistance genes in wastewaters[J]. Frontiers in Microbiology, 2017, 8: 2200. doi: 10.3389/fmicb.2017.02200 [47] DAR D, SHAMIR M, MELLIN J R, et al. Term-seq reveals abundant ribo-regulation of antibiotics resistance in bacteria[J]. Science, 2016, 352(6282): aad9822. doi: 10.1126/science.aad9822 [48] LIU Z, KLUMPER U, LIU Y, et al. Metagenomic and metatranscriptomic analyses reveal activity and hosts of antibiotic resistance genes in activated sludge[J]. Environment International, 2019, 129: 208-220. doi: 10.1016/j.envint.2019.05.036 -


 下载:
下载: