有害藻华(harmful algal blooms, HABs)是指因微藻等生物大量繁殖,导致水产品死亡,破坏生态系统功能,威胁人类健康和生命安全的一种环境异常生态现象[1-4],其频发与人类活动导致的海水富营养化、全球气候变化等问题相关[5-7]。米氏凯伦藻(Karenia mikimotoi)藻华是一种对海水养殖业危害严重的有害藻华,广泛分布于亚洲、欧洲、美洲与非洲的近岸海域[2]。该藻藻华同样是我国近岸主要有害藻华之一[8],自1998年在香港近岸暴发导致大量养殖鱼类死亡以来[9],常年在我国渤海、东海等多地暴发,经济损失以数十亿元计[10],其中仅2012年福建近岸因其导致的鲍鱼养殖业的损失就高达20.1亿元[11]。
高密度的米氏凯伦藻不仅能够导致养殖鱼类与贝类大量死亡,对其他海洋生物也有显著的不利影响[12],如降低甲壳类的运动能力[13]、抑制中华哲水蚤摄食[14]、导致海胆、海星[15]等多种生物死亡。该藻能破坏鱼类与贝类的鳃,使其出现明显的鳃上皮细胞水肿、充血、脱落和皱缩等组织病变现象[16-17],从而影响其呼吸和渗透压调节。米氏凯伦藻还能显著降低贝类淋巴细胞的黏着性与吞噬能力[18],抑制细胞增殖,使鱼类胚胎滞长、畸变[19],影响斑马鱼胆碱能和神经系统的正常发育[20],表现出明显的免疫毒性和细胞毒性。米氏凯伦藻对多种生物的抗氧化、生殖与内分泌系统也有显著的不利影响[18, 20-22]。但该藻对海洋生物分子水平的影响效应及相应机制的探究存在缺失。
转录组测序(RNA-Seq)能够全面快速地获得某一物种特定组织或器官在特定状态下的所能转录出来的所有RNA的总和,以其高通量测序能力、高灵敏度、宽范围、用时短、高效率、高重复性和低成本特点而被广泛应用[23-24]。该技术同样被广泛应用于有害藻毒性效应与机制的研究中,如微囊藻毒素-LR(MC-LR)能通过TLR/MyD88介导的信号通路引发斑马鱼慢性炎症反应[25],并且通过补体和凝血级联途径导致草鱼免疫功能受损[26]。而海水青鳉(Oryzias melastigma)因其个体小、性别易区分、繁殖周期短、产卵率高,可在实验室条件下大规模饲养等优点,已成为我国海洋生态毒理学研究的模式生物[27-29]。Qiao等[30]通过海水青鳉的综合组学分析,发现MC-LR能够影响能量产生、蛋白质生物合成和脂质代谢途径,导致生殖障碍。故本文选取海水青鳉为研究对象,借助RNA-Seq和生物信息学分析方法,探究米氏凯伦藻对海水青鳉分子转录水平的影响。
1 材料与方法(Materials and methods)
1.1 实验生物培养
米氏凯伦藻和对照用小球藻(Chlorella sp.)均由中国科学院海洋研究所藻种中心提供,福建农林大学海洋研究院重点实验室连续培养,所有藻类均使用f/2海水培养基[31]纯种培养,培养/实验环境为温度(19±1)℃,光照强度(2 650±100) lx,光暗比14 h∶10 h。海水青鳉由闽江学院提供,循环水系统连续培养至5月龄成鱼,养殖期间水温22~28℃,光暗比12 h∶12 h。仔鱼到成鱼各阶段每天9:00和17:00分别投喂轮虫、卤虫和优质饲料2次,每周更换新鲜的循环海水。用于藻类培养和实验鱼养殖的海水购自福建省海水公司,藻类培养和实验用海水均经0.45 μm混合纤维滤膜过滤后高温高压消毒后备用。
1.2 实验处理及样品采集
指数生长期的米氏凯伦藻在光学显微镜下记录藻密度,用无菌海水稀释至终密度2×104cells⋅mL-1到500 mL玻璃烧杯中(实验体积400 mL),对照组添加与实验组等生物量的小球藻,空白组添加与实验组等量的无菌海水[32]。大小均一,状态健康的海水青鳉成鱼提前移至实验环境适应24 h后随机挑到各组中,每组3个平行,每平行10条鱼。通过调节曝气保持各组溶解氧水平相当。实验进行96 h,每24 h喂食并更换新鲜藻液与无菌海水。取样前24 h停止喂食,0.02%MS-222溶液将海水青鳉麻醉后迅速完整分离鳃(GI)和肝脏(LI)组织于无酶冻存管中,液氮速冻后-80℃保存。
1.3 转录组测序与分析
Trizol(Invitrogen,美国)法提取组织Total RNA,1%琼脂糖凝胶电泳检测RNA质量,NanoDrop 2000检测RNA浓度和纯度,RNA 6000 Nano Kit检测RNA完整性。反转录构建cDNA文库后用Illumina HiSeq 2000进行测序(上海美吉生物医药科技有限公司)。测序原始样本(raw reads)使用软件Fastp质控后得到高质量的质控数据(clean reads)。使用软件TopHat2进行序列比对(来源:https://www.ncbi.nlm.nih.gov/genome/?term=txid30732[orgn];参考基因来源:Oryzias_melastigma;参考基因组版本:GCF_002922805.1)获得匹配数据(mapped reads),RSeQC-2.3.6整体质量评估后使用拼接软件String Tie进行转录组组装。
利用软件RSEM定量分析基因和转录本的表达量,DESeq2分析鉴定样本间的差异表达基因(differentially expressed genes, DEGs)(差异条件:log2 fold changes≥1且false discovery rate(FDR)<0.05),并用Benjamini and Hochberg(BH)方法对差异检验P-value进行多重检验矫正后构建DEGs基因集。
利用Gene ontology (GO)和Kyoto encyclopedia of genes and genomes (KEGG)数据库进行功能分类。用Fisher精确检验方法,使用Goatools软件和KOBAS软件分别进行GO和KEGG富集分析。并进行BH多重检验,当校正的P<0.05、P<0.01视为显著。
1.4 实时荧光定量PCR(quantitative real-time PCR,qPCR)验证
从测序结果中筛选出12个差异表达基因(serpine1、hsp90b1、bmp10、plat、sugt1、bcl2l1、ddit、nr4a1、dusp1、f2、f5、tbxas1、plg)进行qPCR验证(引物序列见表1)。通过测序提取的Total RNA使用反转录试剂盒(Promega,美国)合成cDNA,经纯化、末端修复、连接接头后进行PCR扩增,得到cDNA文库,qPCR反应程序为95℃预变性150 s;95℃15 s,60℃20 s,72℃20s,40个循环。以18s为内参基因,采用2-ΔΔCt[33]方法分析结果,使用SPSS软件进行统计学分析。
表1 荧光定量PCR引物序列
Table 1 Primer sequences for qPCR
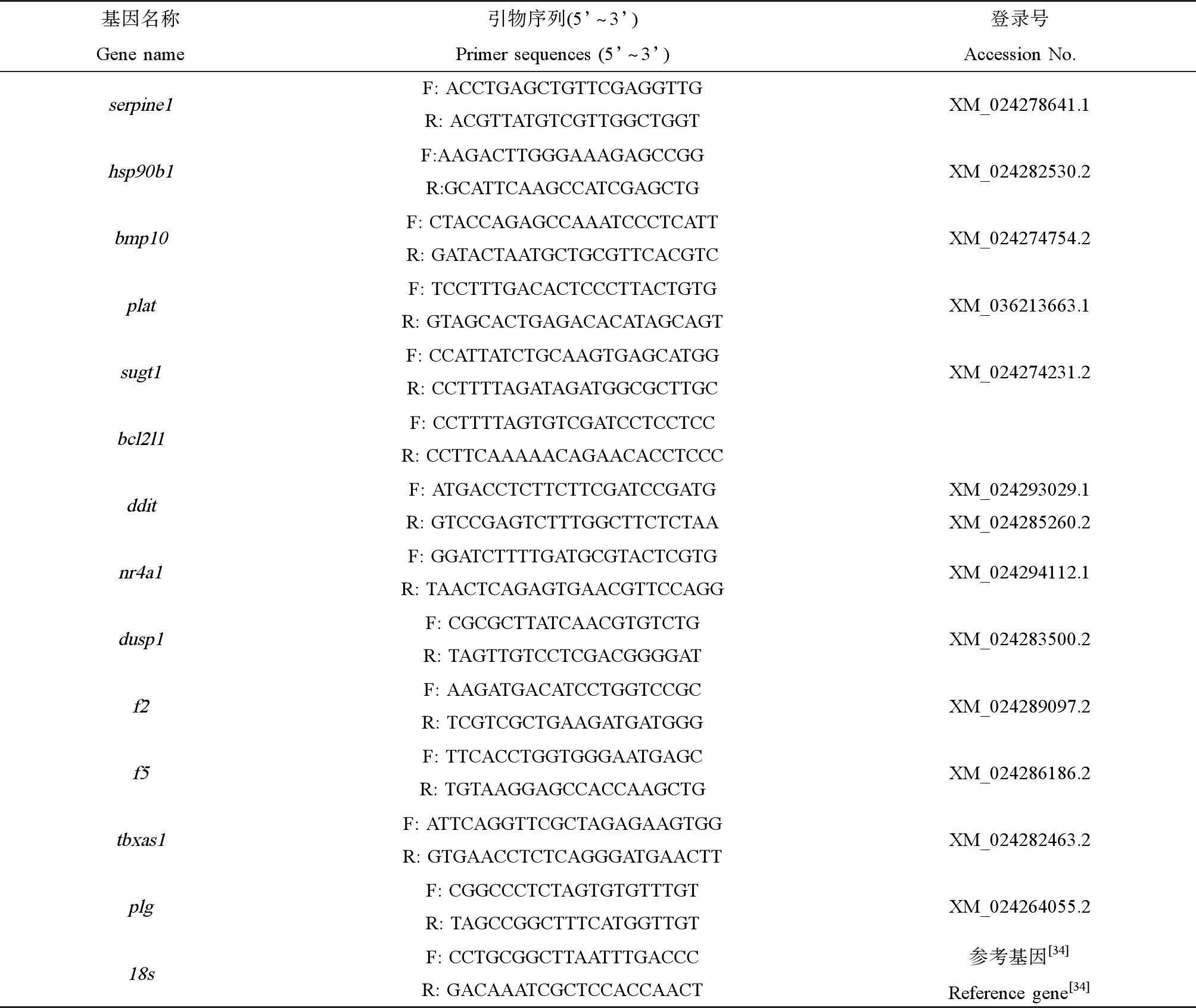
基因名称引物序列(5’^3’)登录号Gene namePrimer sequences (5’^3’)Accession No.serpine1F: ACCTGAGCTGTTCGAGGTTGR: ACGTTATGTCGTTGGCTGGTXM_024278641.1hsp90b1F:AAGACTTGGGAAAGAGCCGGR:GCATTCAAGCCATCGAGCTGXM_024282530.2bmp10F: CTACCAGAGCCAAATCCCTCATTR: GATACTAATGCTGCGTTCACGTCXM_024274754.2platF: TCCTTTGACACTCCCTTACTGTGR: GTAGCACTGAGACACATAGCAGTXM_036213663.1sugt1F: CCATTATCTGCAAGTGAGCATGGR: CCTTTTAGATAGATGGCGCTTGCXM_024274231.2bcl2l1F: CCTTTTAGTGTCGATCCTCCTCCR: CCTTCAAAAACAGAACACCTCCCdditF: ATGACCTCTTCTTCGATCCGATGR: GTCCGAGTCTTTGGCTTCTCTAAXM_024293029.1XM_024285260.2nr4a1F: GGATCTTTTGATGCGTACTCGTGR: TAACTCAGAGTGAACGTTCCAGGXM_024294112.1dusp1F: CGCGCTTATCAACGTGTCTGR: TAGTTGTCCTCGACGGGGATXM_024283500.2f2F: AAGATGACATCCTGGTCCGCR: TCGTCGCTGAAGATGATGGGXM_024289097.2f5F: TTCACCTGGTGGGAATGAGCR: TGTAAGGAGCCACCAAGCTGXM_024286186.2tbxas1F: ATTCAGGTTCGCTAGAGAAGTGGR: GTGAACCTCTCAGGGATGAACTTXM_024282463.2plgF: CGGCCCTCTAGTGTGTTTGTR: TAGCCGGCTTTCATGGTTGTXM_024264055.218sF: CCTGCGGCTTAATTTGACCCR: GACAAATCGCTCCACCAACT参考基因[34]Reference gene[34]
2 结果(Results)
2.1 RNA-Seq数据质量评估及序列比对分析
本实验构建了暴露96 h后海水青鳉鳃和肝脏组织共12个样本的转录组文库,各样本的clean reads均在4 389万条以上,质控数据的测序碱基Error rate均<0.0241%,Q20碱基百分比在98.33%以上,Q30碱基百分比在95.07%以上,说明测序结果质量较好;其中,2种鱼组织对照组(GI_C1、GI_C2、LI_C1、LI_C2)、空白组(GI_B1、GI_B2、LI_B1、LI_B2)和实验组(GI_T1、GI_T2、LI_T1、LI_T2)能成功比对到海水青鳉基因组的clean reads比例分别在92.37%~94.96%之间,说明参考基因组的选择合理;有85.47%~90.33%之间比例的clean reads能成功比对到CDS参考序列上,说明测序深度足以覆盖转录组;样本间相关性分析结果表明同组织组内的相关性系数均>0.8;在样本间PCA分析时(图1),同组织组内的样本都聚集于同一象限中,说明组内重复性较强,组间差异较大,可进行下一步分析。
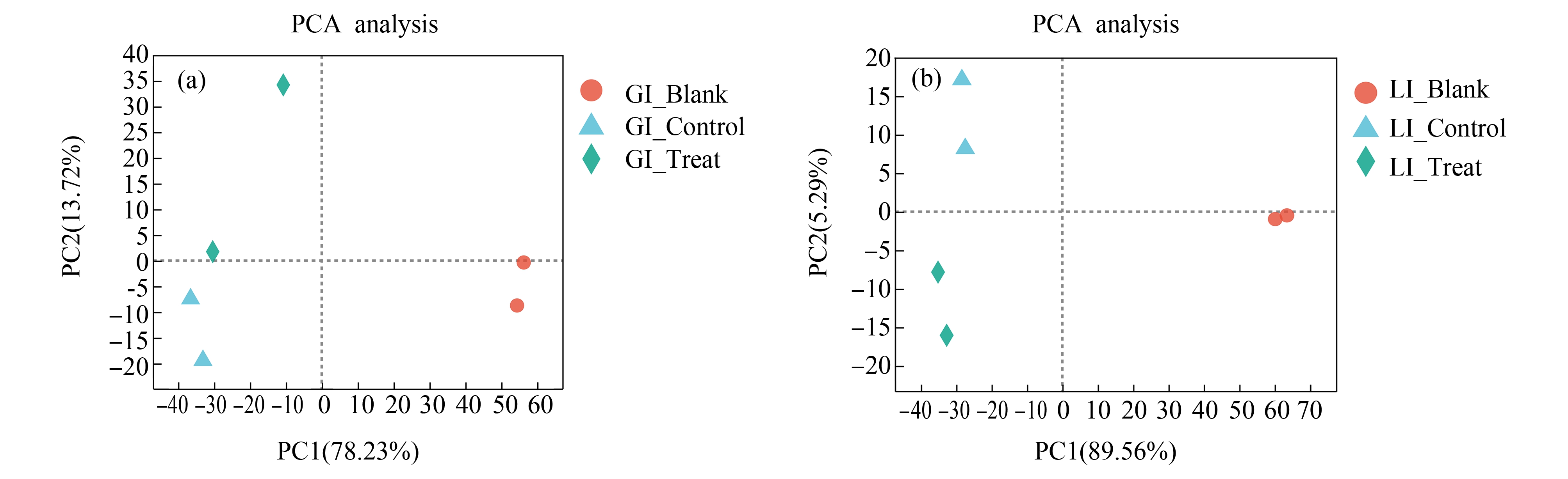
图1 样本间PCA分析
注:(a)鳃;(b)肝脏。
Fig. 1 PCA analysis between samples
Note: (a) Gill; (b) Liver.
2.2 差异表达基因(DEGs)
如图2(a)所示,海水青鳉肝脏实验组与对照组相比,共有604个差异基因,390个上调基因,214个下调基因;空白组与对照组相比,共有10 662个差异基因,4 766个上调基因,5 896个下调基因。鳃实验组与对照组相比,共有508个差异基因,184个上调基因,324个下调基因;空白组与对照组相比,共有6 060个差异基因,3 454个上调基因,2 606个下调基因。不同组间DEGs上下调的分布情况如图2(b)、2(c)、2(d)、2(e)。

图2 表达量差异分析
注:(a)各处理组差异表达基因(DEGs)统计;(b)鳃对照组与实验组DEGs火山分布图;(c)肝脏对照组与实验组DEGs火山分布图;(d)鳃空白组与实验组DEGs火山分布图;(e)肝脏空白组与实验组DEGs火山分布图。
Fig. 2 Expression difference analysis
Note: (a) Differentially expressed genes (DEGs) statistics of each treatment group; (b) DEGs volcanic distribution among gill control and experimental group; (c) DEGs volcanic distribution among liver control and experimental group; (d) DEGs volcanic distribution among gill blank and experimental group; (e) DEGs volcanic distribution among liver blank and experimental group.
2.3 DEGs的GO功能分类和富集分析
如图3所示,在GO功能注释中,DEGs可分为分子功能(molecular function, MF)、生物过程(biological process, BP)和细胞组成(cellular component, CC)三大类。鳃对照与实验组相比,508个DEGs中209个GO terms得以注释,78个GO terms存在显著富集(P<0.01),其中包括53个BP类,21个分子功能(molecular function, MF)类和4个CC类。肝脏对照与实验组相比604个DEGs中279个GO terms得以注释,123个GO terms存在显著富集(P<0.01),其中包括89个BP类,27个MF类,7个CC类。在图3中2种鱼组织存在显著富集的20个GO terms在CC类中共同影响最大的是细胞膜部分(membrane part)和细胞部分(cell part);在MF类中共同影响最大的是催化活性(catalytic activity)和结合(binding);而在BP类中,共同影响较大的是细胞进程(cellular process),其中鳃还显著影响生物调节(biological regulation),肝脏显著影响的是代谢进程(metabolic process)。此外,DEGs的GO在肝脏中还显著富集于氧运输(oxygen transport)、氧结合(oxygen binding)和氧载体活性(oxygen carrier activity);鳃在凝血酶激活受体信号通路(thrombin-activated receptor signaling pathway)、凝血酶激活的受体活性(thrombin-activated receptor activity)和离子跨膜运输(anion transmembrane transport)也存在显著富集。
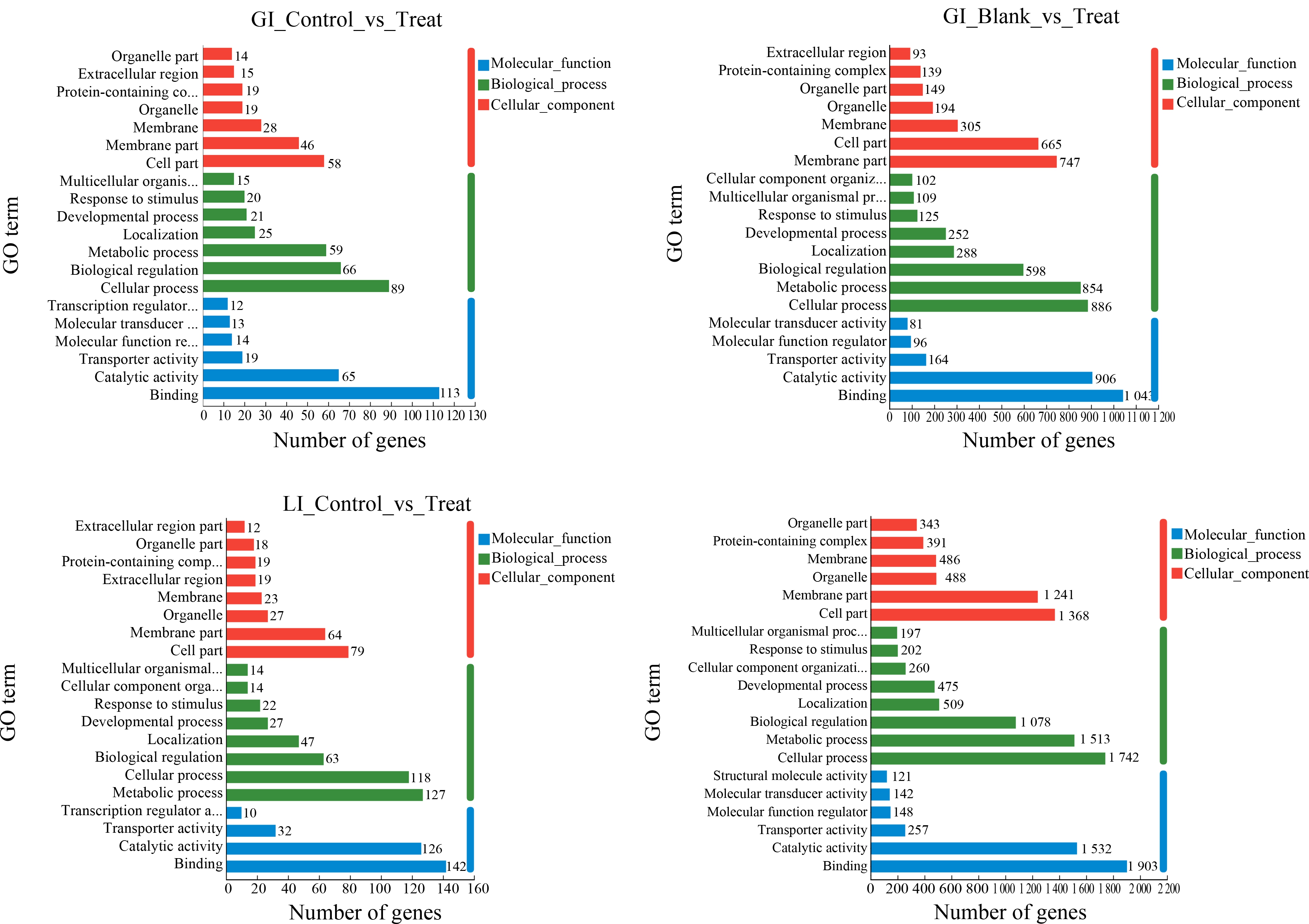
图3 各基因集DEGs分布情况
注:(a)鳃对照组与实验组;(b)鳃空白组与实验组;(c)肝脏对照组与实验组;(d)肝脏空白组与实验组。
Fig. 3 Distribution of DEGs in each gene set
Note: (a) Gill control and experimental group; (b) Gill blank and experimental group; (c) Liver control and experimental group; (d) Liver blank and experimental group.
2.4 DEGs的KEGG信号通路及富集分析
在鳃对照组与实验组的所有DEGs进行KEGG富集分析发现有374个信号通路得以注释,其中24个信号通路存在显著富集(P<0.05),且7个均与免疫系统和信号分子转导通路有关(图4(a)),同样在图4(b)对照组与实验组中也发现此信号通路,包括补体和凝血级联反应(complement and coagulation cascades)、白介素-17(IL-17 signaling pathway)、白细胞跨内皮迁移(leukocyte transendothelial migration)、细胞因子相互作用(cytokine-cytokine receptor interaction)、病毒蛋白与细胞因子和细胞因子受体的相互作用(viral protein interaction with cytokine and cytokine receptor)、低氧诱导因子信号通路(HIF-1 signaling pathway)和肿瘤坏死因子(TNF signaling pathway)信号通路。在肝脏中,有475个信号通路得以注释,其中45个信号通路存在显著富集(P<0.05),受到影响更强的是免疫系统的补体和凝血级联反应(complement and coagulation cascades)和代谢相关的通路(图4(c)、4(d))。同时,为了更全面了解米氏凯伦藻对海水青鳉的免疫毒性效应,本研究获得了免疫系统与信号分子转导信号通路的DEGs信息,并对部分DEGs进行了验证。
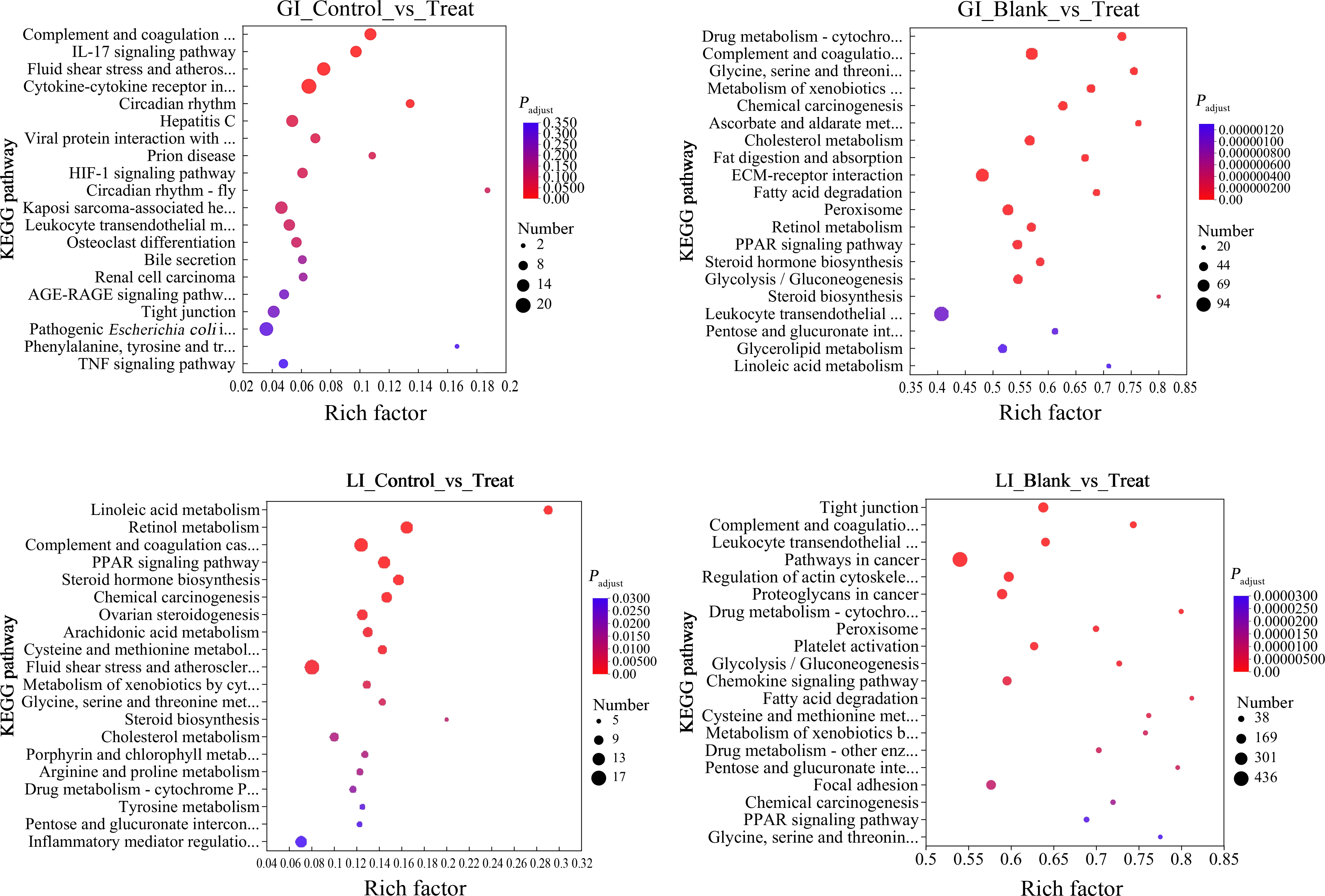
图4 KEGG富集分析
注:(a)鳃对照组与实验组;(b)鳃空白组与实验组;(c)肝脏对照组与实验组;(d)肝脏空白组与实验组。
Fig. 4 KEGG enrichment analysis
Note: (a) Gill control and experimental group; (b) Gill blank and experimental group; (c) Liver control and experimental group; (d) Liver blank and experimental group.
2.5 荧光定量PCR验证RNA-Seq数据
通过qPCR验证部分DEGs显示(图5),米氏凯伦藻刺激海水青鳉96 h后,鳃中serpine1、hsp90b1、bmp10、plat、sugt1、bcl2l1、ddit、dusp1基因表达水平显著下调,肝脏中f2、f5、tbxas1、plg基因表达水平显著上调,这些基因的表达量变化和RNA-Seq数据表达变化一致。

图5 荧光定量PCR验证部分DEGs
Fig. 5 qPCR validation of partial DEGs
3 讨论(Discussion)
米氏凯伦藻是一种典型的鱼毒性赤潮藻肇事种,有研究表明其藻华降解时产生的缺氧环境与氨氮不是造成鱼贝类大量死亡的主要原因[35-36];该藻脂溶性提取物具有较强的溶血毒性和细胞毒性,但该提取物较高浓度下也不能显著降低褶皱臂尾轮虫和皱纹盘鲍的存活率[2, 12];只有完整存活的藻细胞近距离接触海洋生物时,才会对其产生明显的毒性作用[2]。因此,本实验将与米氏凯伦藻直接接触的鳃组织和解毒相关重要器官肝脏作为研究对象进行转录组测序,结果表明该藻对其基因转录水平有着显著的影响,2种组织受藻影响存在一定的差异。KEGG富集结果表明DEGs在鳃中主要集中于免疫系统和和信号分子转导相关通路,比如补体和凝血级联反应、IL-17信号通路、白细胞跨内皮迁移、细胞因子相互作用、低氧诱导因子和肿瘤坏死因子信号通路,在Zhang等[16]研究中同样发现,米氏凯伦藻能激活海水青鳉鳃的免疫反应并增强其信号分子转导通路;而肝脏则显著富集于内分泌系统、脂质和氨基酸代谢相关通路。这可能是因为鳃参与气体交换、进行渗透压和酸碱调节,其不但是鱼类最主要的呼吸器官,更是接触水中有毒物质最直接的器官[37],这才使得米氏凯伦藻能够直接导致鳃应激;而肝脏在脊椎和水生动物蛋白质合成、脂肪代谢、能量生成和药物解毒发挥着极其重要的功能,两者功能的不同导致受藻影响的主要通路也存在一定的区别。与此同时,鱼鳃受藻影响导致的呼吸功能与免疫功能可能同样对肝脏的代谢产生一定的影响。
米氏凯伦藻对青鳉鳃和肝脏中凝血和补体级联反应相关通路都有显著影响,这和MC-LR处理草鱼中显著富集凝血与补体级联反应的免疫差异基因较多的结果为相似[26]。凝血系统可将入侵的微生物凝结在血液凝块中,以增加血管的渗透性和作为吞噬细胞的趋化剂而促进天然免疫系统防御[38];而补体系统在先天免疫和获得性免疫发挥着重要作用,是一种抗微生物防御,免疫调节及介导免疫病理的损伤性反应[39-40]。凝血过程可分为凝血酶原激活物、凝血酶和纤维蛋白的形成3个阶段,而凝血酶激活物可由2种不同的途径激活:一是由组织损伤而引起的外源性途径,二是与血管损伤相联系的内源性途径。在本研究中(图6),由鳃释放的因子Ⅲ(F3)进入血液与有活性的因子Ⅷ a(F7a)结合启动凝血过程,这表明米氏凯伦藻对海水青鳉鳃造成了一定的组织损伤,从而激活外源性途径。肝脏中的活化型因子Xa(F10a)与凝血因子V(F5)共同作用,形成活化的凝血酶Ⅱa(F2a),引起纤维蛋白原的降解,启动纤溶系统(图7)。纤溶系统在维持血液流动性和血管完整性方面起着重要作用。其包括纤溶酶原(plasminogen,Plg)的激活和纤维蛋白原(fibrinogen,Fg)的降解[41]。Plg的激活是纤溶反应的关键,在血液凝固过程中会激活细胞粘附、迁移、增殖通路。在肝脏实验组中(图7),凝血酶F2和Fg明显被激活,Plg的转录水平也显著上调,纤维蛋白形成血凝块,这说明米氏凯伦藻可能导致海水青鳉组织溶血,而被激活的肝脏凝血级联反应,能防止机体过量溶血并阻止病原体的入侵。
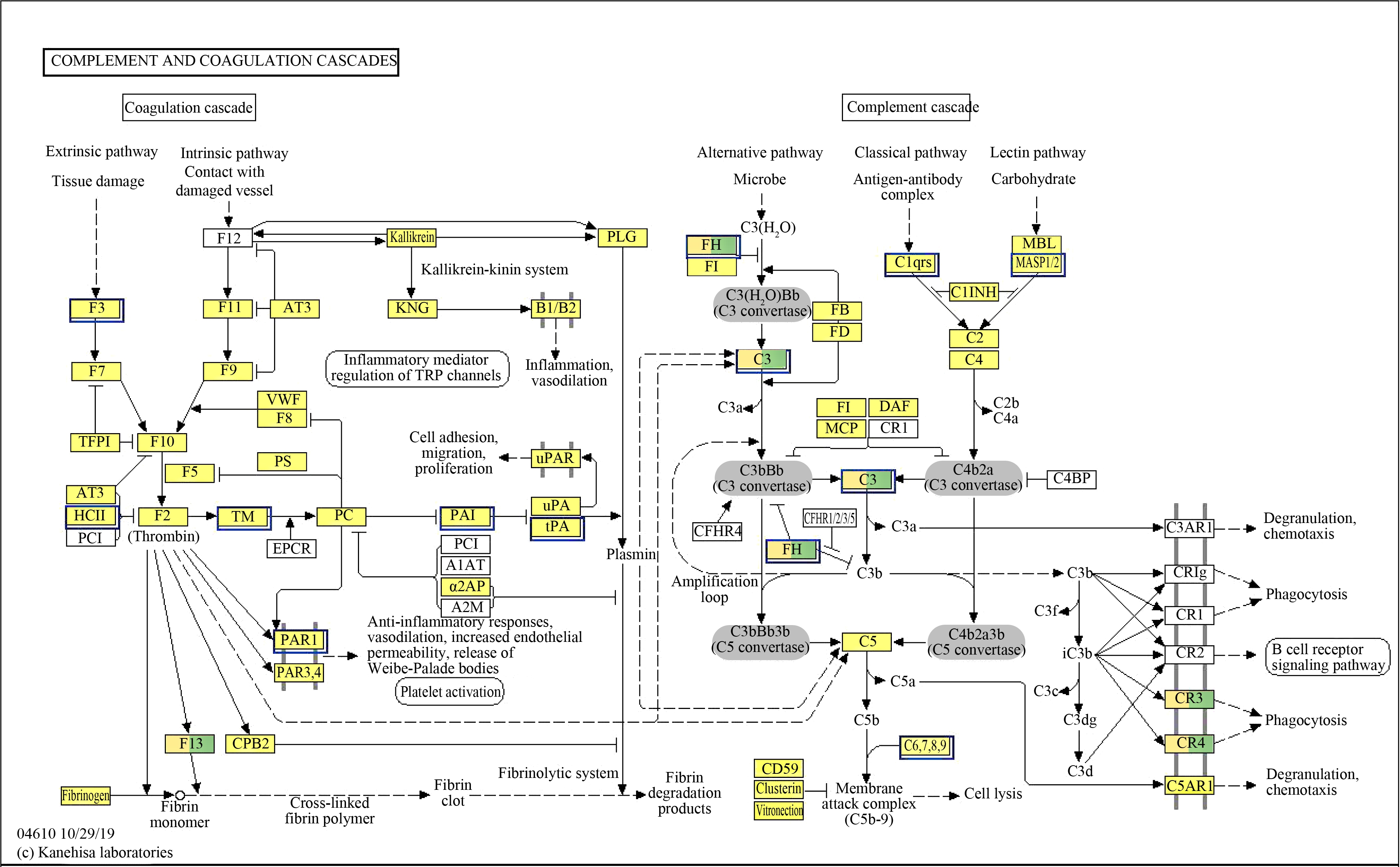

图6 鳃补体和凝血级联反应
注:(a) 鳃对照组与实验组;(b) 鳃空白组与实验组;蓝色方框代表基因/转录本下调;红色方框代表基因/转录本上调。
Fig. 6 Complement and coagulation cascade in gill
Note: (a) Gill control and experimental group; (b) Gill blank and experimental group; blue box represents gene/transcript down-regulation; red box represents gene/transcript up-regulation.
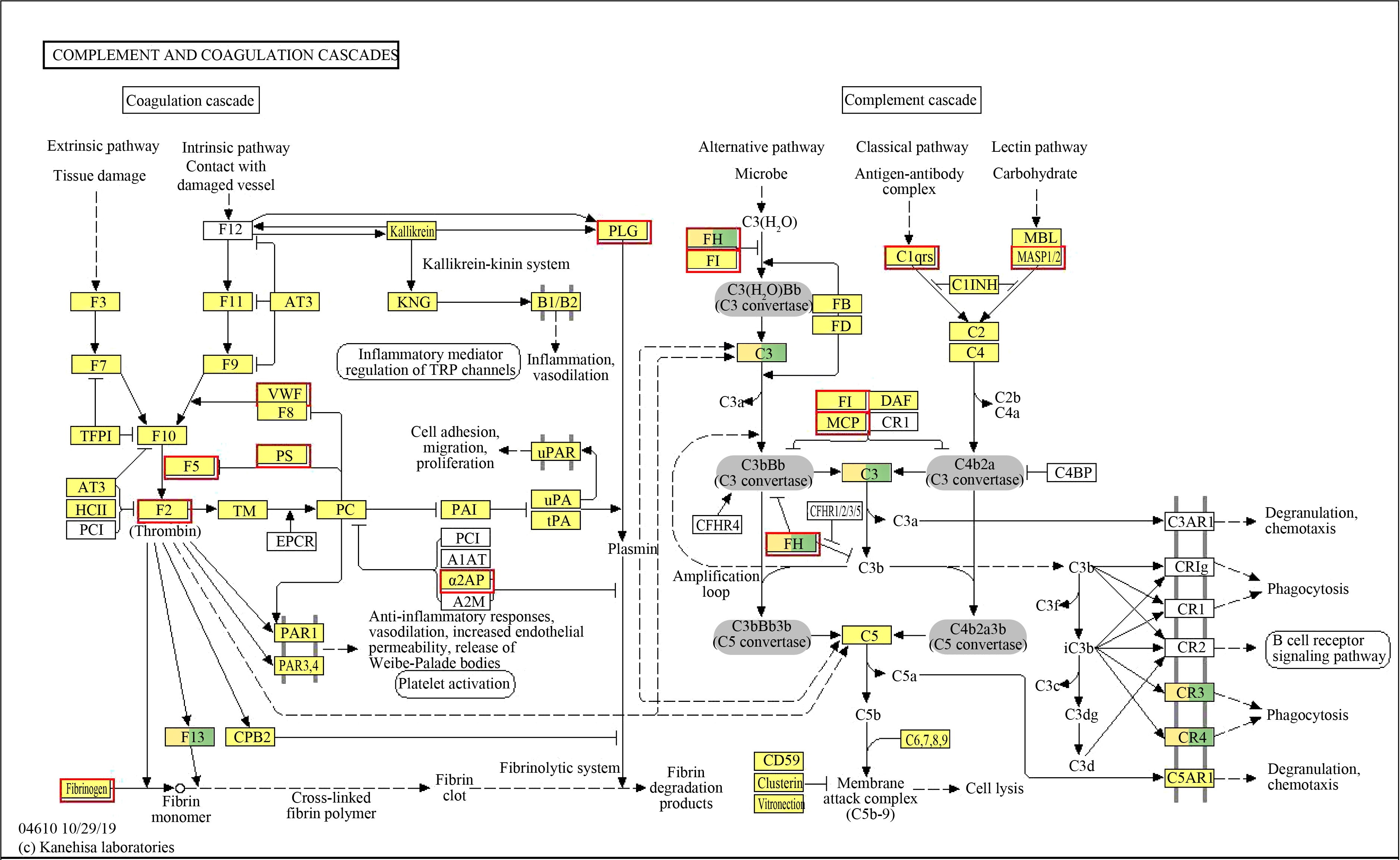
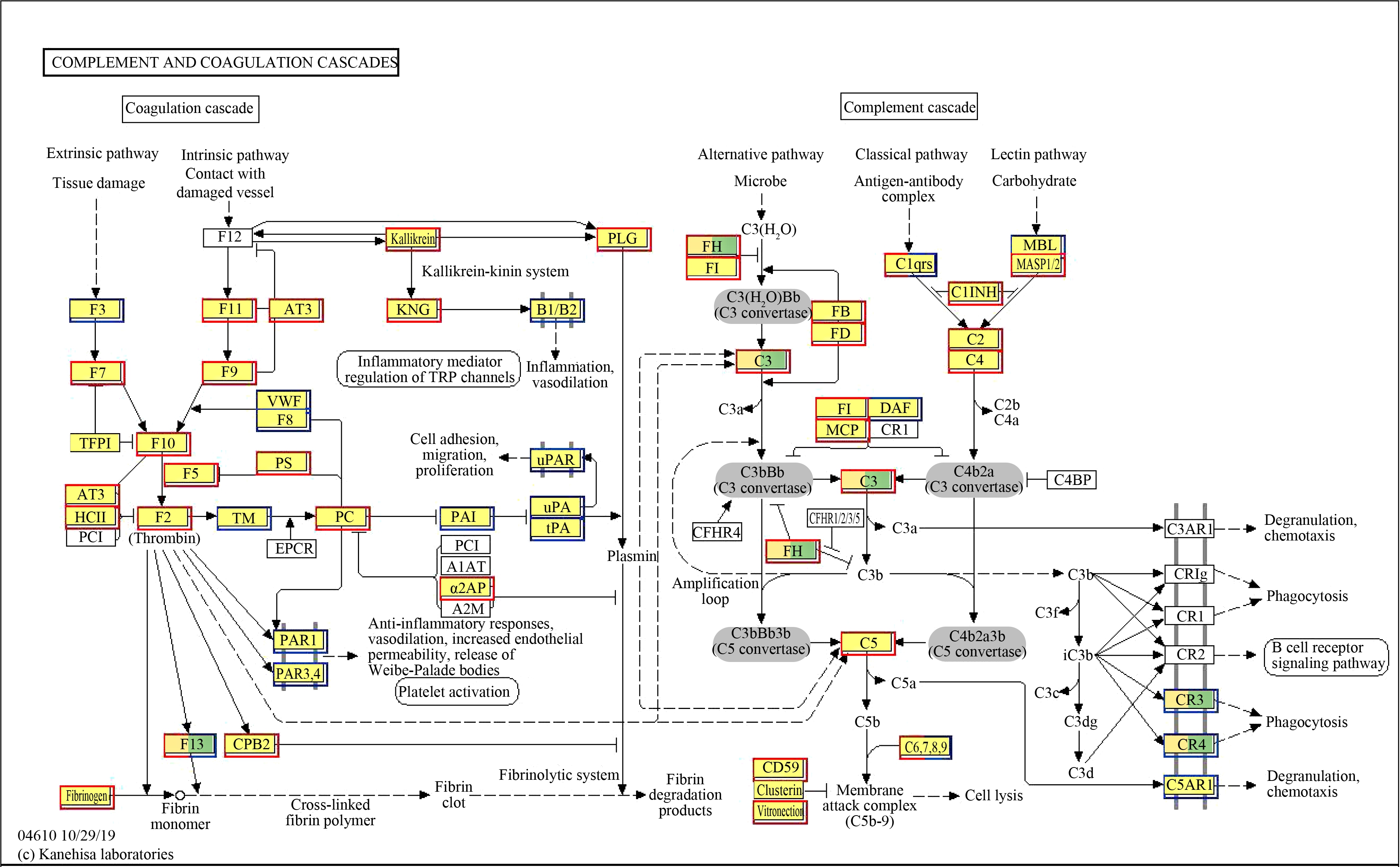
图7 肝脏补体和凝血级联反应
注:(a) 肝脏对照组与实验组;(b) 肝脏空白组与实验组;蓝色方框代表基因/转录本下调;红色方框代表基因/转录本上调。
Fig. 7 Complement and coagulation cascade in liver
Note: (a) Liver control and experimental group; (b) Liver blank and experimental group;blue box represents gene/transcript down-regulation; red box represents gene/transcript up-regulation.
补体系统可通过3种不同的方式激活:一是经典途径(CP);二是旁路途径(AP);三是凝集素途径(MP)。由图6可知,在本实验中主要受到影响的是AP,水解的C3受到因子FH和FI复合物的抑制作用形成C3转化酶,继而激活了C3,引发了酶促级联反应。在膜攻击阶段时,C5转化酶裂解C5产生具有过敏毒素和趋化活性的C5a及易吸附于邻近的细胞表面的C5b,C5b与下调的C6、C7、C8、C9结合形成膜攻击复合物,深入穿透病原体膜,引起细胞膜损伤[42]。补体C3在补体系统中含量最高,在补体系统激活中也起关键作用,而本研究中鳃能明显激活C3,引发细胞溶解,这可能是由于米氏凯伦藻对海水青鳉造成的鳃外源性组织损伤而引起鳃细胞固缩、溶解,进而可能使细胞坏死及凋亡。
米氏凯伦藻对鳃IL-17相关通路有着显著的影响,其下游相关信号通路包括细胞因子相互作用、核因子NF-κB和丝裂原活化蛋白激酶(MAPK)信号通路均可调控细胞核内DNA转录,诱导趋化因子(CXCL1、CXCL2、CXCL8、CXCL10、CCL20)、细胞因子(IL-6、TNF-α、COX2、G-CSF)、抗微生物肽(Defensins、MUC5AC、MUC5B)、基质金属蛋白酶(MMP1、MMP2、MMP9、MMP13)和标志性IL-17炎症因子的表达产生,而以上基因的表达可能会导致机体自身免疫疾病、中性粒细胞浸润、炎症反应和细胞免疫病原体的形成(图8)。
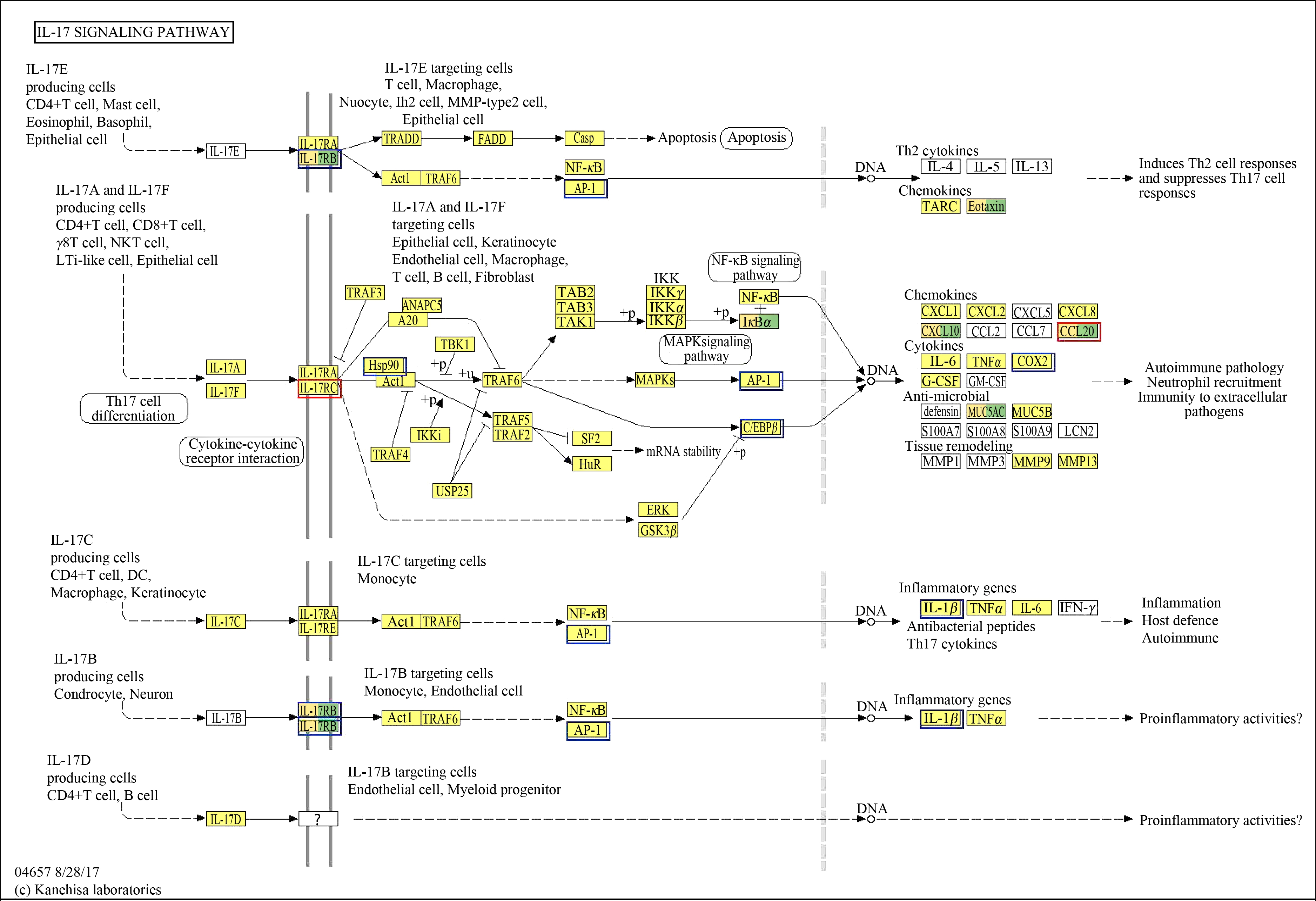
图8 鳃IL-17相关通路
注:蓝色方框代表基因/转录本下调;红色方框代表基因/转录本上调。
Fig. 8 Gill IL-17 related pathway
Note: Blue box represents gene/transcript down-regulation; red box represents gene/transcript up-regulation.
米氏凯伦藻对鳃IL-17相关通路的影响还导致了其下游MAPK信号通路被激活。这与MC-LR通过抑制蛋白磷酸酶PP2A导致MAPK相关通路被激活的结果较为相似[43-44],MAPK被激活后会调节原癌基因的表达,已知靶点包括原癌基因fosab和junba[45-46],这2个基因参与调控与生长、分化和细胞增殖的基因转录[44-45]。本研究中受藻影响的MAPK信号通路激活后下调了原癌基因fos、fosb、jund的转录水平,也影响了肿瘤坏死因子信号通路(图8),这表明米氏凯伦藻可能会诱导海水青鳉肿瘤相关疾病的产生。
同时在鳃IL-17信号通路中也观察到热休克蛋白hsp90的显著下降(图8)。热休克蛋白,已被推荐作为环境压力的生化指标,它可以通过防止重要蛋白质的不可逆损失并促进其随后的再生来保护细胞免受氧化应激[47],hsp70还与先天免疫和适应性免疫有关[48]。在肝脏中也发现氧化还原酶及其过程的显著标记,这或许表明米氏凯伦藻对海水青鳉鳃和肝脏造成了一定程度的氧化损伤,这与相较于海洋卡盾藻对贝类的影响集中于氧化应激通路较为相似[49]。与此同时,米氏凯伦藻对海水青鳉的影响还显著富集到低氧诱导因子信号通路(图4(a)),说明米氏凯伦藻将可能会导致海水青鳉缺氧现象产生。
离子跨膜运输对细胞的生存和生长至关重要,离子平衡维持着体液的稳态[50],而Na+-K+-ATP酶在离子平衡和渗透压调节中起着关键作用,其能为Na+-K+跨膜主动转运提供能量,在鱼鳃中酶活力也最高[51]。同时米氏凯伦藻能在短时间内显著抑制轮虫Na+-K+-ATP酶活性[21],降低海水青鳉Na+-K+-ATP酶相关基因转录表达水平[16],从而影响生物渗透压调节功能。同样在本研究GO富集发现,米氏凯伦藻能显著影响海水青鳉鳃的离子跨膜运输功能,这表明米氏凯伦藻可能导致鳃渗透压失调,细胞破裂,这和之前文献的结果能够对应[12, 52]。
实验结果同样表明米氏凯伦藻对海水青鳉的节律调节有一定的影响。昼夜节律调节在睡眠、摄食、生殖和细胞代谢等多种生理功能中发挥着重要作用,生物钟不仅存在于中枢神经系统,也存在于外周器官和组织[53]。昼夜节律分子机制主要由4个核心蛋白组成,首先是昼夜运动输出蛋白Clock和转录调控时钟控制蛋白BMal1,同时激活周期Per和隐色素Cry蛋白的转录,从而通过抑制Clock:BMal1介导的转录而产生负反馈循环[54]。同时在斑马鱼中发现了编码这4种蛋白质的基因在外周组织能进行有效表达[55]。海水青鳉的繁殖特性也受光周期调节的影响,其基因per1和per3也发生了显著上调,而昼夜节律调节相关基因的扰动是否会影响海水青鳉的生殖功能和代谢途径还有待进一步验证。
通信作者简介:李晓东(1992—),男,博士,副教授,主要研究方向为生态毒理学。
[1] Sha J, Xiong H Y, Li C J, et al. Harmful algal blooms and their eco-environmental indication [J]. Chemosphere, 2021, 274: 129912
[2] 李晓东. 米氏凯伦藻(福建株)毒性效应与毒理机制的研究[D]. 北京: 中国科学院大学, 2018: 1-10
Li X D. Study on toxic effects and toxicological mechanism of Karenia michaeli (Fujian strain)[D].Beijing: University of Chinese Academy of Sciences, 2018: 1-10 (in Chinese)
[3] 孙科. 米氏凯伦藻(Karenia mikimotoi)对海洋生物毒性的实验研究[D]. 北京: 中国科学院研究生院, 2010: 1-7
Sun K. The experimental study on the toxicity of Karenia mikimotoi to marine animals[D]. Beijing: Graduate School of Chinese Academy of Sciences, 2010: 1-7(in Chinese)
[4] 周名江, 于仁成. 有害赤潮的形成机制、危害效应与防治对策[J]. 自然杂志, 2007, 29(2): 72-77, 125
Zhou M J, Yu R C. Mechanisms and impacts of harmful algal blooms and the countmeasures [J]. Chinese Journal of Nature, 2007, 29(2): 72-77, 125 (in Chinese)
[5] Ho J C, Michalak A M, Pahlevan N. Widespread global increase in intense lake phytoplankton blooms since the 1980s [J]. Nature, 2019, 574(7780): 667-670
[6] Xiao X, Agustí S, Pan Y R, et al. Warming amplifies the frequency of harmful algal blooms with eutrophication in Chinese coastal waters [J]. Environmental Science &Technology, 2019, 53(22): 13031-13041
[7] Xu H, Paerl H W, Qin B, et al. Determining critical nutrient thresholds needed to control harmful cyanobacterial blooms in eutrophic Lake Taihu, China [J]. Environmental Science &Technology, 2015, 49(2): 1051-1059
[8] Yan T, Li X D, Tan Z J, et al. Toxic effects, mechanisms, and ecological impacts of harmful algal blooms in China [J]. Harmful Algae, 2022, 111: 102148
[9] Li X D, Yan T, Yu R C, et al. A review of Karenia mikimotoi: Bloom events, physiology, toxicity and toxic mechanism [J]. Harmful Algae, 2019, 90: 101702
[10] 国家海洋局. 2005年中国海洋灾害公报[R]. 北京: 国家海洋局, 2005
[11] 国家海洋局. 2012年中国海洋灾害公报[R]. 北京: 国家海洋局, 2012
[12] 林佳宁. 东海大规模赤潮危害的现场及实验研究[D]. 北京: 中国科学院大学, 2015: 39-45
Lin J N. Field and experimental study on the harm of large-scale red tide in the East China Sea [D]. Beijing: University of Chinese Academy of Sciences, 2015: 39-45 (in Chinese)
[13] 张勇, 杨维东, 李宏业, 等. 米氏凯伦藻对蒙古裸腹溞的毒性及致毒途径分析[J]. 生态毒理学报, 2011, 6(1): 94-98
Zhang Y, Yang W D, Li H Y, et al. Toxicity analysis of Karenia mikimotoi to Moina mongolica [J]. Asian Journal of Ecotoxicology, 2011, 6(1): 94-98 (in Chinese)
[14] 孙军, 宋书群, 徐兆礼, 等. 东海米氏凯伦藻水华中中华哲水蚤的选择性摄食[J]. 海洋与湖沼, 2007, 38(6): 536-541
Sun J, Song S Q, Xu Z L, et al. The selective grazing of Calanus sinicus during a Karenia mikimotoi bloom in the East China Sea [J]. Oceanologia et Limnologia Sinica, 2007, 38(6): 536-541 (in Chinese)
[15] O’Boyle S, McDermott G, Silke J, et al. Potential impact of an exceptional bloom of Karenia mikimotoi on dissolved oxygen levels in waters off western Ireland [J]. Harmful Algae, 2016, 53: 77-85
[16] Zhang P P, Song X X, Zhang Y, et al. Assessing the effect of modified clay on the toxicity of Karenia mikimotoi using marine medaka (Oryzias melastigma) as a model organism [J]. Toxics, 2022, 10(3): 105
[17] Sola F, Masoni A, Fossat B, et al. Toxicity of fatty acid 18: 5n3 from Gymnodinium cf. mikimotoi: Ⅰ. Morphological and biochemical aspects on Dicentrarchus labrax gills and intestine [J]. Journal of Applied Toxicology, 1999, 19(4): 279-284
[18] Hégaret H, da Silva P M, Wikfors G H, et al. In vitro interactions between several species of harmful algae and haemocytes of bivalve molluscs [J]. Cell Biology and Toxicology, 2011, 27(4): 249-266
[19] Wang X J, Feng X Q, Zhuang Y, et al. Effects of ocean acidification and solar ultraviolet radiation on physiology and toxicity of dinoflagellate Karenia mikimotoi [J]. Harmful Algae, 2019, 81: 1-9
[20] Niu X Q, Xu S N, Yang Q Y, et al. Toxic effects of the dinoflagellate Karenia mikimotoi on zebrafish (Danio rerio) larval behavior [J]. Harmful Algae, 2021, 103: 101996
[21] Li X D, Yan T, Zhang Q C, et al. Inhibition to crucial enzymes in the lethal effects of the dinoflagellate Karenia mikimotoi on the rotifer Brachionus plicatilis [J]. Marine Environmental Research, 2020, 157: 104866
[22] De Rijcke M, van Acker E, Nevejan N, et al. Toxic dinoflagellates and Vibrio spp. act independently in bivalve larvae [J]. Fish &Shellfish Immunology, 2016, 57: 236-242
[23] Bentley D R, Balasubramanian S, Swerdlow H P, et al. Accurate whole human genome sequencing using reversible terminator chemistry [J]. Nature, 2008, 456(7218): 53-59
[24] Sun F Y, Liu S K, Gao X Y, et al. Male-biased genes in catfish as revealed by RNA-Seq analysis of the testis transcriptome [J]. PLoS One, 2013, 8(7): e68452
[25] Lin W, Guo H H, Wang L K, et al. Waterborne microcystin-LR exposure induced chronic inflammatory response via MyD88-dependent toll-like receptor signaling pathway in male zebrafish [J]. The Science of the Total Environment, 2020, 702: 134969
[26] Wei L L, Liu Y, Zhong S W, et al. Transcriptome analysis of grass carp provides insights into the immune-related genes and pathways in response to MC-LR induction [J]. Aquaculture, 2018, 488: 207-216
[27] 王晓杰, 肖潇, 李超, 等. 海洋酸化对海水青鳉胚胎骨骼发育的影响[J]. 海洋学报, 2015, 37(12): 116-122
Wang X J, Xiao X, Li C, et al. Impact of ocean acidification on skeletal development in embryonic marine medaka [J]. Haiyang Xuebao, 2015, 37(12): 116-122 (in Chinese)
[28] 王赛男, 刘春艳, 穆景利, 等. 不同分散剂对海水青鳉早期发育毒性影响的比较研究[J]. 海洋环境科学, 2013, 32(6): 801-805
Wang S N, Liu C Y, Mu J L, et al. Comparative study on the toxicity of different dispersants to the early development of marine medaka(Oryzias melatigma) [J]. Marine Environmental Science, 2013, 32(6): 801-805 (in Chinese)
[29] 伍辛泷, 黄乾生, 方超, 等. 新兴海洋生态毒理学模式生物——海洋青鳉鱼(Oryzias melastigma)[J]. 生态毒理学报, 2012, 7(4): 345-353
Wu X L, Huang Q S, Fang C, et al. Oryzias melastigma: A new promising model organism for marine ecotoxicology [J]. Asian Journal of Ecotoxicology, 2012, 7(4): 345-353 (in Chinese)
[30] Qiao Q, Le Manach S, Huet H, et al. An integrated omic analysis of hepatic alteration in medaka fish chronically exposed to cyanotoxins with possible mechanisms of reproductive toxicity [J]. Environmental Pollution, 2016, 219: 119-131
[31] Guillard R R, Ryther J H. Studies of marine planktonic diatoms. I. Cyclotella nana Hustedt, and Detonula confervacea (Cleve) Gran [J]. Canda Journal of Microbiology, 1962, 8: 229-239
[32] 孙军, 刘东艳, 钱树本. 浮游植物生物量研究 Ⅰ.浮游植物生物量细胞体积转化法[J]. 海洋学报, 1999, 21(2): 75-85
Sun J, Liu D Y, Qian S B. Study on phytoplankton biomass Ⅰ. Phytoplankton measurement biomass from cell volume or plasma volume [J]. Acta Oceanologica Sinica, 1999, 21(2): 75-85 (in Chinese)
[33] Schmittgen T D, Livak K J. Analyzing real-time PCR data by the comparative CT method [J]. Nature Protocols, 2008, 3(6): 1101-1108
[34] Bo J, Giesy J P, Ye R, et al. Identification of differentially expressed genes and quantitative expression of complement genes in the liver of marine medaka Oryzias melastigma challenged with Vibrio parahaemolyticus [J]. Comparative Biochemistry and Physiology Part D, Genomics &Proteomics, 2012, 7(2): 191-200
[35] Li X D, Yan T, Lin J N, et al. Detrimental impacts of the dinoflagellate Karenia mikimotoi in Fujian coastal waters on typical marine organisms [J]. Harmful Algae, 2017, 61: 1-12
[36] Lin J N, Yan T, Zhang Q C, et al. Impact of several harmful algal bloom (HAB) causing species, on life history characteristics of rotifer Brachionus plicatilis Müller [J]. Chinese Journal of Oceanology and Limnology, 2016, 34(4): 642-653
[37] Evans D H, Piermarini P M, Choe K P. The multifunctional fish gill: Dominant site of gas exchange, osmoregulation, acid-base regulation, and excretion of nitrogenous waste [J]. Physiological Reviews, 2005, 85(1): 97-177
[38] Zhang X, Wang S L, Chen S L, et al. Transcriptome analysis revealed changes of multiple genes involved in immunity in Cynoglossus semilaevis during Vibrio anguillarum infection [J]. Fish &Shellfish Immunology, 2015, 43(1): 209-218
[39] Mollnes T E, Song W C, Lambris J D. Complement in inflammatory tissue damage and disease [J]. Trends in Immunology, 2002, 23(2): 61-64
[40] Song W C, Rosa Sarrias M, Lambris J D. Complement and innate immunity [J]. Immunopharmacology, 2000, 49(1-2): 187-198
[41] 刘明英, 张士璀. 纤溶酶原: 结构、功能与进化[J]. 中国海洋大学学报(自然科学版), 2010, 40(10): 69-74
Liu M Y, Zhang S C. Plasminogen: Structure, function and evolution [J]. Periodical of Ocean University of China, 2010, 40(10): 69-74 (in Chinese)
[42] Ramm L E, Whitlow M B, Mayer M M. Transmembrane channel formation by complement: Functional analysis of the number of C5b6, C7, C8, and C9 molecules required for a single channel [J]. Proceedings of the National Academy of Sciences of the United States of America, 1982, 79(15): 4751-4755
[43] Gehringer M M. Microcystin-LR and okadaic acid-induced cellular effects: A dualistic response [J]. FEBS Letters, 2004, 557(1-3): 1-8
[44] Wang X T, Chen Y, Zuo X T, et al. Microcystin (-LR) induced testicular cell apoptosis via up-regulating apoptosis-related genes in vivo [J]. Food and Chemical Toxicology, 2013, 60: 309-317
[45] Delaney J, Chiarello R, Villar D, et al. Regulation of c-fos, c-Jun and c-myc gene expression by angiotensin Ⅱ in primary cultured rat astrocytes: Role of ERK1/2 MAP kinases [J]. Neurochemical Research, 2008, 33(3): 545-550
[46] Zegura B, Straser A, ![]() M. Genotoxicity and potential carcinogenicity of cyanobacterial toxins - A review [J]. Mutation Research, 2011, 727(1-2): 16-41
M. Genotoxicity and potential carcinogenicity of cyanobacterial toxins - A review [J]. Mutation Research, 2011, 727(1-2): 16-41
[47] Molina A, Carpeaux R, Martial J A, et al. A transformed fish cell line expressing a green fluorescent protein-luciferase fusion gene responding to cellular stress [J]. Toxicology in Vitro, 2002, 16(2): 201-207
[48] Srivastava P. Roles of heat-shock proteins in innate and adaptive immunity [J]. Nature Reviews Immunology, 2002, 2(3): 185-194
[49] 李晓登. 海洋卡盾藻对翡翠贻贝的致毒效应[D]. 广州: 暨南大学, 2014: 18-45
Li X D. Toxic effects of Chattonella marina to Perna viridis [D]. Guangzhou: Jinan University, 2014: 18-45 (in Chinese)
[50] Guh Y J, Hwang P P. Insights into molecular and cellular mechanisms of hormonal actions on fish ion regulation derived from the zebrafish model [J]. General and Comparative Endocrinology, 2017, 251: 12-20
[51] Zhang X Y, Wen H S, Qi X, et al. Na+-K+-ATPase and nka genes in spotted sea bass (Lateolabrax maculatus) and their involvement in salinity adaptation [J]. Comparative Biochemistry and Physiology Part A, Molecular &Integrative Physiology, 2019, 235: 69-81
[52] 欧祥亚. 米氏凯伦藻赤潮对东海养殖鱼类鳃组织结构损害作用的研究[D]. 广州: 暨南大学, 2006: 16-37
Ou X Y. Histopathological effect of Karenia mikimotoi bloom on gills of common farmed fishes in the East China Sea [D]. Guangzhou: Jinan University, 2006:16-37 (in Chinese)
[53] Singh V K, Berry L, Bernut A, et al. A unique PE_PGRS protein inhibiting host cell cytosolic defenses and sustaining full virulence of Mycobacterium marinum in multiple hosts [J]. Cellular Microbiology, 2016, 18(11): 1489-1507
[54] Dunlap J C. Molecular bases for circadian clocks [J]. Cell, 1999, 96(2): 271-290
[55] Cahill G M. Clock mechanisms in zebrafish [J]. Cell and Tissue Research, 2002, 309(1): 27-34