在商用的纳米材料中,金属及其氧化物是目前生产和应用最多的纳米级产品之一[1-2]。其中纳米氧化钴(cobalt (Ⅱ, Ⅲ) oxide nanoparticles, Co3O4-NPs)因其优异的电催化性能和磁性特点,被广泛用于生产锂电池和气体传感器[3-4]。另外,Co3O4-NPs有望在靶向诊断、治疗和给药过程中发挥重要作用[5-7]。
然而,广泛的应用增加了纳米材料对人类健康和环境暴露的风险。研究表明,活性氧(reactive oxygen species, ROS)和氧化应激反应是进入细胞内的纳米材料引起细胞损伤的主要原因之一[8-11]。另一方面,纳米材料引起的细胞毒性也与其诱导的组蛋白修饰(histone modification)变化密切相关[12-14]。在各种组蛋白修饰中,组蛋白H3修饰程度最高,可通过直接影响染色质结构或招募染色质重塑复合物来调节常染色质与异染色质之间的动态转换[15],在相关修饰酶的作用下,调节基因激活和沉默的动态变化[16]。此外,组蛋白H3修饰参与调控DNA修复[17-18],在决定细胞命运、细胞生长以及致癌作用的过程中也发挥着重要的作用[19]。然而,Co3O4-NPs暴露是否引起组蛋白修饰变化尚不明确,其潜在风险亟待探讨。
基于此,本文在对Co3O4-NPs进行详细表征的基础上,以人永生化角质形成细胞HaCaT为模型,采用蛋白免疫印迹法及流式细胞技术等研究手段,探究了Co3O4-NPs作用后的细胞生存率、细胞内蓄积量以及组蛋白H3修饰变化及其潜在的调控机制。
1 材料与方法(Materials and methods)
1.1 实验材料
HaCaT细胞(上海传秋生物科技有限公司);Co3O4-NPs (平均粒径<50 nm,美国Sigma-Aldrich公司)。聚偏二氟乙烯膜(0.45 μm,美国Millipore公司);3M滤纸(3 mm,美国Thermo Fisher公司)。
1.2 实验试剂
DMEM细胞培养基(01863,美国Thermo Fisher公司);胎牛血清(193254C,美国Thermo Fisher公司);青链霉素双抗(J170027,美国Thermo Fisher公司);2.5%胰蛋白酶溶液(1911594,美国Thermo Fisher公司);WB细胞蛋白裂解液(P0013,上海碧云天生物技术有限公司);抗体(AF1201、AF1180、AF1207、AF5614、AF5704、AF5710、A0208,上海碧云天生物技术有限公司);BCA蛋白定量试剂盒(BCA02,北京鼎国昌盛有限公司);WB化学发光试剂盒(TG268245,美国Thermo Fisher公司);RNA提取试剂盒(R6834-01,美国Omega公司);反转录试剂盒(RR047A,日本Takara公司);荧光定量PCR试剂盒(RR420A,日本Takara公司)。
1.3 实验仪器
场发射扫描电子显微镜(SU8010,日本Hitachi公司);激光粒度仪(WINNER803,济南微纳颗粒仪器股份有限公司,中国);比表面积微孔吸附仪(ASPA2460,美国Micromeritics公司);X射线粉末衍射仪(XRD-6100,日本Shimadzu公司);密闭式超声破碎仪(VCX150,美国Sonics公司);二氧化碳培养箱(3111,美国Thermo Fisher公司);酶标仪(INFINITE F50,瑞士Tecan公司);电泳仪(EPS301,美国Thermo Fisher公司);电泳槽(AE-6500,日本Atto株式会社);转膜仪(TE77,美国Thermo Fisher公司);化学发光成像系统(AI600,美国Thermo Fisher公司);流式细胞仪(A00-1-1102,美国Beckman Coulter公司);荧光定量PCR仪(CFX96,新加坡Bio-Rad公司);梯度PCR仪(T100,新加坡Bio-Rad公司);超微量核酸/蛋白分析仪(Duo+,美国Bio Drop公司)。
1.4 实验方法
1.4.1 样品前处理
实验当日,称取适量的Co3O4-NPs,用含0.5%胎牛血清及1%青链霉素双抗的DMEM细胞培养基(0.5% DMEM)配制成10~50 mg·mL-1的悬浊液。为使Co3O4-NPs分散,使用密闭式超声破碎仪在60 W功率下超声1 min。另外,用于X射线粉末衍射及比表面积微孔表征的Co3O4-NPs及其悬浊液在测试前,经-50 ℃、24 h真空冷冻干燥处理后置于干燥器内保存备用。
1.4.2 纳米材料表征
1.4.2.1 场发射扫描电子显微镜表征
将适量待测样品均匀涂抹或滴加于导电胶上,喷金150 s后上机测试。测试参数为扫描加速电压10~13 kV,加速电流10 μA,观察倍率70 000~110 000倍。
1.4.2.2 动态光散射表征
Co3O4-NPs的流体动力学粒径(二次粒径)及其分布采用动态光散射法(dynamic light scattering, DLS)表征。测试参数为折射角度90°,分散介质折射率1.333,介质黏度0.0008904 Pa·s,粒度测试范围为1~10 000 nm,设备延迟单位时间10 μs。
1.4.2.3 X射线粉末衍射表征
取适量经干燥处理后的待测样品置于样品槽,用玻璃板压片压平待测样品,随后测试。测试参数为仪器铜靶辐射源CuKα (λ=0.154056 nm),扫描速度为2(°)·min-1,扫描角度为4~90°。
1.4.2.4 比表面积微孔表征
比表面积微孔表征采用氮气吸附-脱附(Brunauer-Emmett-Teller, BET)表征方法。分别称取0.1~0.2 g经干燥处理后的待测样品置于BET试管底部,随后使用真空干燥脱气仪在200 ℃条件下对待测样品脱气2 h。在相对压力P/P0为0.99、-195.85 ℃条件下获得氮气吸附-脱附等温曲线。P/P0在0.05~0.35之间线性拟合。
1.5 供试细胞培养
人体可以通过皮肤接触、口鼻吸入、注射等暴露途径吸收和积累纳米材料。其中,皮肤是人体接触纳米材料的主要途径之一。基于此,选取了人永生化角质形成细胞HaCaT为模型进行了实验。HaCaT细胞经0.25%胰蛋白酶溶液消化传代后,接种于含10%胎牛血清及1%青链霉素双抗的DMEM细胞培养基(10% DMEM)的100 mm细胞培养皿中,置于37 ℃、5% CO2及饱和湿度的二氧化碳培养箱中静置培养,细胞贴壁面积达到70%~90%之间时,用0.5% DMEM细胞培养基冲洗细胞2次后,细胞在0.5% DMEM细胞培养基中继续培养24 h。
1.6 Co3O4-NPs处理HaCaT细胞
细胞在0.5% DMEM中培养24 h后,弃掉旧培养基,加入含样品的新鲜0.5% DMEM细胞培养基,使样品最终浓度为0.1、0.3、1、3、10、30、100、300及1 000 μg·mL-1。随后,分别将细胞置于培养箱中静置培养0.5、1、2、3、4、6、8、9、10、12、24、36、48、60及72 h。每批次实验对照组以未加入样品的细胞为空白对照,每个浓度及时间单次实验中设置3个重复,各组实验独立重复3次。
1.7 细胞生存率检测
至暴露时间点,弃培养基,磷酸盐缓冲液(phosphate buffer saline, PBS)冲洗细胞2次,0.25%胰蛋白酶溶液消化并收集细胞,制备单细胞悬液。将细胞悬液与台盼蓝染色液按1∶1吹打混匀后移至血球计数板。使用倒置显微镜观测细胞数量,并记录数值进行统计。每个样品的细胞生存率为4个1 mm×1 mm大方格中的细胞生存率的平均值。1个大方格中的细胞生存率的计算公式为:细胞生存率%=活细胞数/细胞总数×100%。
1.8 蛋白免疫印记法
至暴露时间点,细胞刮刀剥离细胞并移至离心管,PBS冲洗2次后弃上清液。随后向样品中添加适量WB细胞蛋白裂解液,超声破碎1 min后于4 ℃静置2 h。样品蛋白提取后参照BCA蛋白定量试剂盒说明书进行蛋白定量。将定量过的蛋白样品与蛋白凝胶电泳上样缓冲液按1∶1比例充分混合。样品经电泳、转膜、染色、脱色、封闭后,在4 ℃下一抗孵育12 h。随后,室温下二抗孵育2 h后上机检测。蛋白条带的影像采用Photoshop 2020软件进行分析统计。
1.9 纳米材料的细胞内蓄积量检测
利用流式细胞仪侧向散射光(side scattered light, SS)的变化研究细胞内纳米材料的蓄积量[20]。细胞摄入纳米材料颗粒后,细胞内部精细结构和细胞粒度改变,引起SS增加。至暴露时间点,弃培养基,PBS冲洗细胞2次,0.25%胰蛋白酶消化并收集细胞,制备单细胞悬液。将细胞悬液移至新的流式管,使用流式细胞仪检测,每组数据检测10 000个细胞,每组样品检测3次,采用FlowJo 10.7.1软件对所得实验结果进行分析。
1.10 实时荧光定量PCR
至暴露时间点,弃培养基,PBS冲洗细胞2次,0.25%胰蛋白酶溶液消化并收集约107个细胞,使用RNA提取试剂盒提取总RNA。采用超微量核酸/蛋白分析仪定量待测样品RNA浓度。随后使用反转录试剂盒反转录RNA (800 ng)。使用荧光定量PCR试剂盒对获得的cDNA模板进行聚合酶链式扩增反应。反应条件为:预变性(95 ℃/30 s),变性(95 ℃/5 s),退火(64 ℃/30 s),延伸(72 ℃/1 min),共40个循环。本研究采用基因相对表达量2-ΔΔCq公式进行计算,通过SPSS 26软件对数据进行分析。用于实时荧光定量PCR (real-time quantitative PCR, RT-qPCR)的内参基因及扩增引物如表1所示。
表1 RT-qPCR内参基因及扩增引物[21-24]
Table 1 Internal reference genes and amplification primers for RT-qPCR[21-24]

注:GAPDH表示甘油醛-3-磷酸脱氢酶基因;p300表示组蛋白乙酰转移酶p300基因;HDAC1表示组蛋白去乙酰化酶1基因;LSD1表示组蛋白去甲基化酶1基因。
Note: GAPDH stands for glyceraldehyde-3-phosphate dehydrogenase gene; p300 stands for histone acetyltransferase p300 gene; HDAC1 stands for histone deacetylase 1 gene; LSD1 stands for lysine specific demethylase 1 gene.
基因Gene引物序列Primer sequence产物大小/bpProduct size/bpGAPDHForward: GAGTCAACGGATTTGGTCGT; Reverse: TTGATTTTGGAGGGATCTCG238 p300Forward: GGGACTAACCAATGGTGGTG; Reverse: GCCACCAACTCCCATATTGA139 HDAC1Forward: AACCTGCCTATGCTGATGCT; Reverse: CAGGCAATTCGTTTGTCAGA374 LSD1Forward: GCTCGGGGCTCTTATTCCTA; Reverse: CCCAAAAACTGGTCTGCAAT224
2 结果(Results)
2.1 Co3O4-NPs表征
图1(a)为Co3O4-NPs及经0.5% DMEM分散处理的Co3O4-NPs的场发射扫描电子显微镜(scanning electron microscope, SEM)图像。与供应商标注信息基本一致,未分散前的Co3O4-NPs (左)呈现较为规则均匀的球形,单个颗粒粒径(一次粒径)约为50 nm,颗粒之间界线分明,呈分散堆叠、松散聚集状态,表明颗粒之间黏性较低。经0.5% DMEM分散处理后的Co3O4-NPs (右)呈现球形,但颗粒之间界线模糊,呈成片聚集、无序堆叠状态,颗粒之间黏性显著升高,出现明显团聚现象,其粒径(二次粒径)约为100 nm。为了进一步评估Co3O4-NPs在0.5% DMEM中的分散情况,进行了DLS测试分析。经0.5% DMEM分散处理后,Co3O4-NPs的粒径分布集中在100~300 nm之间,分布较为均匀,其DLS平均粒径(二次粒径)为184.88 nm,与一次粒径(<50 nm)相比,由于团聚其二次粒径增大约3倍(图1(b))。另外,由图1(c)可知,2种样品的吸附-脱附等温曲线均为Ⅳ型,在相同压力下,Co3O4-NPs的吸附量大于经0.5% DMEM分散处理后的Co3O4-NPs,其BET比表面积分别为(40.8870±0.1175) m2·g-1及(25.1706±0.1003) m2·g-1。表明Co3O4-NPs堆积松散,其孔间隙更大,而经分散处理后的Co3O4-NPs则由于聚集程度高,其孔间隙更小。X射线粉末衍射仪(X-ray-diffraction, XRD)分析结果表明,Co3O4-NPs的晶型为立方晶(Cubic),PDF卡片号为74-2120,且无论分散(团聚)与否其晶型特征无明显变化,均能保持良好的晶型结构(图1(d)。但值得注意的是,观察到经0.5% DMEM分散处理后的样品中出现NaCl (PDF卡片号为75-0306)的衍射峰。实验结果表明,经0.5% DMEM分散处理后,由于团聚Co3O4-NPs的比表面积减小、粒径增大。
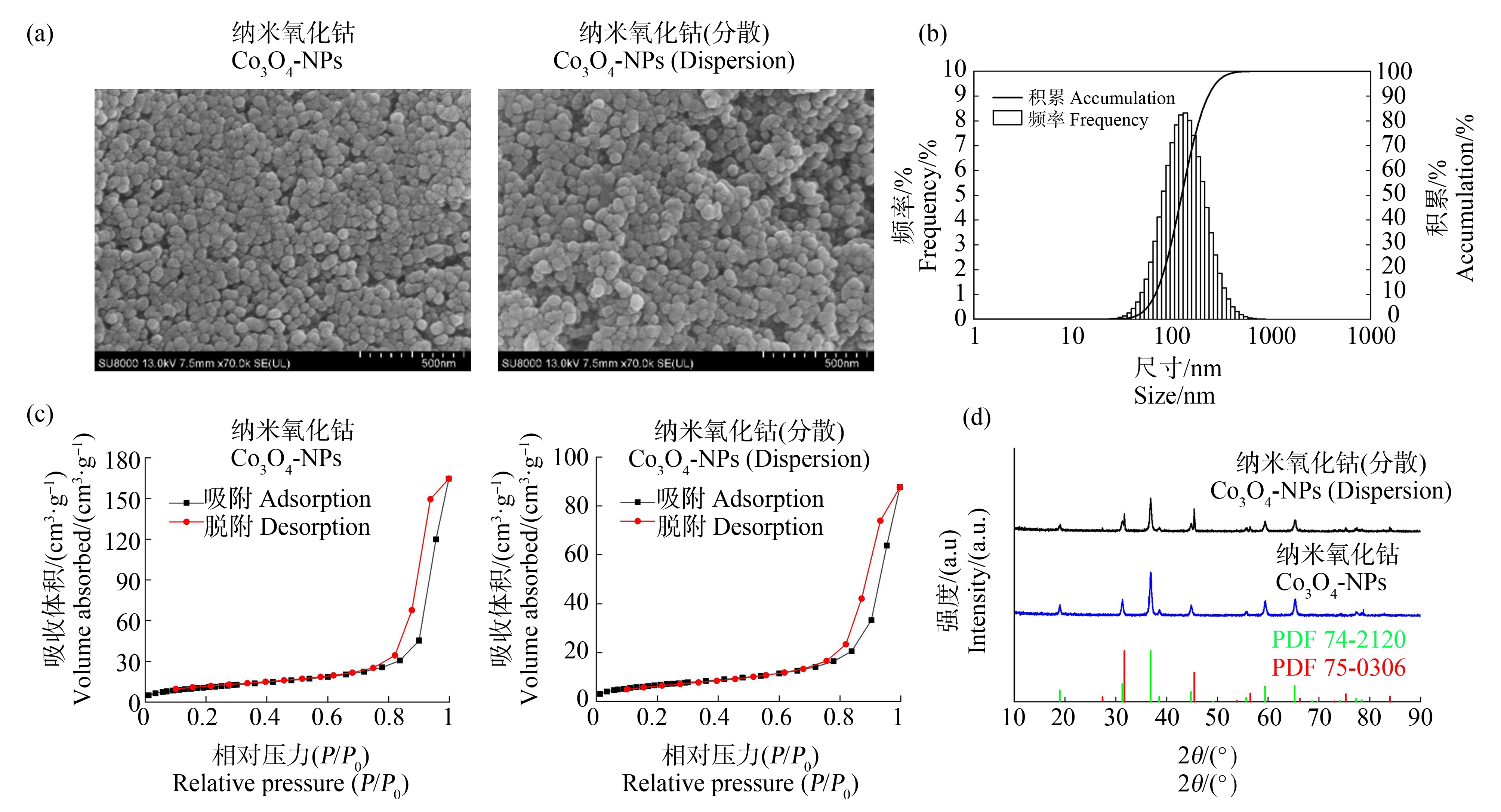
图1 分散介质中Co3O4-NPs的理化性质
注:(a) SEM;(b) DLS;(c) BET;(d) XRD。
Fig. 1 Physicochemical properties of Co3O4-NPs in dispersion medium
Note: (a) SEM; (b) DLS; (c) BET; (d) XRD.
2.2 Co3O4-NPs暴露后细胞生存率的变化
为了确定Co3O4-NPs对HaCaT细胞生存率的影响,利用台盼蓝染色法测定了Co3O4-NPs暴露后的细胞生存率(图2)。实验结果表明,不同剂量梯度的Co3O4-NPs (~1 mg·mL-1)暴露24 h后,HaCaT细胞生存率略有下降,但不明显。实验最大剂量(1 mg·mL-1)作用24 h后,HaCaT细胞生存率仅下降约6%,与对照组生存率(97%)基本一致(图2(a))。另外,观察了较低剂量的Co3O4-NPs长时间暴露对HaCaT细胞生存率的影响。不同时间梯度的实验结果表明,0.1 mg·mL-1 Co3O4-NPs长时间暴露后,HaCaT细胞生存率无明显变化,均高于90% (图2(b)和(c))。

图2 Co3O4-NPs暴露后的细胞生存率
注:(a) 剂量依赖;(b) 时间依赖(~24 h);(c) 时间依赖(~72 h)。
Fig. 2 Cell viability after exposure to Co3O4-NPs
Note: (a) Dose-dependent; (b) Time-dependent (~24 h); (c) Time-dependent (~72 h).
2.3 Co3O4-NPs对组蛋白H3修饰的影响
为了确定Co3O4-NPs对组蛋白H3常见修饰位点的影响,利用蛋白免疫印迹法观察了Co3O4-NPs暴露后HaCaT细胞的组蛋白H3第10位丝氨酸的磷酸化(p-H3S10)、第9位赖氨酸的乙酰化(Ac-H3K9)、第14位赖氨酸的乙酰化(Ac-H3K14)、第4位赖氨酸的三甲基化(Me3-H3K4)及第27位赖氨酸的三甲基化(Me3-H3K27)修饰水平变化(图3)。不同剂量Co3O4-NPs (~1 mg·mL-1)暴露1 h后,p-H3S10修饰水平随着暴露剂量的升高而升高,显示出较为明显的剂量依赖性(图3(a))。与p-H3S10的结果相似,Ac-H3K9、Me3-H3K4及Me3-H3K27修饰水平也随着暴露剂量的升高而升高。但是,未观察到暴露1 h内Ac-H3K14修饰水平有明显变化。另一方面,低剂量0.1 mg·mL-1 Co3O4-NPs长时间(~36 h)暴露的结果表明(图3(b)),Co3O4-NPs暴露后诱导了p-H3S10修饰水平的升高并持续长达24 h。同样的,随着暴露时间的增加,观察到Ac-H3K9、Ac-H3K14及Me3-H3K4修饰水平明显上调。另外,0.1 mg·mL-1 Co3O4-NPs暴露后Me3-H3K27修饰水平上调,但在暴露4 h及8 h时修饰水平降低。实验结果表明,Co3O4-NPs暴露后短时间内(1 h)即引起了p-H3S10、Ac-H3K9及Me3-H3K4的高表达,并持续长达24 h。Co3O4-NPs暴露后引起了Ac-H3K14修饰水平的上调,但晚于其他被测修饰位点。另外,Me3-H3K27修饰的上调在Co3O4-NPs暴露4 h后,出现先下降再升高趋势。

图3 Co3O4-NPs暴露后的组蛋白H3修饰
注:p-H3S10表示组蛋白H3第10位丝氨酸的磷酸化;Ac-H3K9表示组蛋白H3第9位赖氨酸的乙酰化;Ac-H3K14表示组蛋白H3第14位赖氨酸的乙酰化;Me3-H3K4表示组蛋白H3第4位赖氨酸的三甲基化;Me3-H3K27表示组蛋白H3第27位赖氨酸的三甲基化;(a) 剂量依赖;(b) 时间依赖。
Fig. 3 Histone H3 modification after exposure to Co3O4-NPs
Note: p-H3S10 stands for phosphorylation of histone H3 at serine 10; Ac-H3K9 stands for acetylation of histone H3 at lysine 9; Ac-H3K14 stands for acetylation of histone H3 at lysine 14; Me3-H3K4 stands for trimethylation of histone H3 at lysine 4; Me3-H3K27 stands for trimethylation of histone H3 at lysine 27; (a) Dose-dependent; (b) Time-dependent.
2.4 细胞内Co3O4-NPs的蓄积量
不同于传统的材料,纳米材料更容易进入细胞,从而在细胞内引起各种功能损伤[9, 11]。为了探究Co3O4-NPs对组蛋白H3修饰影响的机制,利用流式细胞仪的侧向散射光SS分析检测了细胞内Co3O4-NPs的蓄积量。图4(a)显示了Co3O4-NPs (~1 mg·mL-1)暴露1 h后,流式细胞仪的前向散射光(forward scattered light, FS)和SS的点状图。实验结果表明,Co3O4-NPs作用对FS的强度没有影响,而平均SS强度(mean SS)以剂量依赖的方式增加,各样本平均SS强度分别为74.994、75.840、76.527、78.439、81.189、86.691、89.397、90.320、104.852及118.632。另外,观察到SS强度并未随着1 mg·mL-1 Co3O4-NPs暴露时间的增加而增强,各暴露时间点的结果与1 h时基本一致(图4(b))。
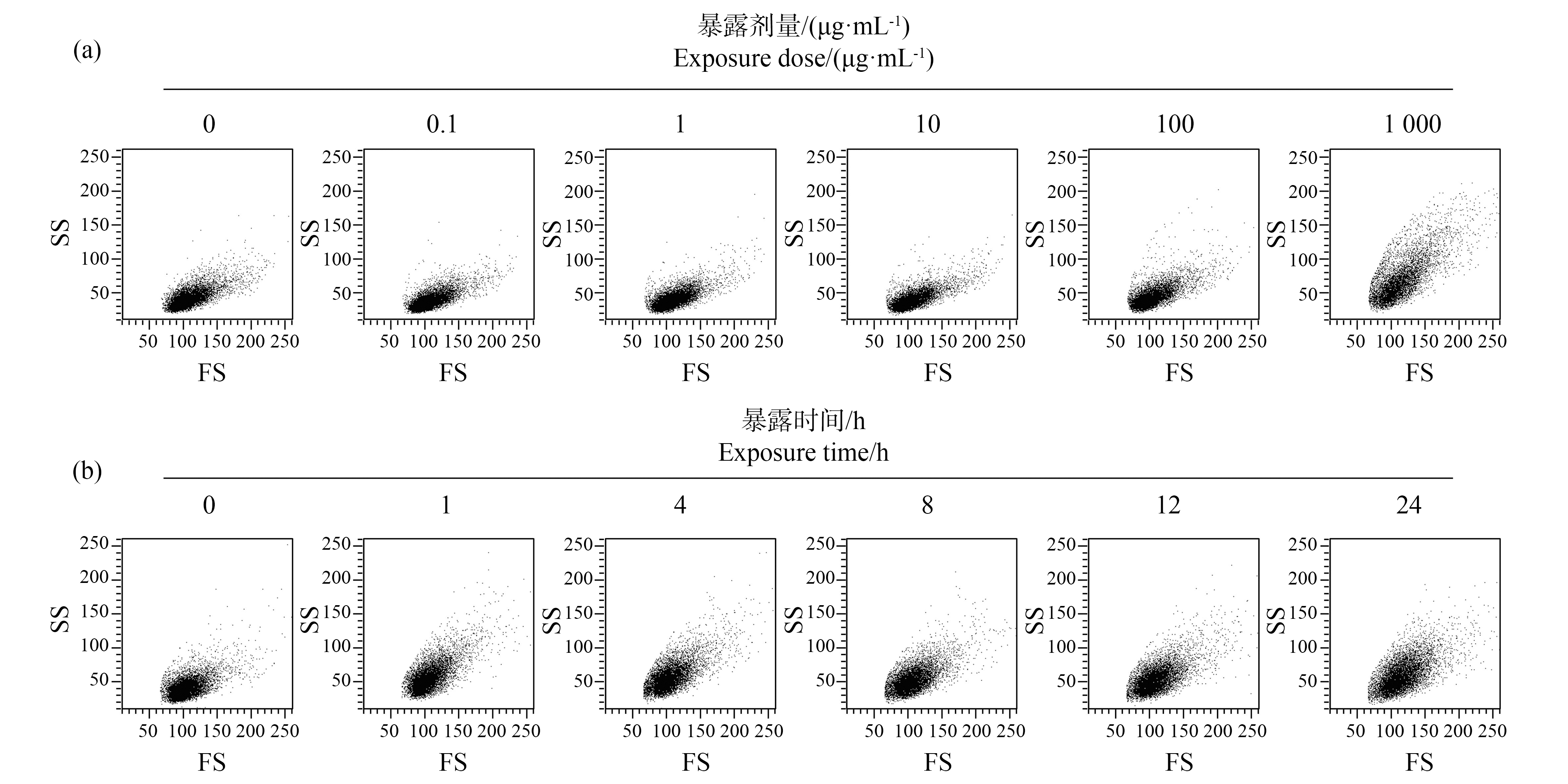
图4 Co3O4-NPs暴露后的细胞内蓄积量
注:SS表示侧向散射光;FS表示前向散射光;(a) 剂量依赖;(b) 时间依赖。
Fig. 4 Intercellular accumulation after exposure to Co3O4-NPs
Note: SS stands for side scattered light; FS stands for forward scattered light; (a) Dose-dependent; (b) Time-dependent.
为了进一步确定细胞内Co3O4-NPs的蓄积量与组蛋白修饰之间的关系,使用Photoshop 2020软件定量了图2(a)中所示的p-H3S10、Ac-H3K9及Me3-H3K4条带,并计算了SS和各组蛋白H3修饰强度的平均值。随着Co3O4-NPs的暴露剂量的增加,Co3O4-NPs的蓄积量(平均SS强度)和p-H3S10、Ac-H3K9及Me3-H3K4的强度平行增加(图5)。各自的相关系数为r2=0.67767、r2=0.81936及r2=0.80852。
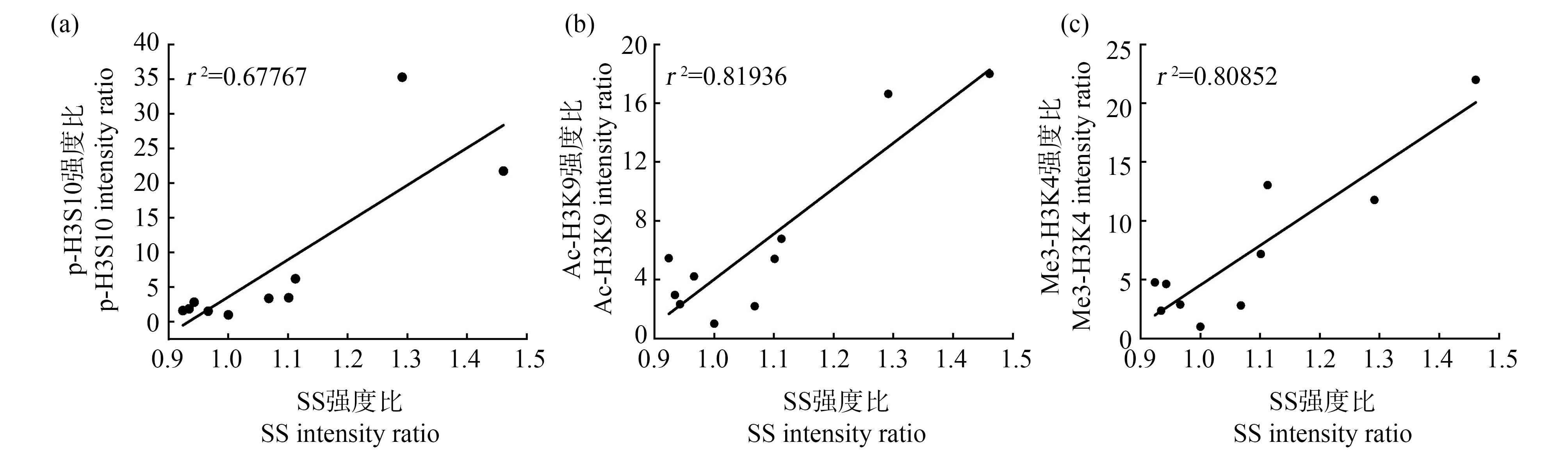
图5 Co3O4-NPs的细胞内蓄积量与组蛋白H3修饰之间的相关性
注:使用Photoshop 2020提取了蛋白免疫印记法测定后的组蛋白H3修饰强度;计算了Co3O4-NPs处理与未处理细胞的组蛋白H3修饰程度及细胞内蓄积量的比值(p-H3S10、Ac-H3K9、Me3-H3K4及SS强度比);使用最小二乘法计算了相关性;(a) p-H3S10与SS;(b) Ac-H3K9与SS;(c) Me3-H3K4与SS。
Fig. 5 Correlation between intercellular accumulation of Co3O4-NPs and histone H3 modification
Note: Histone H3 modification was determined using Western blotting, where the intensity of each band was extracted using Photoshop 2020; the intensity ratio of histone H3 modification and intracellular accumulation in cells treated with Co3O4-NPs, versus untreated control cells, was calculated; correlations were calculated using the least-squares method; (a) p-H3S10 and SS; (b) Ac-H3K9 and SS; (c) Me3-H3K4 and SS.
2.5 Co3O4-NPs诱导DNA损伤形成
为了确定进入细胞内的Co3O4-NPs是否引起DNA损伤,观察了Co3O4-NPs暴露后组蛋白H2AX第139位丝氨酸的磷酸化(γ-H2AX)修饰变化(图6)。不同剂量Co3O4-NPs (~1 mg·mL-1)暴露1 h后,γ-H2AX修饰水平均随着暴露剂量的升高而升高,显示出明显的剂量依赖性(图6(a))。0.1 mg·mL-1 Co3O4-NPs长时间(~36 h)暴露后诱导了γ-H2AX修饰水平的升高并持续长达36 h (图6(b))。

图6 Co3O4-NPs暴露后的γ-H2AX修饰
注:γ-H2AX表示组蛋白H2AX第139位丝氨酸的磷酸化;(a) 剂量依赖;(b) 时间依赖。
Fig. 6 γ-H2AX modification after exposure to Co3O4-NPs
Note: γ-H2AX stands for phosphorylation of histone H2AX at serine 139; (a) Dose-dependent; (b) Time-dependent.
为了进一步确定细胞内Co3O4-NPs的蓄积量与DNA损伤以及DNA损伤与组蛋白H3修饰之间的关系,绘制了DNA损伤(γ-H2AX)与SS强度、DNA损伤与p-H3S10、Ac-H3K9、Me3-H3K4相关性的散点图(图7)。Co3O4-NPs的细胞内蓄积量与其诱导的DNA损伤高度相关(r2=0.70403)。另外,DNA损伤与p-H3S10、Ac-H3K9及Me3-H3K4的强度平行增加,各自的相关系数为r2=0.98644、r2=0.88165和r2=0.5501。
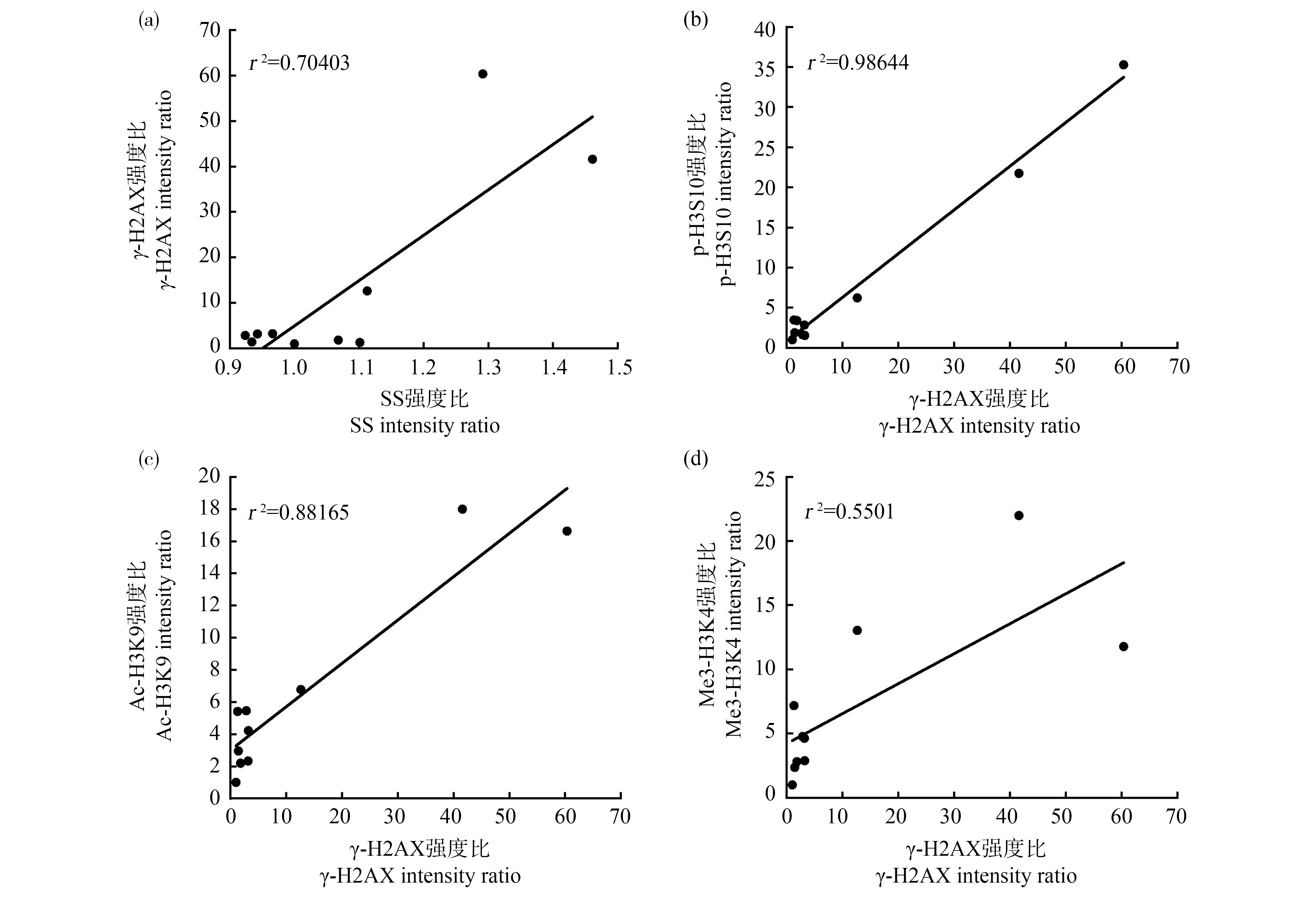
图7 DNA损伤与细胞内蓄积量及组蛋白H3修饰之间的相关性
注:使用Photoshop 2020提取了蛋白免疫印记法测定后的组蛋白H3修饰及DNA损伤强度;计算了Co3O4-NPs处理与未处理细胞的组蛋白H3修饰程度、DNA损伤程度及细胞内蓄积量的比值(p-H3S10、Ac-H3K9、Me3-H3K4、γ-H2AX及SS强度比);使用最小二乘法计算了相关性;(a) γ-H2AX与SS;(b) p-H3S10与γ-H2AX;(c) Ac-H3K9与γ-H2AX;(d) Me3-H3K4与γ-H2AX。
Fig. 7 Correlation between DNA damage and intercellular accumulation and histone H3 modification
Note: DNA damage and histone H3 modification was determined using Western blotting, where the intensity of each band was extracted using Photoshop 2020; the intensity ratio of histone H3 modification, DNA damage and intracellular accumulation in cells treated with Co3O4-NPs, versus untreated control cells, was calculated; correlations were calculated using the least-squares method; (a) γ-H2AX and SS; (b) p-H3S10 and γ-H2AX; (c) Ac-H3K9 and γ-H2AX; (d) Me3-H3K4 and γ-H2AX.
2.6 Co3O4-NPs对组蛋白H3修饰酶的影响
组蛋白修饰酶含有特定的结构域,在促进组蛋白修饰功能的多样性和维持细胞稳态发挥先锋作用[25]。采用RT-qPCR法考察了Co3O4-NPs暴露后3种常见的组蛋白修饰酶LSD1 (lysine specific demethylase)、HDAC1 (histone deacetylase 1)、p300 (histone acetyltransferase p300)的mRNA水平的变化。然而仅有LSD1显示出时间依赖性的变化,即随着0.1 mg·mL-1 Co3O4-NPs暴露时间的增加,LSD1的mRNA表达量呈下降趋势(图8)。其余2种组蛋白修饰酶的mRNA水平与空白对照相比差异无统计学意义。

图8 Co3O4-NPs暴露后LSD1的基因表达水平
注:LSD1表示组蛋白去甲基化酶1基因;与空白对照组相比差异显著,**P<0.01。
Fig. 8 Gene expression levels of LSD1 after exposure to Co3O4-NPs
Note: LSD1 stands for lysine specific demethylase 1 gene; compared with blank control group, there is significant difference, **P<0.01.
3 讨论(Discussion)
纳米材料的理化性质是影响其毒性的重要因素。观察到Co3O4-NPs无论分散与否,其颗粒形状大部分为球形(图1(a))。在其他条件相同的情况下,球形颗粒的表面积更大,表面反应位点更多。我们在经0.5% DMEM分散处理后的Co3O4-NPs的XRD分析中观察到了NaCl的衍射峰(图1(d))。这可能与Co3O4-NPs的磁性和导电性将DMEM培养基中的无机盐组分大量吸附在其表面有关。
纳米材料可以通过内吞作用或自由扩散等不同途径被细胞蓄积[26]。观察到将Co3O4-NPs分散在0.5% DMEM中后,明显出现团聚现象(图1)。一般而言,纳米颗粒的尺寸越小越容易进入细胞[27]。纳米颗粒在分散介质中的团聚明显会影响其进入细胞的能力。然而,也有报道指出“蛋白冠”的形成会为纳米颗粒表面提供新的生物结合位点。在生物系统内,纳米颗粒表面吸附的组分可为免疫系统提供特异性,从而使纳米颗粒顺利进入细胞[28]。多项研究证实纳米颗粒诱导的组蛋白H3修饰及DNA损伤与其细胞内蓄积量高度相关[29-30]。这与我们观察到的结果一致,随着Co3O4-NPs暴露剂量的增加,Co3O4-NPs的细胞内蓄积量及其诱导的组蛋白H3修饰及γ-H2AX显著增加(图3(a)、图4及图6),进一步分析表明,Co3O4-NPs的细胞内蓄积量与组蛋白H3修饰及γ-H2AX形成高度相关(图5及图7(a)),间接证明了Co3O4-NPs的细胞内蓄积是引起组蛋白修饰的关键因素之一。
组蛋白修饰在控制染色质结构与调控基因转录中发挥着重要作用[15-16]。我们的实验结果表明,Co3O4-NPs能诱导p-H3S10的持续高表达(图3)。然而,p-H3S10在Co3O4-NPs暴露后立即被诱导,这与有丝分裂相关的p-H3S10存在明显差异。另一方面,p-H3S10的高表达也是诱导原癌基因表达的关键因素之一[31-33]。一些致癌性金属,如镍、砷等能引起p-H3S10修饰的改变,从而导致基因表达的改变[32-33]。p-H3S10的增加会促进Ac-H3K14修饰水平上调[34-35]。Ac-H3K9及Ac-H3K14主要位于启动子区域,其高表达通常与基因激活相关[36]。在相同暴露时间点下我们观察到p-H3S10与Ac-H3K9及Ac-H3K14存在协同作用(图3(b))。另一方面,Me3-H3K4与组蛋白H3的乙酰化之间也存在协同作用[37],而Me3-H3K27是异染色质区域的主要标志,其高表达常常被认为是转录抑制的关键[19],这与我们观察到的Co3O4-NPs诱导的组蛋白H3修饰变化结果高度一致。这些结果强烈表明,Co3O4-NPs诱导的p-H3S10与基因激活高度相关。
另一方面,组蛋白修饰变化在DNA损伤修复过程中具有重要作用[17-18]。组蛋白H3乙酰化创造了开放的染色质结构,为募集DNA修复蛋白提供了良好的环境[17]。DNA损伤也可以引起相关修饰酶的变化,进而对组蛋白修饰产生影响。研究表明,甲基转移酶SUV39H1可被迅速募集到DNA损伤区域并引起H3K9甲基化的迅速上调,进而激活DNA损伤信号蛋白[38]。我们的分析结果表明,Co3O4-NPs引起的DNA损伤(γ-H2AX)与其诱导的组蛋白H3修饰变化强度呈线性相关(图7(b)、(c)和(d))。这些结果间接证明了Co3O4-NPs引起的DNA损伤是其诱导组蛋白H3修饰变化的原因之一。DNA损伤修复相关的组蛋白修饰不局限于一种组蛋白或一种修饰[39]。许多甲基转移酶和去甲基化酶被招募至DNA损伤位点,作用相应修饰的染色质。然而Co3O4-NPs暴露后,我们仅观察到LSD1随暴露时间的增加而下降(图8)。今后,我们将继续深入探讨其内在的分子机制。
组蛋白修饰是表观遗传学领域重要的研究内容之一,参与染色质动态结构的维持和调节、基因激活及沉默、DNA损伤修复等多种重要的生物学过程,与神经系统疾病、糖尿病和癌症等各类疾病有关[15-19]。对Co3O4-NPs等金属氧化物纳米材料诱导的组蛋白修饰的分子机制的进一步探究,必将为推动纳米安全性评价系统的发展与完善,为评估金属氧化物纳米材料的生物效应和安全性及设计应用于各种领域的金属氧化物纳米材料提供理论基础。
通信作者简介:赵晓旭(1984—),男,博士,副教授,主要研究方向为环境毒理学。
共同通信作者简介:吕源财(1986—),男,博士,副教授,主要研究方向为纳米材料的环境风险。
[1] ![]() N Z, Plata D L. Engineered nanomaterials in the context of global element cycles [J]. Environmental Science: Nano, 2019, 6(9): 2697-2711
N Z, Plata D L. Engineered nanomaterials in the context of global element cycles [J]. Environmental Science: Nano, 2019, 6(9): 2697-2711
[2] Aitken R J, Chaudhry M Q, Boxall A B, et al. Manufacture and use of nanomaterials: Current status in the UK and global trends [J]. Occupational Medicine, 2006, 56(5): 300-306
[3] Li W Y, Xu L N, Chen J. Co3O4 nanomaterials in lithium-ion batteries and gas sensors [J]. Advanced Functional Materials, 2005, 15(5): 851-857
[4] Li Y G, Tan B, Wu Y Y. Mesoporous Co3O4 nanowire arrays for lithium ion batteries with high capacity and rate capability [J]. Nano Letters, 2008, 8(1): 265-270
[5] Hoseinzadeh E, Makhdoumi P, Taha P, et al. A review on nano-antimicrobials: Metal nanoparticles, methods and mechanisms [J]. Current Drug Metabolism, 2017, 18(2): 120-128
[6] Tonelli A M, Venturini J, Arcaro S, et al. Novel core-shell nanocomposites based on TiO2-covered magnetic Co3O4 for biomedical applications [J]. Journal of Biomedical Materials Research Part B, Applied Biomaterials, 2020, 108(5): 1879-1887
[7] Pankhurst Q A, Thanh N K T, Jones S K, et al. Progress in applications of magnetic nanoparticles in biomedicine [J]. Journal of Physics D: Applied Physics, 2009, 42(22): 224001
[8] Zhao F, Zhao Y, Liu Y, et al. Cellular uptake, intracellular trafficking, and cytotoxicity of nanomaterials [J]. Small, 2011, 7(10): 1322-1337
[9] Sengul A B, Asmatulu E. Toxicity of metal and metal oxide nanoparticles: A review [J]. Environmental Chemistry Letters, 2020, 18(5): 1659-1683
[10] Liu N, Tang M. Toxic effects and involved molecular pathways of nanoparticles on cells and subcellular organelles [J]. Journal of Applied Toxicology, 2020, 40(1): 16-36
[11] McShan D, Ray P C, Yu H T. Molecular toxicity mechanism of nanosilver [J]. Journal of Food and Drug Analysis, 2014, 22(1): 116-127
[12] Wong B S E, Hu Q D, Baeg G H. Epigenetic modulations in nanoparticle-mediated toxicity [J]. Food and Chemical Toxicology, 2017, 109: 746-752
[13] Stoccoro A, Karlsson H L, Coppedè F, et al. Epigenetic effects of nano-sized materials [J]. Toxicology, 2013, 313(1): 3-14
[14] Shyamasundar S, Ng C T, Yung L Y, et al. Epigenetic mechanisms in nanomaterial-induced toxicity [J]. Epigenomics, 2015, 7(3): 395-411
[15] Berger S L. The complex language of chromatin regulation during transcription [J]. Nature, 2007, 447(7143): 407-412
[16] Kouzarides T. Chromatin modifications and their function [J]. Cell, 2007, 128(4): 693-705
[17] Price B D, D’Andrea A D. Chromatin remodeling at DNA double-strand breaks [J]. Cell, 2013, 152(6): 1344-1354
[18] Wang S Y, Meyer D H, Schumacher B. H3K4me2 regulates the recovery of protein biosynthesis and homeostasis following DNA damage [J]. Nature Structural &Molecular Biology, 2020, 27(12): 1165-1177
[19] Lawrence M, Daujat S, Schneider R. Lateral thinking: How histone modifications regulate gene expression [J]. Trends in Genetics, 2016, 32(1): 42-56
[20] Zhao X X, Ibuki Y. Evaluating the toxicity of silver nanoparticles by detecting phosphorylation of histone H3 in combination with flow cytometry side-scattered light [J]. Environmental Science &Technology, 2015, 49(8): 5003-5012
[21] Zhao X X, Toyooka T, Ibuki Y. Silver nanoparticle-induced phosphorylation of histone H3 at serine 10 is due to dynamic changes in actin filaments and the activation of Aurora kinases [J]. Toxicology Letters, 2017, 276: 39-47
[22] Liu Y, Mayo M W, Nagji A S, et al. BRMS1 suppresses lung cancer metastases through an E3 ligase function on histone acetyltransferase p300 [J]. Cancer Research, 2013, 73(4): 1308-1317
[23] Jin K L, Pak J H, Park J Y, et al. Expression profile of histone deacetylases 1, 2 and 3 in ovarian cancer tissues [J]. Journal of Gynecologic Oncology, 2008, 19(3): 185-190
[24] Kumar A, Kumari N, Nallabelli N, et al. Expression profile of H3K4 demethylases with their clinical and pathological correlation in patients with clear cell renal cell carcinoma [J]. Gene, 2020, 739: 144498
[25] de la Cruz X, Lois S, Sánchez-Molina S, et al. Do protein motifs read the histone code? [J]. BioEssays, 2005, 27(2): 164-175
[26] Oh N, Park J H. Endocytosis and exocytosis of nanoparticles in mammalian cells [J]. International Journal of Nanomedicine, 2014, 9(Suppl.1): 51-63
[27] Liu W, Wu Y, Wang C, et al. Impact of silver nanoparticles on human cells: Effect of particle size [J]. Nanotoxicology, 2010, 4(3): 319-330
[28] Donaldson K, Aitken R, Tran L, et al. Carbon nanotubes: A review of their properties in relation to pulmonary toxicology and workplace safety [J]. Toxicological Sciences: An Official Journal of the Society of Toxicology, 2006, 92(1): 5-22
[29] Abudayyak M, Gurkaynak T A, Özhan G. In vitro toxicological assessment of cobalt ferrite nanoparticles in several mammalian cell types [J]. Biological Trace Element Research, 2017, 175(2): 458-465
[30] Abudayyak M, Gurkaynak T A, Özhan G. In vitro evaluation of cobalt oxide nanoparticle-induced toxicity [J]. Toxicology and Industrial Health, 2017, 33(8): 646-654
[31] Rossetto D, Avvakumov N, C té J. Histone phosphorylation: A chromatin modification involved in diverse nuclear events [J]. Epigenetics, 2012, 7(10): 1098-1108
té J. Histone phosphorylation: A chromatin modification involved in diverse nuclear events [J]. Epigenetics, 2012, 7(10): 1098-1108
[32] Li J, Gorospe M, Barnes J, et al. Tumor promoter arsenite stimulates histone H3 phosphoacetylation of proto-oncogenes c-fos and c-Jun chromatin in human diploid fibroblasts [J]. The Journal of Biological Chemistry, 2003, 278(15): 13183-13191
[33] Ke Q D, Li Q, Ellen T P, et al. Nickel compounds induce phosphorylation of histone H3 at serine 10 by activating JNK-MAPK pathway [J]. Carcinogenesis, 2008, 29(6): 1276-1281
[34] Lo W S, Trievel R C, Rojas J R, et al. Phosphorylation of serine 10 in histone H3 is functionally linked in vitro and in vivo to Gcn5-mediated acetylation at lysine 14 [J]. Molecular Cell, 2000, 5(6): 917-926
[35] Salvador L M, Park Y, Cottom J, et al. Follicle-stimulating hormone stimulates protein kinase A-mediated histone H3 phosphorylation and acetylation leading to select gene activation in ovarian granulosa cells [J]. The Journal of Biological Chemistry, 2001, 276(43): 40146-40155
[36] Karmodiya K, Krebs A R, Oulad-Abdelghani M, et al. H3K9 and H3K14 acetylation co-occur at many gene regulatory elements, while H3K14ac marks a subset of inactive inducible promoters in mouse embryonic stem cells [J]. BMC Genomics, 2012, 13: 424
[37] Zhang T Y, Cooper S, Brockdorff N. The interplay of histone modifications - writers that read [J]. EMBO Reports, 2015, 16(11): 1467-1481
[38] Ayrapetov M K, Gursoy-Yuzugullu O, Xu C, et al. DNA double-strand breaks promote methylation of histone H3 on lysine 9 and transient formation of repressive chromatin [J]. Proceedings of the National Academy of Sciences of the United States of America, 2014, 111(25): 9169-9174
[39] van Attikum H, Gasser S M. Crosstalk between histone modifications during the DNA damage response [J]. Trends in Cell Biology, 2009, 19(5): 207-217