近20多年来,在气候变化和人类活动的多重压力下,内陆水体蓝藻水华发生频率和强度呈快速增加趋势,特别是产毒水华蓝藻严重威胁水生态系统健康和饮用水安全[1]。除了在浅水湖泊常见的微囊藻水华之外,新型水华蓝藻拉氏尖头藻(Raphidiopsis raciborskii)由于其高毒性、暴发性和入侵性日益受到关注[2-3]。拉氏尖头藻曾被称为拉氏拟柱胞藻(Cylindrospermopsis raciborskii),由于其细胞形态和16S rRNA基因序列与尖头藻高度相似,因此分类学者目前在蓝藻分类系统中将拟柱胞藻属和尖头藻属统一合并为尖头藻属[4]。
拉氏尖头藻隶属蓝藻门、念珠藻目、束丝藻科[5],包括产毒株和无毒株,两者在形态和16S rRNA基因方面难以区分,但是产拟柱胞藻毒素的藻株含有完整的拟柱胞藻毒素合成基因[6-7]。在同一水体分离到的拉氏尖头藻细胞和藻丝体长度和宽度可能具有差异[8],拉氏尖头藻产毒藻株和无毒藻株可同时共存于相同水体中[9-10]。目前,利用分子标记和产毒基因可以进行拉氏尖头藻分析,系统发育分析常用16S rRNA基因、rpoC1基因、16S~23S转录间隔区(ITS)等序列[11-12],其中16S rRNA基因测序能够有效地区分蓝藻属种间和种内的变异[13-14]。此外,基于DNA序列的产毒蓝藻PCR检测方法已被成功应用于环境监测,可以通过扩增毒素合成基因来实现,例如微囊藻毒素和拟柱胞藻毒素(CYNs)合成基因检测[15-16]。其中,cyrJ基因是能够进行CYNs生物合成的蓝藻所特有的[6]。CYNs合成基因簇包括编码氨基转移酶的cyrA、一个混合聚酮合成酶/非核糖体肽合成酶(PKS/NRPS)模块(cyrB)、4个PKS(cyrC、cyrD、cyrE和cyrF)模块和剪切酶等[6]。拟柱胞藻毒素是一种三环胍类生物碱,已报道5种异构体,其中拟柱胞藻毒素(CYN)和脱氧拟柱胞藻毒素(deoxy-CYN)最为常见[17]。
拉氏尖头藻常见于热带深水水体,能够耐受不利环境条件如低光照强度和低营养水平,目前CYNs是全球第二常见的蓝藻毒素[7,18-20]。有研究者将其广泛分布归因于其较高的表型可塑性、产生化感物质和气候变化等[21-24]。从21世纪初开始,越来越多的证据显示拉氏尖头藻呈现出从热带逐渐向亚热带和温带地区扩散的趋势[2-3]。在我国,尤其在亚热带地区深水水库拉氏尖头藻水华发生频率逐渐增加,近年来已逐渐成为我国东南地区水库水华蓝藻优势种群[25-26]。国内学者虽然已经成功分离多株拉氏尖头藻藻株,但是产毒藻株较为稀缺。目前仅在浙江省杭州市湘湖分离3株产CYN藻株,广东省千灯湖分离1株产deoxy-CYN藻株[27-29]。
本研究从福建省厦门市石兜水库分离到4株拉氏尖头藻,通过形态特征观察、16S rRNA基因和cyr产毒基因序列分析,结合ELISA和LC-MS/MS化学检测确定分离到的藻株均为产毒拉氏尖头藻,研究结果有助于深入理解产毒拉氏尖头藻的地理分布与扩散规律,可为新型有害蓝藻监测预警和水华防控研究提供基础数据。
1 材料与方法(Materials and methods)
1.1 藻种的分离鉴定和培养(Isolation,identification and culture of Raphidiopsis raciborskii)
石兜水库位于福建省厦门市苎溪上游(图1),1959年10月建成。本研究中所用的拉氏尖头藻均采集、分离自石兜水库(24°42' N,118°00'E),拉氏尖头藻纯化培养后,保种培养于中国科学院城市环境研究所水生态健康研究组。石兜水库库容为6.14×107 m3,目前为厦门市主要的饮用水水源之一[30]。厦门市属于亚热带季风性湿润气候区,多年平均气温和降水量分别为20.7 ℃和1 335.8 mm。水生态健康研究组于2010年1月在石兜水库发现藻类水华,鉴定水华种类为拉氏尖头藻(亦称拉氏拟柱胞藻),随后对石兜水库进行长期连续定位监测,发现拉氏尖头藻水华具有年际演替性[31],特别是在厦门水库枯水年份(2009—2010年、2014—2015年、2017—2018年和2020年)拉氏尖头藻均为绝对优势藻类,并形成不同程度的蓝藻水华。
2018年4月,采集石兜水库表层0.5 m的原水,在培养皿中利用毛细管挑取单藻丝体进行培养(图1)。首先,在200倍倒置光学显微镜下观察拉氏尖头藻形态,用毛细管吸取单藻丝体,在预先准备的超纯水中反复冲洗,至少冲洗5~6次,去除藻细胞表面附带的杂物异物;其次,将洗涤后的藻丝体转移至含有200 μL BG11培养基的96孔培养板中,放置于光照培养箱(宁波赛福公司,PG250)进行培养。设置培养温度为(27±1) ℃,光照强度为10 μmol·m-2·s-1,光照周期为12 h∶12 h;最后,每2~3 d观察一次培养的拉氏尖头藻,2~3周后再次镜检进行物种鉴定,将生长良好的藻株转移至50 mL锥形瓶进行扩大培养。本研究中,共分离培养出4株生长良好的拉氏尖头藻(图1),命名为R. raciborskii XM1~XM4。

图1 石兜水库地理位置(a)和拉氏尖头藻形态特征(b)
注:红色箭头指向XM1和XM2的异形胞。
Fig.1 The location of Shidou Reservoir (a) and photographs of Raphidiopsis raciborskii showing the morphological characteristics of four strains (b)
Note:Red arrows indicate heterocysts in XM1 and XM2 strains.
用于实验研究的藻株均同时接种于150 mL BG11培养基中,当藻株处于对数生长后期时(细胞密度约为108 cell·L-1),同时对4株藻株取样后进行各项指标测定分析,以确保实验过程中的一致性。
1.2 拟柱胞藻毒素分析(Cylindrospermopsin analysis)
首先使用Abraxis酶联免疫吸附分析(ELISA)试剂盒(美国Abraxis,#522011)测定CYNs总浓度。将培养至对数生长后期的4株拉氏尖头藻培养液分别取出2 mL,置于5 mL离心管中利用超声波破碎仪裂解藻细胞,将裂解后的藻液离心后收集上清液,4 ℃保存备用、测定。按照说明书测定CYNs浓度,为防止毒素浓度过高,对上清液10倍稀释后进行测定,每个样品测定3次重复。
使用LC-MS/MS(日本岛津,AB SCIEX API 3200TM)对拉氏尖头藻藻液中CYNs进行测定分析。将培养至对数生长后期的4株拉氏尖头藻培养液分别取出5 mL,超声波细胞粉碎仪进行细胞破碎,使用0.45 μm滤膜(Acrodisc® Syringe Filter)过滤后-20 ℃保存备用。随后,使用AB SCIEX API 3200TM LC-MS/MS系统进行毒素测定,离子源为ESI,采用多反应监测(MRM)正离子模式。CYN扫描MRM定量离子对为416.1/194.3,deoxy-CYN为400/194.3。液相色谱采用梯度模式洗脱,流动相为5% (V∶V)乙腈,色谱柱为Agilent Poroshell 120 EC-C18(4.6 mm×50 mm,2.7 μm),柱温为40 ℃,进样量为20 μL,流速为0.6 mL·min-1。CYN和deoxy-CYN的定量均根据CYN的标准曲线进行计算。通过将CYNs浓度(μg·L-1)除以细胞丰度(cell·L-1),计算CYNs细胞配额,最后将结果乘以106将μg·cell-1转化为pg·cell-1 [32]。
1.3 DNA提取、PCR扩增和测序(DNA extraction,PCR amplification and sequencing)
在拉氏尖头藻的对数生长后期分别吸取纯培养液15 mL,使用0.22 μm聚碳酸酯滤膜(直径47 mm)过滤收集藻细胞,使用超纯水进行重悬浮和过滤,洗涤5~6次,将滤膜在无菌条件下剪至适当大小,按照说明书使用FastDNA提取试剂盒(MP Biomedicals)进行DNA提取。通过超微量分光光度计(Nanodrop 2.0)检测DNA的浓度和质量。
利用细菌16S rRNA基因通用引物27F(5’-AGAGTTTGATCCTGGCTCAG-3’)和1492R(5’-TACGGTTACCTTGTTACGACTT-3’)[33],对4株拉氏尖头藻基因组进行PCR扩增。PCR扩增程序为预变性95 ℃、2 min;循环扩增35次:95 ℃、30 s,55 ℃、30 s,72 ℃、60 s;随后延伸72 ℃、10 min;最后4 ℃保温。将PCR扩增产物进行2%的琼脂糖凝胶电泳,切取纯化单一明亮的目标条带,进行大肠杆菌连接、转化和涂平板,挑取单克隆进行摇菌和DNA测序。测序成功的单克隆DNA序列在BioEdit软件(美国Borland)中进行人工质量确认和16S rDNA序列拼接,在NCBI中进行BLAST相似性分析。
1.4 系统发育分析(Phylogenetic analysis)
从4株拉氏尖头藻中成功获得14条16S rRNA基因克隆序列(表1),结合GenBank数据库中来自不同国家的166株拉氏尖头藻和10株外类群蓝藻的基因序列构建系统发育树。将190条16S rRNA基因序列使用MUSCLE软件进行序列比对[34],然后利用trimAl软件[35]进行低质量以及高变异度序列的过滤和修剪,最终用于构建系统树的DNA序列长度为1 004 bp。使用IQ-TREE最大似然法(ML)构建系统树[36],所使用的最优模型为HKY+F+R2[37]。为进一步验证系统进化树的可靠性,同时使用MEGA X软件构建邻接关系(NJ)系统发育树[38]。对系统发育树的置信度进行1 000次重复抽样(Bootstrap)验证。最后在MEGA X中编辑系统发育树。
表1 厦门石兜水库拉氏尖头藻藻株16S rRNA基因序列GenBank登录号
Table 1 GenBank accession numbers for 16S rRNA gene sequence of Raphidiopsis raciborskii strains from Shidou Reservoir in Xiamen

藻株名称Name of strains藻株克隆Clone names of strainsGenBank登录号GenBank accession numbersR.raciborskii XM1R.raciborskii XM1aMZ536613R.raciborskii XM1bMZ536614R.raciborskii XM1cMZ536615R.raciborskii XM2R.raciborskii XM2aMZ536616R.raciborskii XM2bMZ536617R.raciborskii XM2cMZ536618R.raciborskii XM3R.raciborskii XM3aMZ536619R.raciborskii XM3bMZ536620R.raciborskii XM3cMZ536621R.raciborskii XM4R.raciborskii XM4aMZ536622R.raciborskii XM4bMZ536623R.raciborskii XM4cMZ536624R.raciborskii XM4dMZ536625R.raciborskii XM4eMZ536626
1.5 基因组Survey测序(Genome survey sequencing)
将检测质量和浓度合格的R. raciborskii XM2基因组DNA构建小片段文库,通过Illumina HiSeq平台进行PE150测序。将序列数据使用Trimmomatic软件进行质量过滤[39],去除低质量的序列,再使用SOAPdenovo软件[40]依次选择21~81的kmer值进行基因组拼接,最后基于N50值确定最优结果。选择GenBank中R. raciborskii AWT205的CYN合成基因簇为参考序列,使用本地BLAST通过序列比较获取藻株的CYN合成基因簇。R. raciborskii XM2基因组序列已经提交至NCBI数据库,BioProject为PRJNA796232,登录号为SRR1757-4388。
2 结果(Results)
2.1 拉氏尖头藻形态与产毒特征(Morphological and toxic characteristics of Raphidiopsis raciborskii)
经显微镜鉴定,在厦门石兜水库所分离的4株藻株均为拉氏尖头藻(图1),分别将其命名为R. raciborskii XM1、XM2、XM3和XM4。对4株拉氏尖头藻的16S rRNA基因进行克隆分析,共获得14条序列(表1),同已知的拉氏尖头藻16S rRNA基因相比,序列相似性为99.8%~100%,表明本研究分离的4株藻株均为拉氏尖头藻。
形态上,4株拉氏尖头藻藻丝体均为直线型、不弯曲(图1(b)),无鞘、具有气囊,藻丝体平均长度分别为(199.22±11.57) μm、(168.31±6.57) μm、(191.90±12.83) μm和(122.23±3.50) μm(n=100)。藻细胞为圆柱形或圆筒形,长大于宽,4个藻株的细胞平均长度为(4.26±0.32) μm、(4.25±0.21) μm、(4.10±0.24) μm和(3.79±0.19) μm(n=20)。分离培养初期,藻丝体顶端生有异形胞,异形胞呈卵形或者水滴形,有时略弯曲,具单孔。在富含氮条件培养过程中随着藻丝的生长,异形胞消失、顶端变尖细;缺氮培养处理后,藻丝体顶端异形胞会重新出现(图1(b))。藻丝体细胞横壁明显、常收缢,藻丝体末端细胞呈圆锥形,圆锥体顶部较尖,少数藻丝体较钝。此外,在培养过程中可观察到藻丝体的厚壁孢子,为亚端生。
ELISA检测结果显示,XM1、XM2、XM3和XM4拟柱胞藻毒素均为阳性(表2)。LC-MS/MS检测结果显示,4株藻株能够同时合成CYN和deoxy-CYN(表2);其中XM1、XM2和XM3主要合成CYN,CYN平均浓度分别为0.87、1.02和0.71 pg·cell-1,deoxy-CYN平均浓度分别为0.15、0.16和0.13 pg·cell-1;XM4主要合成deoxy-CYN,CYN浓度为0.03 pg·cell-1,deoxy-CYN浓度为0.42 pg·cell-1。
表2 ELISA和LC-MS/MS方法测定拉氏尖头藻藻株的拟柱胞藻毒素(CYNs)
Table 2 The cylindrospermopsins (CYNs) in four Raphidiopsis raciborskii strains determined by ELISA and LC-MS/MS

藻株名称Name of strainsELISALC-MS/MSCYNsCYN/(pg·cell-1)deoxy-CYN/(pg·cell-1)R. raciborskii XM1+0.87±0.150.15±0.02R.raciborskii XM2+1.02±0.100.16±0.02R.raciborskii XM3+0.71±0.060.13±0.01R. raciborskii XM4+0.03±0.020.42±0.06
注:+代表拟柱胞藻毒素检测呈阳性。
Note:+ indicates test positive for CYNs.
2.2 系统发育树分析(Phylogenetic tree analysis)
16S rRNA基因系统发育树显示,180株拉氏尖头藻形成四大进化枝(图2):非洲类群枝(MJ树Bootstrap值为22%,n=10)、亚洲和欧洲类群枝(MJ树Bootstrap值为45%,n=106)、大洋洲类群枝(主要为澳大利亚藻株)(MJ树Bootstrap值为32%,n=28)和美洲类群枝(美国和巴西的藻株)(MJ树Bootstrap值为18%,n=36)。
有趣的是,厦门分离的4株拉氏尖头藻分别位于不同的进化枝(图2)。其中,XM2藻株16S rRNA基因序列与非洲藻株遗传距离最近;另外3个藻株(XM1、XM3和XM4)位于亚洲/欧洲类群分枝。XM1、XM3和XM4藻株基因序列与已报道中国杭州藻株、泰国藻株、日本藻株和德国藻株遗传距离较近。
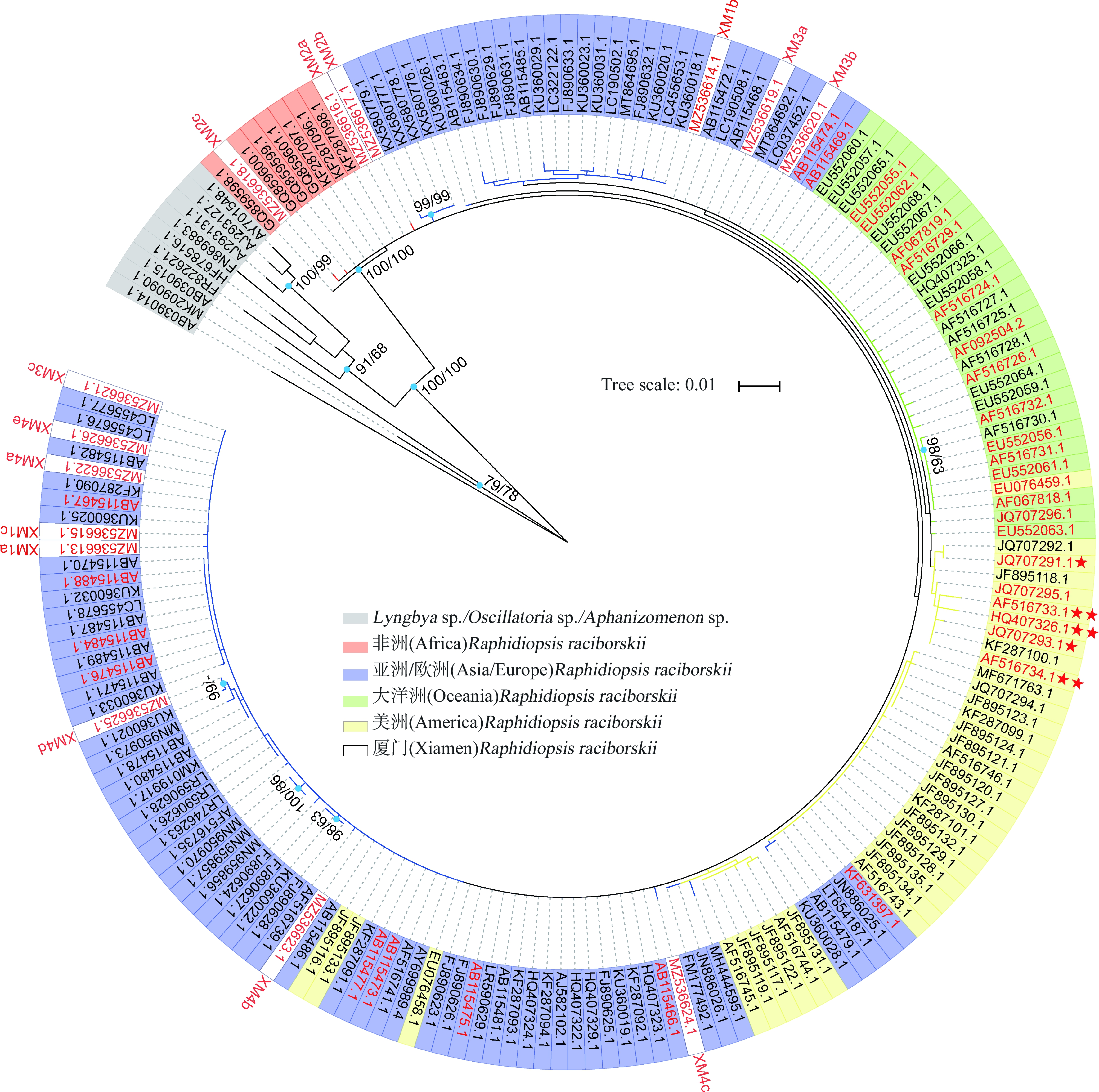
图2 基于180个Raphidiopsis raciborskii藻株和10个外类群蓝藻物种的16S rRNA基因序列构建的系统发育树
注:节点上分别为ML和NJ的Bootstrap值,红色字体不标星表示产拟柱胞藻毒素株,红色字体标单星(★)表示产蛤蚌毒素株,红色字体标双星(★★)表示产麻痹性贝类毒素株;发育树中<60的支持值未标注。
Fig.2 Phylogenetic tree inferred from 180 Raphidiopsis raciborskii strains (clones) and 10 cyanobacterial species strains based on 16S rRNA gene
Note:The nodes are indicated by ML and NJ Bootstrap values;red color without star indicates CYN-producing strain,red color with one star (★) indicates saxitoxin-producing strain,and red color with double-star (★★) indicates paralytic shellfish poisoning-producing strain;for simplicity,note whose support values below 60 are not indicated in the tree node.
2.3 拉氏尖头藻cyr基因簇(cyr gene cluster of Raphidiopsis raciborskii)
选取R. raciborskii XM2进行基因组测序后,对cyr基因簇进行组装、分析和注释,其完整长度约为38 kb(图3和表3)。XM2藻株cyr基因簇包含12个cyr基因。基因序列与拉氏尖头藻AWT205、CHAB3438和CHAB358藻株同源性较高(98.97%~99.84%);而且cyr簇中基因排列比较显示,不同藻株的cyr基因序列较为保守,但排列模式各不相同。相比而言,XM2藻株的cyrB基因序列被分割成2部分,cyrI基因被分割为3部分;XM2藻株cyr基因簇中缺少cyrL、cyrN和cyrO基因(图3和表3)。
表3 拉氏尖头藻XM2藻株cyr基因簇注释
Table 3 The annotation of the genes of the cyr cluster from Raphidiopsis raciborskii XM2
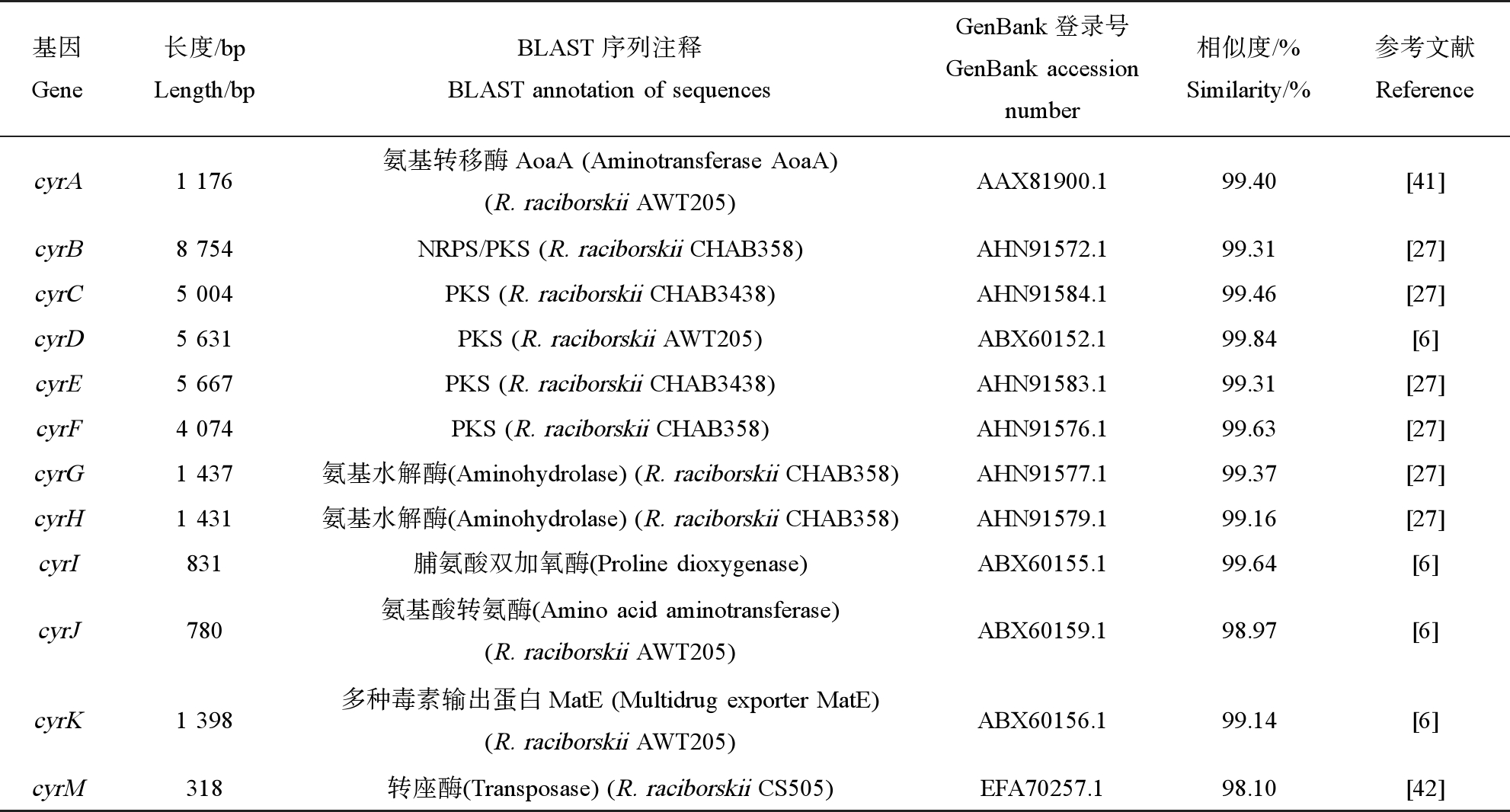
基因Gene长度/bpLength/bpBLAST序列注释BLAST annotation of sequencesGenBank登录号GenBank accession number相似度/%Similarity/%参考文献ReferencecyrA1 176氨基转移酶AoaA (Aminotransferase AoaA) (R. raciborskii AWT205)AAX81900.199.40[41]cyrB8 754NRPS/PKS (R. raciborskii CHAB358)AHN91572.199.31[27]cyrC5 004PKS (R. raciborskii CHAB3438)AHN91584.199.46[27]cyrD5 631PKS (R. raciborskii AWT205)ABX60152.199.84[6]cyrE5 667PKS (R. raciborskii CHAB3438)AHN91583.199.31[27]cyrF4 074PKS (R. raciborskii CHAB358)AHN91576.199.63[27]cyrG1 437氨基水解酶(Aminohydrolase) (R. raciborskii CHAB358)AHN91577.199.37[27]cyrH1 431氨基水解酶(Aminohydrolase) (R. raciborskii CHAB358)AHN91579.199.16[27]cyrI831脯氨酸双加氧酶(Proline dioxygenase)ABX60155.199.64[6]cyrJ780氨基酸转氨酶(Amino acid aminotransferase) (R. raciborskii AWT205)ABX60159.198.97[6]cyrK1 398多种毒素输出蛋白MatE (Multidrug exporter MatE) (R. raciborskii AWT205)ABX60156.199.14[6]cyrM318转座酶(Transposase) (R. raciborskii CS505)EFA70257.198.10[42]
注:NRPS,非核糖体肽合成酶;PKS,聚酮合成酶。
Note:NRPS,nonribosomal peptide synthetase;PKS,polyketide synthase.
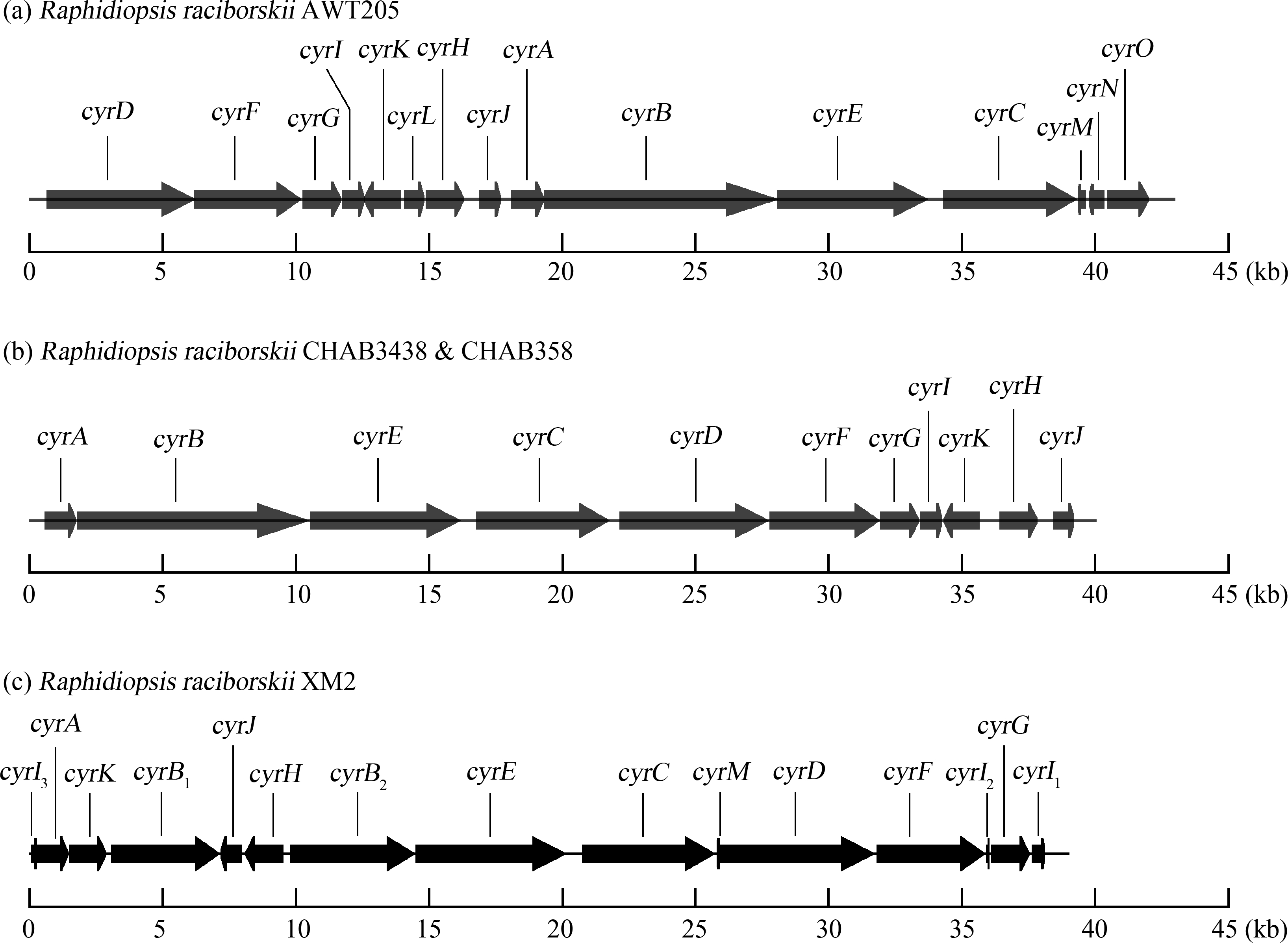
图3 Raphidiopsis raciborskii AWT205(澳大利亚藻株)[6]、CHAB3438/CHAB358(浙江藻株)[25]与XM2(福建藻株)的cyr基因簇基因结构比较
Fig. 3 Comparison of the gene organizations of cyr gene clusters from R. raciborskii AWT205 (Australia) [6],CHAB3438/CHAB358 (Zhejiang Province,China) [25] and XM2 (Fujian Province,China)
3 讨论(Discussion)
本研究首次在福建省分离到4株产毒拉氏尖头藻,通过CYNs合成基因分析和毒素测定证实了其具有产生CYNs的能力。厦门石兜水库产毒拉氏尖头藻的细胞形态基本相同,但是跟其他地区不同藻株间的藻丝体形态特征具有一定的差异。厦门藻株平均细胞长度(3.79~4.26 μm)小于澳大利亚分离的拉氏尖头藻藻株细胞平均长度(5~10 μm)[10],但厦门藻株藻丝体平均长度(122~199 μm)长于广东千灯湖藻株(41~78 μm)[28]。
蓝藻的同一物种可能包含多种生态型,它们对环境变化的多样化响应导致藻株生态类型的差异[43]。拉氏尖头藻具有高度的环境适应性,在生理和形态方面可塑性较高[44],不同地理位置和不同水体生境可能会造成拉氏尖头藻生态类型的变化。福建厦门拉氏尖头藻藻株具有异形胞,在富含氮的条件下培养一段时间后消失,在澳大利亚藻株和泰国藻株中同样发现在氮源充足条件下异形胞消失的概率增加[13,45]。异形胞主要功能为固氮,当水体氮源充足时,异形胞分化基因可能不表达[3,46]。即使在4株厦门藻株藻丝体形态之间也有轻微的形态差异,这表明在种群和个体水平上藻株也存在形态多样性。
本研究同时使用ELISA试剂盒和LC-MS/MS方法对拟柱胞藻毒素进行定量和定性分析,确定所分离的4株拉氏尖头藻均为产毒藻株。其中,XM1、XM2和XM3藻株所产毒素以CYN为主,在拉氏尖头藻对数生长后期培养液中浓度为0.71~1.02 pg·cell-1;XM4则以deoxy-CYN为主,浓度为0.42 pg·cell-1。目前,我国东南地区水库拉氏尖头藻的检出率和检出地区急剧增加,在研究人员分离的上百株藻株中仅报道4株是产毒株:2012年和2020年浙江省杭州市湘湖分别分离到2株产CYN藻株和1株产CYN藻株,产毒量为1 268~2 637 μg·g-1 [27,29];2017年在广东省千灯湖分离到1株产deoxy-CYN藻株,产毒量为1 745.19 μg·L-1 [28]。遗憾的是,对我国内陆水体尖头藻的多数研究中并没有分析检测CYNs,本研究将我国已知的产毒拉氏尖头藻纯培养藻株扩大到8株。因此,未来的研究中不仅需要关注拉氏尖头藻水华,更应当重视研究拉氏尖头藻产毒特性和机理。
早期研究基于16S rRNA基因序列分析可以大致区分来自不同地理区域的拉氏尖头藻藻株[14],有的研究则将16S rRNA基因与ITS、rpoC1等基因结合,在更高的分辨率层面评估五大洲拉氏尖头藻的遗传变异与种群分化[47]。本研究从16S rRNA基因来看,XM1、XM3和XM4藻株汇聚到亚洲/欧洲进化枝,亚洲和欧洲拉氏尖头藻藻株之间具有更高的同源性[3,46],然而大洋洲和美洲的藻株将亚欧进化分枝分为2部分;值得注意的是,XM2藻株与非洲藻株同源性较高。这说明厦门石兜水库中的拉氏尖头藻可能存在不同的基因型,遗传多样性较高。我们认为可能的解释有3种:扩散假说、分化假说和扩散分化混合假说。扩散假说可以理解为,石兜水库不同基因型的拉氏尖头藻分别来源于不同地方,共存于石兜水库水体;分化假说可以理解为,石兜水库不同基因型的拉氏尖头藻具有同一个来源,是在中国福建当地遗传分化的结果;混合假说可以理解为拉氏尖头藻物种扩散和遗传分化同时发生的结果,并不能用单一的扩散过程或分化过程解释。
毫无疑问,拉氏尖头藻遗传多样性是长期演化的产物,较高的遗传多样性可能意味着较快的演化速率。在扩散过程中的演化导致了基因型的多样化,其扩散过程可能是不同环境需求对其基因型进行选择的过程[12]。国际上对于拉氏尖头藻的起源、地理分布和扩散具有不同的解释,Padisák[44]提出拉氏尖头藻起源于非洲,随后扩散到赤道地区和美洲中部、或者以澳大利亚为中心逐渐向热带、亚热带和温带地区扩散。有研究认为欧洲大陆藻株最有可能起源于亚洲和澳大利亚[3,14]。Gugger等[11]综合利用16S rRNA基因、ITS、ropC1和nifH基因分析了美洲、欧洲、非洲和澳大利亚的18株尖头藻,认为更新世冰期时代的干冷气候导致尖头藻在美洲和欧洲绝大多数地区灭绝,仅有南方少数温暖地区的尖头藻生存下来,当前美洲和欧洲地区尖头藻可能主要来自其南方温暖区域株系的扩散。仅仅从16S rRNA基因序列单基因分析,难以判断厦门拉氏尖头藻是否来自非洲或者澳大利亚的扩散。从产毒特性方面来看,由于目前在非洲未发现产CYNs藻株,因此在厦门发现的藻株更有可能是由于澳大利亚藻株的扩散所导致的结果。事实上,候鸟迁徙携带拉氏尖头藻,可能会导致藻株基因型之间的频繁交换。例如,候鸟在迁徙过程中,其足和喙能够携带拉氏尖头藻厚壁孢子[48],在全球候鸟迁飞区中,中国东部沿海地区位于东亚-澳大利西亚迁飞区,在该迁飞区内,每年有数以千万计的水鸟在我国东部沿海地区迁徙过境或中转停歇;涉及到亚洲与非洲的迁飞区是西亚-东非路线,因此很少有候鸟在非洲和东亚地区之间迁徙[49]。浙江、福建和广东均位于东亚-澳大利西亚候鸟迁飞区,在此迁徙过境和停歇的部分水鸟(特别是数量众多的鹬类)在澳大利亚越冬。因此推测近年在浙江和广东发现的产毒藻株可能与候鸟迁徙携带藻株的传播和扩散存在密切关系。此外,大陆之间往来的运输、船舶压载水、热带鱼的进口和科学样本的转移等也可能是重要的传播媒介[50]。总之,福建石兜水库修建于20世纪50年代末,至今已有60年的历史,石兜水库拉氏尖头藻的来源十分复杂,可能存在多种途径,目前的数据仍然缺乏对拉氏尖头藻地理分布、起源扩散的一致性解释,因此,未来需要全球科学家通力合作,综合野外观测、控制实验和模型模拟等方法进一步深入研究其遗传多样性与地理分布之间的关系,为这一新型水华蓝藻防控提供基础数据。
厦门拉氏尖头藻同其他大洲的产毒藻株与无毒藻株聚在一起,进一步说明基于16S rRNA基因难以区分产毒与无毒藻株,这也暗示CYNs合成基因可能会在藻株之间进行水平转移;或者,在一些拉氏尖头藻藻株中,由于基因突变和缺失、序列重复和转座子插入等造成CYNs生物合成基因簇中关键基因失活,进而丧失了产毒能力[17,27]。拉氏尖头藻XM2藻株的cyr基因簇中未发现cyrN和cyrO基因,这与在浙江杭州分离获得的CHAB3438/CHAB358藻株cyr基因簇一致[27]。虽然在澳大利亚AWT205藻株cyr基因簇中报道了这2个基因,并推测这2种基因可能在毒素合成的转录调控中具有一定的作用[6],但是由于这2种基因仅在cyr基因簇末端被发现,且不与基因簇中的其他基因相邻,所以可能不属于cyr基因的核心组分[51-52]。值得注意的是,XM2藻株中cyr基因发生重新排列,与杭州2个CHAB藻株的cyr基因簇排列最为相似,事实上这3株藻株分离地点最近,均位于中国东南。在XM2藻株cyr基因簇内呈现重排,可能是在DNA复制过程中发生了变异,导致基因结构发生了改变,使cyrK、cyrJ和cyrH基因从末端被移除,并插入了cyrB所在的位置。这不仅导致了cyr簇基因序列的重排,还导致了cyrB和cyrI基因的分割。另一种假设认为这些cyr簇基因在进化过程中是相关的,但可能存在不同的祖先[51]。
综上所述,福建省厦门市石兜水库分离到4株拉氏尖头藻,从细胞形态和16S rRNA基因序列方面都鉴定为拉氏尖头藻,不同藻株之间藻细胞相同、但是藻丝体略有差异,在富含氮源的培养条件下异形胞会消失。CYNs合成基因分析、CYNs化学测定表明4株拉氏尖头藻均可同时产生CYN和deoxy-CYN。16S rRNA基因系统发育分析表明厦门拉氏尖头藻种群遗传多样性较高,推测可能是扩散过程和分化过程同时发生的结果。此外,福建厦门产毒藻株CYN合成基因簇与浙江杭州产毒藻株基因簇最为相似、高度同源,推测这些产毒藻株可能来自澳大利亚藻株的扩散,与东亚-澳大利西亚迁飞区候鸟迁徙密切相关。未来的工作中,我们将结合多种标记基因和基因组分析对拉氏尖头藻地理种群的扩散分化进行深入的探讨。
拉氏尖头藻是一类新型水华蓝藻,已经在我国东南地区水库中广泛分布,能够在中营养水库形成蓝藻水华,但是尚未引起管理部门和公众的足够重视。在气候变暖背景下水库型饮用水水源地拉氏尖头藻监测、预警及其水华防控值得深入研究,特别应该加大投入支持研究中国水库拉氏尖头藻传播扩散的生态过程、生态机制与生态效应,进而保障饮用水安全。
[1] Paerl H W,Otten T G.Environmental science.Blooms bite the hand that feeds them [J].Science,2013,342(6157):433-434
[2] Antunes J T,Leão P N,Vasconcelos V M.Cylindrospermopsis raciborskii:Review of the distribution,phylogeography,and ecophysiology of a global invasive species [J].Frontiers in Microbiology,2015,6:473
[3] Panou M,Zervou S K,Kaloudis T,et al.A Greek Cylindrospermopsis raciborskii strain:Missing link in tropic invader’s phylogeography tale [J].Harmful Algae,2018,80:96-106
[4] Aguilera A,Gómez E B,![]() J,et al.The polyphasic analysis of two native Raphidiopsis isolates supports the unification of the genera Raphidiopsis and Cylindrospermopsis (Nostocales,Cyanobacteria) [J].Phycologia,2018,57(2):130-146
J,et al.The polyphasic analysis of two native Raphidiopsis isolates supports the unification of the genera Raphidiopsis and Cylindrospermopsis (Nostocales,Cyanobacteria) [J].Phycologia,2018,57(2):130-146
[5] Komarek J,Kastovsky J,Mares J,et al.Taxonomic classification of cyanoprokaryotes (cyanobacterial genera) 2014,using a polyphasic approach [J].Preslia,2014,86(4):295-335
[6] Mihali T K,Kellmann R,Muenchhoff J,et al.Characterization of the gene cluster responsible for cylindrospermopsin biosynthesis [J].Applied and Environmental Microbiology,2008,74(3):716-722
[7] Yang Y M,Yu G L,Chen Y X,et al.Four decades of progress in cylindrospermopsin research:The ins and outs of a potent cyanotoxin [J].Journal of Hazardous Materials,2021,406:124653
[8] Saker M L,Neilan B A,Griffiths D J.Two morphological forms of Cylindrospermopsis raciborskii (cyanobacteria) isolated from Solomon Dam,Palm Island,Queensland [J].Journal of Phycology,1999,35(3):599-606
[9] Burford M A,Davis T W,Orr P T,et al.Nutrient-related changes in the toxicity of field blooms of the cyanobacterium,Cylindrospermopsis raciborskii [J].FEMS Microbiology Ecology,2014,89(1):135-148
[10] Willis A,Chuang A W,Woodhouse J N,et al.Intraspecific variation in growth,morphology and toxin quotas for the cyanobacterium,Cylindrospermopsis raciborskii [J].Toxicon,2016,119:307-310
[11] Gugger M,Molica R,Le Berre B,et al.Genetic diversity of Cylindrospermopsis strains (cyanobacteria) isolated from four continents [J].Applied and Environmental Microbiology,2005,71(2):1097-1100
[12] Piccini C,Aubriot L,Fabre A,et al.Genetic and eco-physiological differences of South American Cylindrospermopsis raciborskii isolates support the hypothesis of multiple ecotypes [J].Harmful Algae,2011,10(6):644-653
[13] Saker M L,Neilan B A.Varied diazotrophies,morphologies,and toxicities of genetically similar isolates of Cylindrospermopsis raciborskii (Nostocales,Cyanophyceae) from Northern Australia [J].Applied and Environmental Microbiology,2001,67(4):1839-1845
[14] Moreira C,Fathalli A,Vasconcelos V,et al.Genetic diversity and structure of the invasive toxic cyanobacterium Cylindrospermopsis raciborskii [J].Current Microbiology,2011,62(5):1590-1595
[15] Hoff-Risseti C,Dörr F A,Schaker P D C,et al.Cylindrospermopsin and saxitoxin synthetase genes in Cylindrospermopsis raciborskii strains from Brazilian freshwater [J].PLoS One,2013,8(8):e74238
[16] Adriana S L,Mathias A C,Viviane P S,et al.Microcystins and cylindrospermopsins molecular markers for the detection of toxic cyanobacteria:A case study of northeastern Brazilian reservoirs [J].Limnetica,2015(34):269-282
[17] Rzymski P,Poniedzia ek B.In search of environmental role of cylindrospermopsin:A review on global distribution and ecology of its producers [J].Water Research,2014,66:320-337
ek B.In search of environmental role of cylindrospermopsin:A review on global distribution and ecology of its producers [J].Water Research,2014,66:320-337
[18] Amaral V,Bonilla S,Aubriot L.Growth optimization of the invasive cyanobacterium Cylindrospermopsis raciborskii in response to phosphate fluctuations [J].European Journal of Phycology,2014,49(1):134-141
[19] Dokulil M T.Vegetative survival of Cylindrospermopsis raciborskii (cyanobacteria) at low temperature and low light [J].Hydrobiologia,2016,764(1):241-247
[20] Briand J F,Leboulanger C,Humbert J F,et al.Cylindrospermopsis raciborskii (cyanobacteria) invasion at mid-latitudes:Selection,wide physiological tolerance,or global warming?[J].Journal of Phycology,2004,40(2):231-238
[21] Griffiths D J,Saker M L.The Palm Island mystery disease 20 years on:A review of research on the cyanotoxin cylindrospermopsin [J].Environmental Toxicology,2003,18(2):78-93
[22] Figueredo C C,Giani A.Does allelopathy contribute to Cylindrospermopsis raciborskii (cyanobacteria) bloom occurrence and geographic expansion?[J].Journal of Phycology,2007,43(2):256-265
[23] Bonilla S,Aubriot L,Soares M C S,et al.What drives the distribution of the bloom-forming cyanobacteria Planktothrix agardhii and Cylindrospermopsis raciborskii?[J].FEMS Microbiology Ecology,2012,79(3):594-607
[24] Sinha R,Pearson L A,Davis T W,et al.Increased incidence of Cylindrospermopsis raciborskii in temperate zones - Is climate change responsible?[J].Water Research,2012,46(5):1408-1419
[25] Lei L M,Peng L,Huang X H,et al.Occurrence and dominance of Cylindrospermopsis raciborskii and dissolved cylindrospermopsin in urban reservoirs used for drinking water supply,South China [J].Environmental Monitoring and Assessment,2014,186(5):3079-3090
[26] Xie J L,Yu G L,Xu X D,et al.The morphological and molecular detection for the presence of toxic Cylindrospermopsis (Nostocales,Cyanobacteria) in Beijing City,China [J].Journal of Oceanology and Limnology,2018,36(2):263-272
[27] Jiang Y G,Xiao P,Yu G L,et al.Sporadic distribution and distinctive variations of cylindrospermopsin genes in cyanobacterial strains and environmental samples from Chinese freshwater bodies [J].Applied and Environmental Microbiology,2014,80(17):5219-5230
[28] 路琰,雷敏婷,叶金梅,等.广东省千灯湖拟柱孢藻(Cylindrospermopsis raciborskii)的形态和产毒能力的株间差异及系统进化[J].湖泊科学,2020,32(1):144-153
Lu Y,Lei M T,Ye J M,et al.Intraspecific variation of morphological traits and toxin-producing capacity and phylogenetic analysis for Cylindrospermopsis raciborskii from Qiandenghu Lake,Guangdong Province [J].Journal of Lake Sciences,2020,32(1):144-153 (in Chinese)
[29] 晁爱敏,于海燕,肖鹏,等.杭州湘湖拉氏拟柱孢藻(Cylindrospermopsis raciborskii)藻株的分离及其特征研究[J].河南师范大学学报:自然科学版,2021,49(4):106-113
Chao A M,Yu H Y,Xiao P,et al.Isolation and characterization of a Cylindrospermopsis raciborskii strain from Lake Xianghu,Hangzhou [J].Journal of Henan Normal University:Natural Science Edition,2021,49(4):106-113 (in Chinese)
[30] Yang J,Yu X Q,Liu L M,et al.Algae community and trophic state of subtropical reservoirs in southeast Fujian,China [J].Environmental Science and Pollution Research,2012,19(5):1432-1442
[31] Yang J R,Lv H,Isabwe A,et al.Disturbance-induced phytoplankton regime shifts and recovery of cyanobacteria dominance in two subtropical reservoirs [J].Water Research,2017,120:52-63
[32] Orr P T,Willis A,Burford M A.Application of first order rate kinetics to explain changes in bloom toxicity—The importance of understanding cell toxin quotas [J].Journal of Oceanology and Limnology,2018,36(4):1063-1074
[33] Hongoh Y,Yuzawa H,Ohkuma M,et al.Evaluation of primers and PCR conditions for the analysis of 16S rRNA genes from a natural environment [J].FEMS Microbiology Letters,2003,221(2):299-304
[34] Edgar R C.MUSCLE:Multiple sequence alignment with high accuracy and high throughput [J].Nucleic Acids Research,2004,32(5):1792-1797
[35] Capella-Gutiérrez S,Silla-Martínez J M,Gabaldón T.trimAl:A tool for automated alignment trimming in large-scale phylogenetic analyses [J].Bioinformatics,2009,25(15):1972-1973
[36] Nguyen L T,Schmidt H A,von Haeseler A,et al.IQ-TREE:A fast and effective stochastic algorithm for estimating maximum-likelihood phylogenies [J].Molecular Biology and Evolution,2014,32(1):268-274
[37] Manya W F,Lizárraga W C,Mormontoy C G,et al.Complete genome sequence of Halomonas sp.strain SH5A2,a dye-degrading halotolerant bacterium isolated from the Salinas and Aguada Blanca national reserve in Peru [J].Microbiology Resource Announcements,2021,10(2):e01083-e01020
[38] Kumar S,Stecher G,Li M,et al.MEGA X:Molecular evolutionary genetics analysis across computing platforms [J].Molecular Biology and Evolution,2018,35(6):1547-1549
[39] Bolger A M,Lohse M,Usadel B.Trimmomatic:A flexible trimmer for Illumina sequence data [J].Bioinformatics,2014,30(15):2114-2120
[40] Luo R B,Liu B H,Xie Y L,et al.SOAPdenovo2:An empirically improved memory-efficient short-read de novo assembler [J].GigaScience,2012,1(1):2047-217X
[41] Kellmann R,Mills T,Neilan B A.Functional modeling and phylogenetic distribution of putative cylindrospermopsin biosynthesis enzymes [J].Journal of Molecular Evolution,2006,62(3):267-280
[42] Stucken K,John U,Cembella A,et al.The smallest known genomes of multicellular and toxic cyanobacteria:Comparison,minimal gene sets for linked traits and the evolutionary implications [J].PLoS One,2010,5(2):e9235
[43] Cohan F M,Koeppel A F.The origins of ecological diversity in prokaryotes [J].Current Biology,2008,18(21):R1024-R1034
[44] Padisák J.Cylindrospermopsis raciborskii (Woloszynska) Seenayya et Subba Raju,an expanding,highly adaptive cyanobacterium:Worldwide distribution and review of its ecology [J].Archiv Für Hydrobiologie Supplementband,1997,107:563-593
[45] Tawong W,Pongcharoen P,Nishimura T,et al.Molecular characterizations of Thai Raphidiopsis raciborskii (Nostocales,Cyanobacteria) based on 16S rDNA,rbcLX,and cylindrospermopsin synthetase genes [J].Plankton and Benthos Research,2019,14(4):211-223
[46] Shafik H M.Morphological characteristics of Cylindrospermopsis raciborskii (wo oszyńska) seenayya et subba raju in laboratory cultures [J].Acta Biologica Hungarica,2003,54(1):121-136
oszyńska) seenayya et subba raju in laboratory cultures [J].Acta Biologica Hungarica,2003,54(1):121-136
[47] Neilan B A,Saker M L,Fastner J,et al.Phylogeography of the invasive cyanobacterium Cylindrospermopsis raciborskii [J].Molecular Ecology,2003,12(1):133-140
[48] Atkinson K M.Birds as transporters of algae [J].British Phycological Journal,1972,7(3):319-321
[49] Mundkur T,Galbraith C,Jones T,et al.A review of migratory bird flyways and priorities for management [R].Bonn:UNEP/CMS Secretariat,2014
[50] Atkinson K M.Experiments in dispersal of phytoplankton by ducks [J].British Phycological Journal,1980,15(1):49-58
[51] Mazmouz R,Chapuis-Hugon F,Mann S,et al.Biosynthesis of cylindrospermopsin and 7-epicylindrospermopsin in Oscillatoria sp.strain PCC 6506:Identification of the cyr gene cluster and toxin analysis [J].Applied and Environmental Microbiology,2010,76(15):4943-4949
[52] Jiang Y G,Xiao P,Yu G L,et al.Molecular basis and phylogenetic implications of deoxycylindrospermopsin biosynthesis in the cyanobacterium Raphidiopsis curvata [J].Applied and Environmental Microbiology,2012,78(7):2256-2263