近年来,大量的塑料碎片通过直接或间接的方式进入水环境,成为最常见的海洋垃圾[1-2]。 塑料废弃物在自然环境中存留时间长,通过物理、化学和生物降解等方式裂解为更小的碎片[3-4],一般把粒径<5 mm 的塑料碎片定义为微塑料[5]。 微塑料广泛存在于海洋、河流和湖泊等各类环境介质和生物体中[6-7],因其尺寸小、比表面积大、疏水性高等表面特性,微生物更容易粘附定殖在其表面进而形成异质性结构组成的生物膜,被称之为“塑料圈(plastisphere)”[1,8]。 相比于其他天然基体和周围水环境,微塑料生物膜具有特异性的结构和生物群落组成[1,9-10],并且更易成为一些病原菌的聚集地[11-12],从而可能会影响抗生素抗性基因(antibiotic resistance genes, ARGs)在微塑料生物膜上的赋存和动态演化特征。 由于微塑料在水环境中大量存在且具有时间尺度的持久性、空间尺度高传输性[13],还可通过食物链在生态圈中传递[14],其携带ARGs 的扩散传播作用不容忽视,会对整个水生态圈安全甚至人类健康造成潜在威胁。
已有研究表明,河流、河口和海洋环境中的微塑料会对 ARGs 选择性富集,成为 ARGs 的库[15-17]。基体上生物膜的形成除了受基体特性的影响外,还取决于时间演替[18]。 因此,探究微塑料上ARGs 时间序列动态变化特征,对了解微塑料对ARGs 传播扩散的影响具有重要意义。 已有研究表明,聚乙烯(polyethylene, PE)微塑料生物膜上ARGs 和可移动元件具有时间动态演化特征,病原菌富集、可移动元件持久性以及对抗生素的耐受性是使得微塑料上ARGs 传播扩散风险增加的主要因素[19]。 抗生素的选择压力作用显著影响着ARGs 的传播[20-21],在抗生素胁迫下,PE 生物膜对ARGs 和Ⅰ类整合子整合酶基因(intI1)具有明显的选择富集作用[15],其中intI1 常被用作ARGs 在环境中水平转移的重要标记物[22]。 然而,抗生素胁迫浓度以及微塑料种类对其生物膜上ARGs 时间动态演化特征的影响目前尚不清楚。
本研究选择实际水环境中2 种常检出的微塑料PE 和聚苯乙烯(polystyrene, PS)[6-7],构建水-沉积物-微塑料微反应体系模拟实际水环境特征,研究不同浓度抗生素胁迫下,不同种类微塑料生物膜上ARGs、intI1 和微生物群落的时间序列动态变化特征及其关联性;探究不同微塑料生物膜上ARGs 差异性响应机制,进一步明确微塑料对ARGs 传播扩散的影响,对有效遏制微塑料引起的ARGs 传播风险具有重要科学意义。
1 材料与方法(Materials and methods)
1.1 实验设计
采集巢湖0 ~5 cm 表层沉积物和上覆水样,分别过 2 mm 筛和 20 ~25 μm 膜(Grade 4, Whatman,UK)去除大的颗粒物和浮游生物。 根据OECD 308指南,取80 g 沉积物和250 mL 水样在1 L 的锥形瓶中构建水-沉积物体系。 10 g 灭菌PE 颗粒(粒径为 3 ~4 mm,密度为 0.95 g·cm-3)、10 g 灭菌 PS 颗粒(粒径为3 ~4 mm,密度为1.05 g·cm-3)分别添加到水-沉积物体系(图1)。 水样中添加矿物营养(包含矿物盐、微量元素和生长因子)促进微生物增殖,矿物营养组分参照OECD 301 指南。 添加Na2CO3 维持体系中pH 值在6.8 ~7.2 之间,用透气隔菌膜封住瓶口。 所有反应体系置于22 ℃、恒温振荡培养箱90 r·min-1培养一周以达到稳定状态。 磺胺类抗生素磺胺甲恶唑(sulfamethoxazole, SMX)在我国地表水环境中具有较高的检出率和浓度水平[23],并常被用作抗生素胁迫作用研究中的代表性抗生素[24]。 选取EUCAST 数据库中SMX 的观察最低最小抑制浓度 1 000 μg·L-1作为高胁迫水平[25],并以 1/10 倍和1/100 倍浓度 SMX 作为中(100 μg·L-1)和低(10 μg·L-1)胁迫水平,对反应体系进行抗生素胁迫。 设置无抗生素胁迫的控制对照组(CK)。 每组均设3个平行实验组。 在第0 天(SMX 添加前),添加 SMX 后第15 天、第 30 天、第45 天和第 60 天取等量微塑料,并提取微塑料生物膜DNA,在-80 ℃保存用于进一步分析。 每次取微塑料前测定水相中SMX 浓度,向系统中补充一定量SMX 至初始设定浓度。

图1 磺胺甲恶唑(SMX)对聚乙烯(PE)和聚苯乙烯(PS)微塑料生物膜抗生素抗性基因(ARGs)的影响
Fig.1 The effect of sulfamethoxazole (SMX) on antibiotic resistance genes (ARGs) in polyethylene (PE)and polystyrene (PS) microplastic biofilms
1.2 生物膜分离和DNA 提取
在超净工作台中,用<3 mm 的灭菌漏网取2.5 g微塑料置于无菌烧杯中,用0.9%无菌NaCl 溶液轻轻冲洗微塑料颗粒,在50 mL 50 mmol·L-1 无菌焦磷酸缓冲液(Na4P2O7-Tween80, 0.05%)中涡旋2 min[26],分离并收集菌落生物膜,重复2 次,混匀后过0.22 μm 微孔滤膜。 所有滤膜置于冻存管,快速液氮冷冻后保存于-80 ℃冰箱待用。 用 FastDNA®SPIN 土壤基因组DNA 试剂盒提取微塑料生物膜中的总DNA,保存于-80 ℃冰箱。 超微量分光光度计(NanoDrop One, ThermoFisher)测定 DNA 的 OD260/OD280 值在 1.8 ~2.0 之间。
1.3 ARGs 分析
利用实时荧光定量 PCR (LightCycler® 96,Roche)对 3 种磺胺类抗性基因(sul1、sul2 和 sul3)、16S rRNA、intI1 基因进行定量分析。 以目标基因质粒的梯度稀释溶液作为模板DNA 进行qPCR 扩增,扩增体系共20 μL,包括2×SGExcel FastSYBR Mixture 10 μL,上、下引物(10 μmol·L-1)各 0.4 μL,模板DNA 1 μL,灭菌 ddH2O 补充至 20 μL。 引物和qPCR 扩增信息如表 1 所示。 按照 LightCycler® 96荧光定量PCR 仪默认程序进行熔点曲线分析,验证特异性扩增。 各目的基因的qPCR 标准曲线扩增效率为84% ~110%,R2 >0.99。 利用超微量分光光度计测得的质粒DNA 浓度,通过标准曲线即可得出目的基因的绝对丰度。 目标基因的相对丰度通过16S rRNA 进行归一化。
表1 目标基因的qPCR 引物序列和退火温度
Table 1 qPCR analysis primers and annealing temperature for target genes

目标基因Target genes引物Primer序列(5’ ~3’)Sequences (5’ ~3’)退火温度/℃Annealing temperature/℃片段大小/bp Amplicon size/bp参考文献Reference 16S rRNA 341F CCTACGGGAGGCAGCAG 534R TTACCGCGGCTGCTGGCAC 60 193 [27]intI1 intI1-F CCTCCCGCACGATGATC intI1-R TCCACGCATCGTCAGGC 60 280 [28]sul1 sul1-F CACCGGAAACATCGCTGCA sul1-R AAGTTCCGCCGCAAGGCT 57.5 158 [29]sul2 sul2-F TCCGGTGGAGGCCGGTATCTGG sul2-R CGGGAATGCCATCTGCCTTGAG 55 191 [30]sul3 sul3 -F CCCATACCCGGATCAAGAATAA sul3-R CAGCGAATTGGTGCAGCTACTA 57 143 [29]
1.4 16S rRNA 扩增子测序和数据处理
基于上海美吉生物医药科技有限公司的Illumina MiSeq 平台(Illumina, San Diego, USA)对 CK 和SMX 胁迫第60 天PE 和PS 生物膜样品的细菌群落16S rRNA 进行测序。 首先利用热循环 PCR 系统(GeneAmp 9700, ABI, USA),采用引物 338F(5’-ACTCCTACGGGAGGCAGCAG-3’) 和 806R (5’-GGACTACHVGGGTWTCTAAT-3’) 对 细 菌 16S rRNA 基因的V3-V4 高变异区进行Miseq 扩增子测序。 对MiSeq 测序得到的是双端序列数据,进行拼接、质控过滤、去接头等得到优化数据。 基于优化数据按照97%相似性对非重复序列进OTU 聚类(版本7.1,http://drive5.com/uparse/)。 比对 Silva 数据库,采用RDP 分类器贝叶斯算法(http://rdp.cme.msu.edu/)对97%相似水平的OTU 代表序列进行分类学分析。
1.5 统计学分析
利用SPSS 22.0 统计软件(IBM, USA)对目标基因进行显著性检验及相关性分析。 针对符合正态分布的数据,显著性检验采用独立样本t 检验和单因素方差分析;非正态分布的数据采用Wilcox 秩和检验组间非参数检验法。 基于Bray-Curtis 距离算法采用主坐标分析(PCoA)法分析不同微塑料在OTU水平上的细菌群落结构差异性,并利用相似性分析(ANOSIM)[31]对差异性进行检验(999 次置换)。 利用R vegan 包进行 PCoA 和 ANOSIM 分析。 利用 R Hmisc 包对ARGs、intI1 及抗性基因细菌宿主进行Spearman 秩相关性分析,并通过Gephi 平台(V0.9.1)对相关分析结果进行网络可视化分析。
2 结果与讨论(Results and discussion)
2.1 抗生素胁迫下不同微塑料生物膜上ARGs 和intI1 分布特征
PS 与PE 生物膜中所有样品中除sul3 外,sul1、sul2 和intI1 基因均被检出。 在 CK 组和 SMX 选择压力下,PS 与 PE 生物膜中 sul1、sul2 和 intI1 基因相对丰度均无显著差异(t 检验,P>0.05)(图2(a)和图2(b))。 此外,有研究也表明长江口5 种不同微塑料上ARGs 丰度总体上无显著性差异[17]。 可见,不同的微塑料上ARGs 丰度可能不具有显著差异。 由于目标微塑料种类和基因有限,需采用高通量qPCR 等技术手段进一步分析更多的微塑料上ARGs 丰度水平,以确定微塑料生物膜上ARGs 是否存在差异性分布特征。
在SMX 的选择压力作用下,PE 和PS 生物膜上sul1、sul2 和intI1 基因总丰度显著高于无抗生素选择的 CK 组(t 检验,PE 生物膜:P<0.05;PS 生物膜:P<0.01)(图 2(a)和图 2(b))。 可见,抗生素的胁迫作用会进一步增加微塑料上ARGs 丰度水平。 此外,微塑料生物膜中sul1 与intI1 之间存在显著相关性(PE 生物膜:r=0.78, P<0.05;PS 生物膜:r=0.88, P<0.05),研究证实sul1 基因位于intI1 的保守片段上[32],因此在微塑料生物膜上intI1 丰度增加是促进ARGs 扩散的关键因素之一。
不同浓度的抗生素胁迫会对ARGs 扩散产生差异性影响[33]。 对不同浓度 SMX 胁迫下,微塑料生物膜上磺胺类抗性基因(sul)和intI1 基因相对丰度进行比较(图 2(c)和图 2(d))。 在低浓度(10 μg·L-1)和中等浓度(100 μg·L-1)SMX 胁迫下,sul 和 intI1基因丰度未产生显著性差异(单因素方差分析,P>0.05),高浓度(1 000 μg·L-1)SMX 胁迫使得基因相对丰度显著增加(单因素方差分析,P<0.05),其原因可能是由于与低浓度胁迫相比,1 000 μg·L-1 SMX胁迫下ARGs 接合转移频率更高[34],且亚抑制浓度抗生素胁迫也会增加抗性突变概率[35]。 此外,有研究表明受高浓度SMX 胁迫的活性污泥反应器中ARGs 丰度也会明显增加[36]。 PE 和 PS 生物膜上sul 和intI1 基因总体的相对丰度在时间序列上均无显著变化(单因素方差分析,P>0.05)。 然而,高浓度SMX(1 000 μg·L-1)胁迫下明显增加了 sul 和 intI1基因相对丰度,且随着时间变化具有显著性差异,在胁迫15 d 后增速开始明显下降;中浓度SMX 胁迫下sul 基因随时间增加无显著变化;低浓度胁迫下sul 和intI1 基因相对丰度在第60 天显著增加。 高浓度抗生素胁迫下微塑料上ARGs 丰度增速明显降低的主要原因可能是由于微塑料生物膜逐渐形成并成熟,会对抗生素产生耐受[37],从而降低对ARGs 的选择压力作用。 此外,长时间高浓度SMX 胁迫会改变微生物群落结构,同时也可能抑制微塑料上ARGs 潜在宿主菌的生长[38]。 然而,低浓度 SMX 会逐渐在微塑料生物膜表面积累产生选择压力作用,随着时间演化进一步促进ARGs 传播。
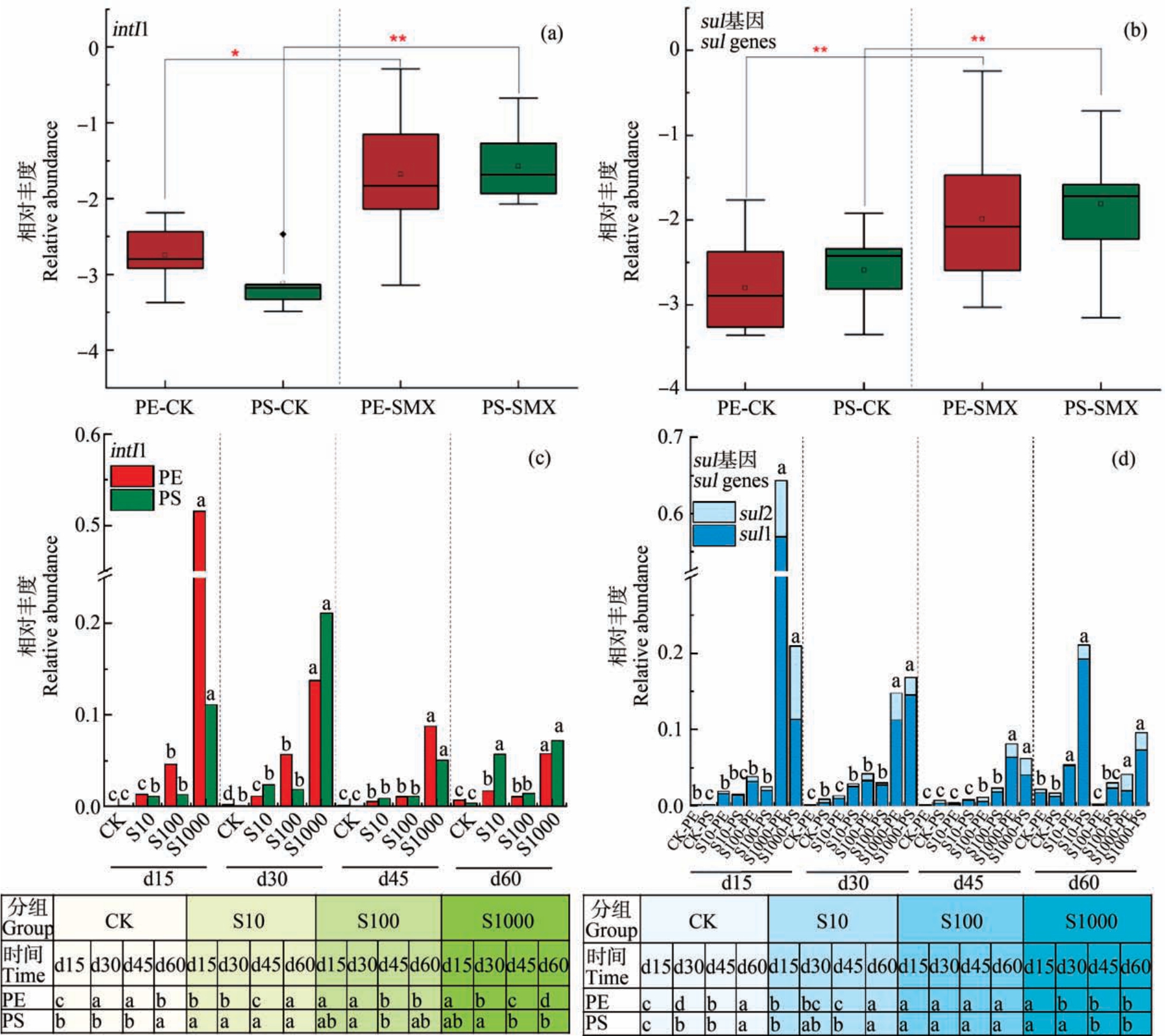
图2 intI1 和ARGs 在PE 和PS 生物膜上相对丰度(log10 转换值)的比较(a)~(b)与不同浓度抗生素胁迫下PE 与PS 生物膜上ARGs 和intI1 的时间序列变化特征和比较(c)~(d)
注:CK、S10、S100、S1000 分别表示 0、10、100 和 1 000 μg·L-1的 SMX 浓度;d15、d30、d45 和 d60 表示采样时间在第 15 天、第 30 天、第45 天和第60 天;图中不同字母表示不同选择压力下intI1 和ARGs 具有显著性差异;*P<0.05,**P<0.01。
Fig.2 Comparisons of the relative abundance (log10 conversion value) of intI1 and ARGs in PE and PS biofilms (a)~(b), and the time series changes and comparisons of ARGs and intI1 in PE and PS biofilms under antibiotic stress at different concentrations (c)~(d)
Note: CK, S10, S100 and S1000 indicate the concentrations of SMX at 0,10,100, and 1 000 μg·L-1, respectively; d15, d30, d45, and d60 indicate the sampling time at day 15, day 30, day 45, and day 60, respectively; different letters indicate significant differences between intI1 and ARGs under different selection pressures and among time series changes; *P<0.05 and **P<0.01.
2.2 不同微塑料生物膜细菌群落的差异性
PE 和PS 微塑料生物膜上的OTU 总数分别为4 384个和5 369个,可见PS 微塑料细菌群落的物种较丰富。 对 PE、PS 生物膜细菌群落进行 Alpha多样性分析(表2),Coverage 值>98%,说明测序结果完整、可信度高。 PS 生物膜的 Sobs、ACE 和 Chao指数都显著高于PE 生物膜(P<0.01),表明PS 生物膜上细菌群落丰富度较高。 PS 的Shannon 指数较高且Simpson 指数较小,表明PS 生物膜具有较高的均匀度,但2 种微塑料生物膜细菌群落均匀度无显著差异(P>0.05)。 整体而言,PE 和 PS 生物膜细菌群落的多样性存在一定的差异性。
表2 PE 和PS 微塑料生物膜上Alpha 多样性比较
Table 2 Comparisons of Alpha diversity in PE and PS microplastic biofilms
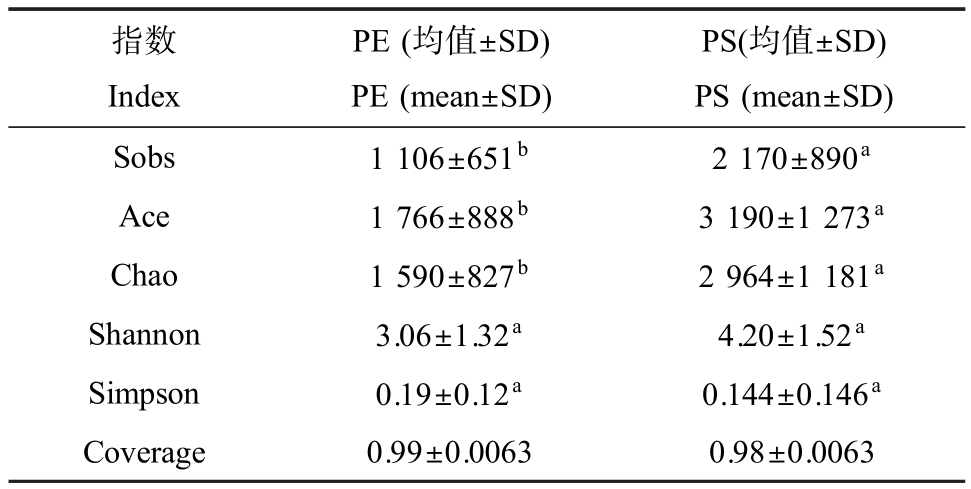
注:组间差异性检验采用Wilcox 秩和检验;不同字母表示具有显著性差异,相同字母表示无显著性差异;SD 为标准偏差。
Note: Wilcox rank sum test was used to test the differences among groups; different letters indicate significant differences, and the same letters indicate no significant differences; SD is the standard deviation.
指数Index PE (均值±SD)PE (mean±SD)PS(均值±SD)PS (mean±SD)Sobs 1 106±651b 2 170±890a Ace 1 766±888b 3 190±1 273a Chao 1 590±827b 2 964±1 181a Shannon 3.06±1.32a 4.20±1.52a Simpson 0.19±0.12a 0.144±0.146a Coverage 0.99±0.0063 0.98±0.0063
变形菌(Proteobacteria)、拟杆菌(Bacteroidetes)和放线菌(Actinobacteria)在2 种微塑料生物膜上占主导,与长江口实际水环境中微塑料上细菌门种类较一致[9]。 PCoA 分析和 ANOSIM 显著性检验显示PE 和PS 生物膜细菌群落结构存在显著差异(R=0.423, P<0.01)(图 3(a))。 不同浓度 SMX 胁迫对微塑料细菌群落结构产生显著性影响(PS: R=1.00, P<0.01; PE: R=1.00, P<0.01),且 PC1 和 PC2 共解释度均超过90%(图3(b)和图3(c))。 可见,不同种类微塑料生物膜细菌群落结构存在显著差异,不同浓度SMX 胁迫会明显改变微塑料生物膜上细菌群落结构。 其中,酸杆菌门(Acidobacteria)、绿弯菌门(Chloroflexi)、硝化螺旋菌门(Nitrospirae)和疣微菌门(Verrucomicrobia)等优势菌在PE 和PS 生物膜上具有显著差异,且在PS 生物膜中相对丰度均显著高于PE生物膜(图 4(a)和图 4(b))。
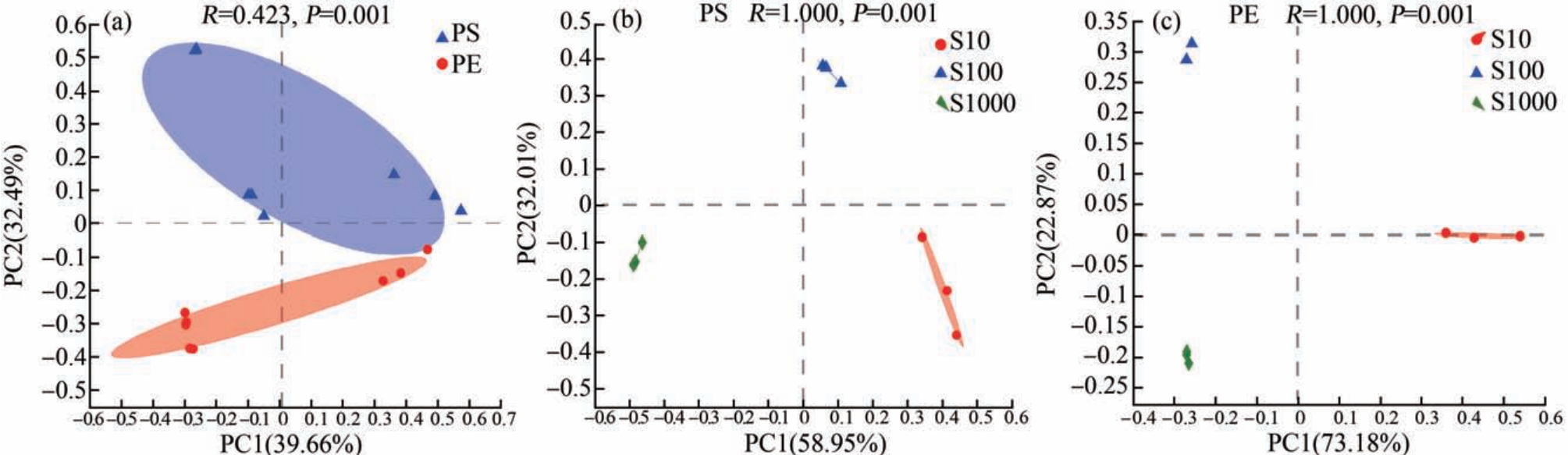
图3 PS 与PE 生物膜细菌群落比较分析(OTU 水平)(a)与不同浓度SMX 对PS 和PE 生物膜细菌群落的差异性影响(OTU 水平)(b~c)
Fig.3 Comparative analysis of bacterial communities in PS and PE biofilm (at OTU level) (a), and influence of SMX at difference concentrations on the bacterial communities in PS and PE biofilms (at OTU level) (b~c)
目前微塑料种类对附着微生物群落结构的影响研究尚未获得一致的结论。 Zettler 等[1]对北大西洋不同微塑料样品的分析表明,PE 和聚丙烯(polypropylene, PP)生物膜微生物群落存在差异性;然而,de Tender 等[39]对北海PE 和PP 生物膜样品中微生物群落未观察到明显的差异性;此外,Li 等[40]对海河口PE、PP 和PS 这3 种微塑料生物膜细菌群落结构也未观察到明显的差异性。 然而本研究中PE 和PS微塑料生物膜细菌群落结构存在差异性。 目前有关微塑料材质引起的微生物群落结构差异性的机制尚不清楚,一种原因可能是由于不同微塑料生物膜结构组成存在差异[41],同时不同的生物膜结构组成对抗生素的胁迫响应也存在差别,从而会导致对微塑料生物膜中微生物群落产生不同的选择压力作用。
2.3 细菌群落与ARGs 和intI1 的共现性网络分析
细菌群落的演变会引起 ARGs 丰度水平变化[42]。 对不同种类微塑料生物膜细菌群落与ARGs进行网络分析,确定不同分类水平下细菌群落与ARGs 间的关联性,找出2 种微塑料上的潜在ARGs宿主菌。 PE 生物膜上细菌群落5个细菌门与sul1、sul2 和intI1 具有明显关联性(图5(a));其中,变形菌门和放线菌门为PE 生物膜上优势细菌,分别占总关联菌的52.17%和21.74%。 PS 生物膜上细菌群落6个细菌门(24个潜在宿主菌属)与 sul1、sul2 和intI1 具有明显相关关系(图5(b));其中,厚壁菌门(Firmicutes)和变形菌门为PS 生物膜上的优势细菌,均占总体的33.33%。 已有研究表明细菌间ARGs 水平转移主要发生在变形菌门、放线菌门和厚壁菌门基因组中[43-45]。 然而,PE 和 PS 上这 3 种与 ARGs 扩散密切相关的细菌在门水平无显著性差异(图4(b))。

图4 PE 和PS 微塑料生物膜细菌群落门水平结构和差异性比较
注:Wilcoxon 秩和检验,*P<0.05。
Fig.4 The structure characteristics of bacterial communities in PE and PS biofilms at phylum level and their comparisons
Note: Wilcoxon rank sum test, *P<0.05.
在属水平,PE 和 PS 生物膜上分别有23个和24个细菌与ARGs 和intI1 具有明显共线性,且2 种微塑料上ARGs 潜在宿主菌并不相同(图5(a))。 在PE 生物膜上的潜在宿主菌中,贪噬菌属(Variovorax)和Curvibacter 已在瓶装矿泉水中被发现具有多重耐药性[46];Nordella 携带sul 基因对磺胺类抗生素具有抗性[47];奈瑟菌属(Neisseria)和红球菌属(Rhodococcus)具有多重耐药性[48-49]。 在 PS 生物膜潜在宿主菌中,假单胞菌属(Pseudomonas)作为一种常见的ARGs 宿主菌,在SMX 胁迫下假单胞菌属多重耐药性明显增强[50];在剩余污泥中副球菌属(Paracoccus)被发现具有多重耐药性[51];在SMX 对序批式反应器(SBR)系统胁迫的活性污泥和水样中动胶菌属(Zoogloea)为产生似sul1 的主要细菌[52];在鸡肠道微生物共生体中Porphyromonadaceae 具有四环素抗性[53],PS 生物膜上该菌与sul1 共现,说明其具有多重耐药性。 由此可见,在属水平,PE 和PS 生物膜上的一些细菌已被证实是ARGs 潜在宿主菌,且2 种微塑料生物膜上潜在宿主菌具有显著差异。 此外,PS 和PE 生物膜上sul1 与intI1 均具有明显的关联性,并在潜在宿主菌中具有共现性,可见intI1 在微塑料ARGs 传播扩散中具有重要作用。 intI1 在2 种微塑料上无显著差异也是PE 和PS 上ARGs 相对丰度无显著差异的主要因素之一。
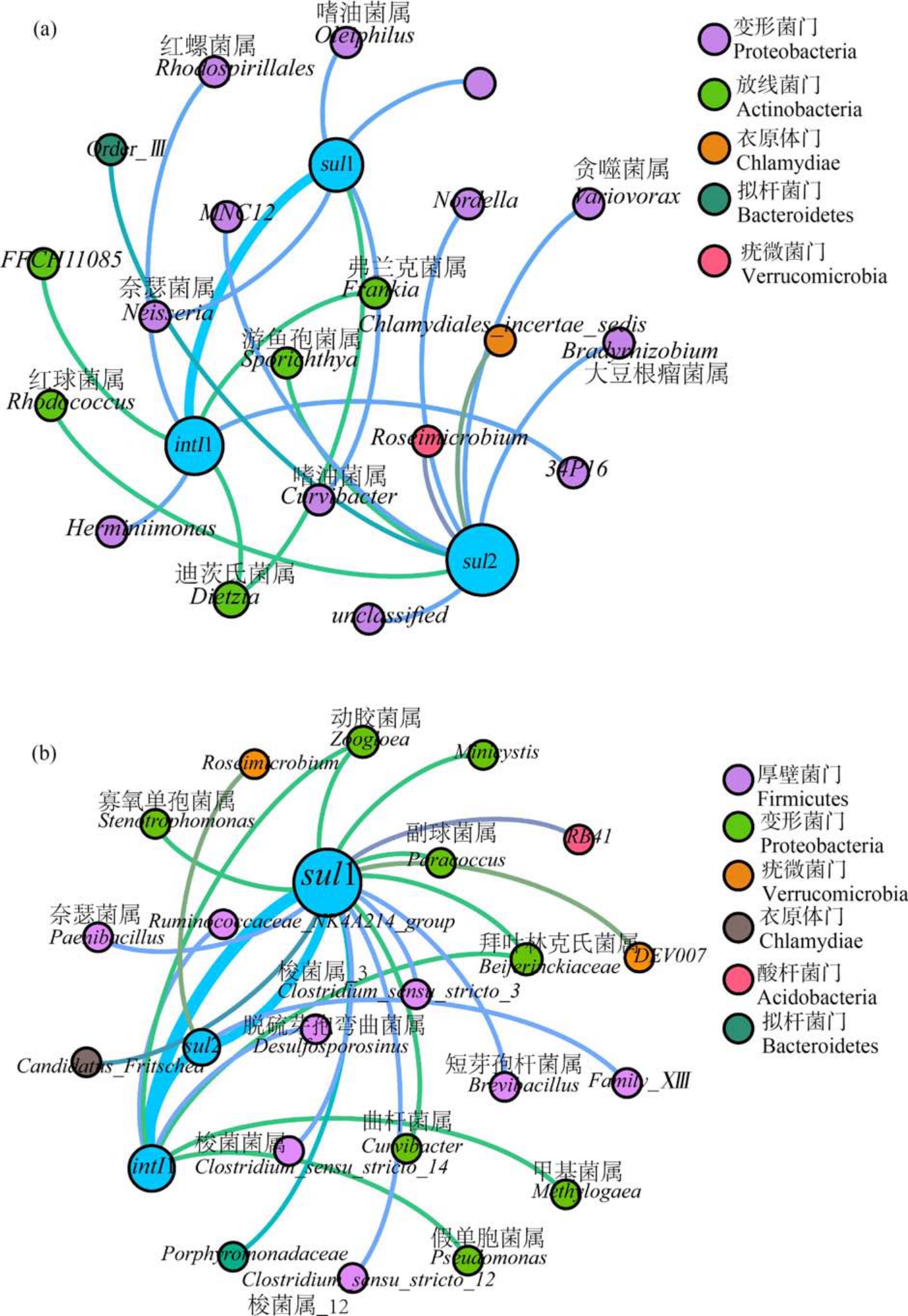
图5 PE(a)和PS(b)生物膜中ARGs、intI1 和细菌群落(属水平)间的共现模式
注:节点按微生物群落门分类进行着色,每个节点的大小与连接的数量(度)成正比,连线表示强的相关性(r≥0.7,P<0.05)。
Fig.5 The co-occurrence pattern among ARGs, intI1 and bacterial communities (genus level) in PE (a) and PS (b) biofilms
Note: The nodes are colored according to the microbial community classification at phylum level, and the size of each node is proportional to the number of connections (degrees); the line indicates a strong correlation (r≥0.7, P<0.05).
本研究未发现 SMX 胁迫对不同微塑料上ARGs 的相对丰度产生显著影响。 然而,SMX 的胁迫作用可以通过增加intI1 丰度显著提高微塑料上ARGs 的丰度水平。 其中,高浓度的 SMX 胁迫对ARGs 丰度具有更明显的促进作用,但随着时间演化促进作用明显减弱。 尽管2 种微塑料生物膜上细菌群落组成结构具有明显差异,而与ARGs 强关联性的细菌门的群落组成并无显著性差异;然而,在属水平上2 种微塑料ARGs 潜在宿主菌明显不同,但未明显影响其生物膜上ARGs 丰度水平。 微塑料生物膜上intI1 与ARGs 的相关性,及其在细菌群落网络中与ARGs 的共现性,均表明intI1 也是促进微塑料上ARGs 扩散的主要因素,但2 种微塑料上intI1无明显差异,未引起微塑料上ARGs 差异性分布。由于目的ARGs 和微塑料种类有限,研究结果可能存在局限性,因此为探明微塑料种类对ARGs 传播的影响,需对更多种类的微塑料上多种ARGs 和可移动元件的富集特征进行研究,进一步明确不同微塑料上ARGs 分布特征和演化规律,及引起微塑料上ARGs 传播的关键因子。
[1]Zettler E R, Mincer T J, Amaral-Zettler L A. Life in the“plastisphere”:Microbial communities on plastic marine debris [J].Environmental Science& Technology,2013,47(13):7137-7146
[2]刘彬, 侯立安, 王媛, 等. 我国海洋塑料垃圾和微塑料排放现状及对策[J]. 环境科学研究, 2020, 33(1): 174-182
Liu B, Hou L A, Wang Y, et al. Emission estimate and countermeasures of marine plastic debris and microplastics in China [J]. Research of Environmental Sciences,2020,33(1):174-182 (in Chinese)
[3]Cooper D A, Corcoran P L. Effects of mechanical and chemical processes on the degradation of plastic beach debris on the island of Kauai, Hawaii [J]. Marine Pollution Bulletin,2010,60(5):650-654
[4]O’Brine T,Thompson R C.Degradation of plastic carrier bags in the marine environment [J].Marine Pollution Bulletin,2010,60(12):2279-2283
[5]Thompson R C, Olsen Y, Mitchell R P, et al. Lost at sea:Where is all the plastic? [J]. Science, 2004, 304(5672):838
[6]张子琪, 高淑红, 康园园, 等. 中国水环境微塑料污染现状及其潜在生态风险[J]. 环境科学学报, 2020, 40(10):3574-3581
Zhang Z Q, Gao S H, Kang Y Y, et al. Current status of microplastics contamination in China’ s water environment and its potential ecological risks [J]. Acta Scientiae Circumstantiae,2020,40(10):3574-3581 (in Chinese)
[7]李富云, 贾芳丽, 涂海峰, 等. 海洋中微塑料的环境行为和生态影响[J]. 生态毒理学报,2017,12(6):11-18
Li F Y,Jia F L,Tu H F,et al.Environmental behavior and ecological effects of microplastics in the ocean [J]. Asian Journal of Ecotoxicology,2017,12(6):11-18 (in Chinese)
[8]季梦如, 马旖旎, 季荣. 微塑料圈: 环境微塑料对微生物的载体作用[J]. 环境保护,2020,48(23):19-27
Ji M R, Ma Y N, Ji R. Plastisphere:The vector effects of microplastics on microbial communities [J]. Environmental Protection,2020,48(23):19-27 (in Chinese)
[9]Jiang P L, Zhao S Y, Zhu L X, et al.Microplastic-associated bacterial assemblages in the intertidal zone of the Yangtze Estuary [J]. The Science of the Total Environment,2018,624:48-54
[10]Vosshage A T L, Neu T R, Gabel F. Plastic alters biofilm quality as food resource of the freshwater gastropod Radix balthica [J]. Environmental Science & Technology,2018,52(19):11387-11393
[11]Wu N, Zhang Y, Zhao Z, et al. Colonization characteristics of bacterial communities on microplastics compared with ambient environments (water and sediment) in Haihe Estuary [J]. The Science of the Total Environment, 2020,708:134876
[12]Wu X J,Pan J,Li M,et al.Selective enrichment of bacterial pathogens by microplastic biofilm [J]. Water Research,2019,165:114979
[13]张晨, 王清, 赵建民. 海洋微塑料输运的数值模拟研究进展[J]. 地球科学进展,2019,34(1):72-83
Zhang C, Wang Q, Zhao J M. Numerical simulation of transportation of marine microplastics: A review [J]. Advances in Earth Science,2019,34(1):72-83 (in Chinese)
[14]Carbery M, O’Connor W, Palanisami T. Trophic transfer of microplastics and mixed contaminants in the marine food web and implications for human health [J].Environment International,2018,115:400-409
[15]Wang S S,Xue N N,Li W F,et al.Selectively enrichment of antibiotics and ARGs by microplastics in river, estuary and marine waters [J]. The Science of the Total Environment,2020,708:134594
[16]Yang Y Y, Liu G H, Song W J, et al. Plastics in the marine environment are reservoirs for antibiotic and metal resistance genes [J].Environment International,2019,123:79-86
[17]Guo X P, Sun X L, Chen Y R, et al.Antibiotic resistance genes in biofilms on plastic wastes in an estuarine environment [J].Science of the Total Environment,2020,745:140916
[18]Rijavec T,Zrimec J,van Spanning R,et al.Natural microbial communities can be manipulated by artificially constructed biofilms [J]. Advanced Science, 2019, 6(22):1901408
[19]Yang K, Chen Q L, Chen M L, et al. Temporal dynamics of antibiotic resistome in the plastisphere during microbial colonization [J]. Environmental Science & Technology,2020,54(18):11322-11332
[20]Deng C X,Liu X W,Li L L,et al.Temporal dynamics of antibiotic resistant genes and their association with the bacterial community in a water-sediment mesocosm under selection by 14 antibiotics [J]. Environment International,2020,137:105554
[21]He X L, Xu Y B, Chen J L, et al. Evolution of corresponding resistance genes in the water of fish tanks with multiple stresses of antibiotics and heavy metals [J].Water Research,2017,124:39-48
[22]Dickinson A W,Power A,Hansen M G,et al.Heavy metal pollution and co-selection for antibiotic resistance: A microbial palaeontology approach [J]. Environment International,2019,132:105117
[23]Li S,Shi W Z,Liu W,et al.A duodecennial national synthesis of antibiotics in China’ s major rivers and seas(2005—2016) [J]. The Science of the Total Environment,2018,615:906-917
[24]Aydin S,Ince B,Ince O.Development of antibiotic resistance genes in microbial communities during long-term operation of anaerobic reactors in the treatment of pharmaceutical wastewater [J].Water Research,2015,83:337-344
[25]Bengtsson-Palme J, Larsson D G J.Concentrations of antibiotics predicted to select for resistant bacteria: Proposed limits for environmental regulation [J].Environment International,2016,86:140-149
[26]Arias-Andres M, Klümper U,Rojas-Jimenez K,et al.Microplastic pollution increases gene exchange in aquatic ecosystems [J]. Environmental Pollution, 2018, 237: 253-261
[27]Koike S, Krapac I G, Oliver H D, et al. Monitoring and source tracking of tetracycline resistance genes in lagoons and groundwater adjacent to swine production facilities over a 3-year period [J]. Applied and Environmental Microbiology,2007,73(15):4813-4823
[28]Schmitt H, Stoob K, Hamscher G, et al.Tetracyclines and tetracycline resistance in agricultural soils:Microcosm and field studies [J]. Microbial Ecology,2006,51(3):267-276
[29]Luo Y, Mao D Q, Rysz M, et al. Trends in antibiotic resistance genes occurrence in the Haihe River, China [J].Environmental Science & Technology, 2010, 44 (19):7220-7225
[30]Pei R T, Kim S C, Carlson K H, et al. Effect of river landscape on the sediment concentrations of antibiotics and corresponding antibiotic resistance genes (ARG) [J].Water Research,2006,40(12):2427-2435
[31]Clarke K R, Warwick R M.Change in Marine Communities: An Approach to Statistical Analysis and Interpretation [M]. Boston: Plymouth Marine Laboratory, Natural Environment Research Council,1994:23-30
[32]Zhang X X, Zhang T, Zhang M, et al. Characterization and quantification of class 1 integrons and associated gene cassettes in sewage treatment plants [J]. Applied Microbiology and Biotechnology,2009,82(6):1169-1177
[33]卢文强, 孙昊宇, 王雅娟, 等. 抗生素的胁迫与抗生素抗性基因产生与传播关系的研究[J]. 生态毒理学报,2020,15(4):129-138
Lu W Q, Sun H Y, Wang Y J, et al. The relationship of antibiotic stress with emergence and dissemination of antibiotic resistance genes [J].Asian Journal of Ecotoxicology,2020,15(4):129-138 (in Chinese)
[34]Jutkina J, Marathe N P, Flach C F, et al. Antibiotics and common antibacterial biocides stimulate horizontal transfer of resistance at low concentrations [J]. The Science of the Total Environment,2018,616-617:172-178
[35]Andersson D I,Hughes D.Microbiological effects of sublethal levels of antibiotics [J]. Nature Reviews Microbiology,2014,12(7):465-478
[36]Zhao R X, Feng J, Liu J, et al. Deciphering of microbial community and antibiotic resistance genes in activated sludge reactors under high selective pressure of different antibiotics [J]. Water Research,2019,151:388-402
[37]Wang L F, Li Y, Wang L, et al. Extracellular polymeric substances affect the responses of multi-species biofilms in the presence of sulfamethizole [J]. Environmental Pollution,2018,235:283-292
[38]Zhang Y Y, Geng J J, Ma H J, et al. Characterization of microbial community and antibiotic resistance genes in activated sludge under tetracycline and sulfamethoxazole selection pressure [J]. The Science of the Total Environment,2016,571:479-486
[39]de Tender C A,Devriese L I,Haegeman A,et al.Bacterial community profiling of plastic litter in the Belgian part of the North Sea [J]. Environmental Science & Technology,2015,49(16):9629-9638
[40]Li W J,Zhang Y,Wu N,et al.Colonization characteristics of bacterial communities on plastic debris influenced by environmental factors and polymer types in the Haihe Estuary of Bohai Bay, China [J]. Environmental Science &Technology,2019,53(18):10763-10773
[41]Vosshage A T L, Neu T R, Gabel F. Plastic alters biofilm quality as food resource of the freshwater gastropod Radix balthica [J]. Environmental Science & Technology,2018,52(19):11387-11393
[42]Udikovic-Kolic N, Wichmann F, Broderick N A, et al.Bloom of resident antibiotic-resistant bacteria in soil following manure fertilization [J].Proceedings of the National Academy of Sciences of the United States of America,2014,111(42):15202-15207
[43]Cheng D M, Liu Y W, Shehata E, et al.In-feed antibiotic use changed the behaviors of oxytetracycline, sulfamerazine, and ciprofloxacin and related antibiotic resistance genes during swine manure composting [J]. Journal of Hazardous Materials,2021,402:123710
[44]Han X M,Hu H W,Chen Q L,et al.Antibiotic resistance genes and associated bacterial communities in agricultural soils amended with different sources of animal manures[J]. Soil Biology and Biochemistry,2018,126:91-102
[45]Lu J,Zhang Y X,Wu J,et al.Occurrence and spatial distribution of antibiotic resistance genes in the Bohai Sea and Yellow Sea areas,China [J].Environmental Pollution,2019,252(Pt A):450-460
[46]Falcone-Dias M F, Vaz-Moreira I, Manaia C M. Bottled mineral water as a potential source of antibiotic resistant bacteria [J]. Water Research,2012,46(11):3612-3622
[47]Chen J F, Tong T L, Jiang X S, et al. Biodegradation of sulfonamides in both oxic and anoxic zones of vertical flow constructed wetland and the potential degraders [J].Environmental Pollution,2020,265(Pt B):115040
[48]Mayers D L, Sobel J D, Ouellette M, et al. Antimicrobial Drug Resistance: Clinical and Epidemiological Aspects,Volume 2 [M]. New York: Springer,2009:763-782
[49]Álvarez-Narváez S, Giguère S, Cohen N, et al. Spread of multidrug-resistant Rhodococcus equi, United States [J].Emerging Infectious Diseases,2021,27(2):529-537
[50]Yan W F, Bai R, Wang S Q, et al. Antibiotic resistance genes are increased by combined exposure to sulfamethoxazole and naproxen but relieved by low-salinity [J].Environment International,2020,139:105742
[51]Shi Z J, Zhao R X, Wan J J, et al. Metagenomic analysis reveals the fate of antibiotic resistance genes in two-stage and one-stage anaerobic digestion of waste activated sludge [J]. Journal of Hazardous Materials, 2021, 406:124595
[52]张滢楹. 典型抗生素对SBR 系统微生物活性与种群结构的影响研究[D]. 南京: 南京大学,2016:54-59
Zhang Y Y. Study on effects of typical antibiotics on the microbial activity and community structure in SBR system [D]. Nanjing: Nanjing University, 2016: 54-59 (in Chinese)
[53]Juricova H, Matiasovicova J, Kubasova T, et al. The distribution of antibiotic resistance genes in chicken gut microbiota commensals [J]. Scientific Reports, 2021, 11(1):3290